- Главная
- Разное
- Дизайн
- Бизнес и предпринимательство
- Аналитика
- Образование
- Развлечения
- Красота и здоровье
- Финансы
- Государство
- Путешествия
- Спорт
- Недвижимость
- Армия
- Графика
- Культурология
- Еда и кулинария
- Лингвистика
- Английский язык
- Астрономия
- Алгебра
- Биология
- География
- Детские презентации
- Информатика
- История
- Литература
- Маркетинг
- Математика
- Медицина
- Менеджмент
- Музыка
- МХК
- Немецкий язык
- ОБЖ
- Обществознание
- Окружающий мир
- Педагогика
- Русский язык
- Технология
- Физика
- Философия
- Химия
- Шаблоны, картинки для презентаций
- Экология
- Экономика
- Юриспруденция
Хромосомный микроматричный анализ презентация
Содержание
- 1. Хромосомный микроматричный анализ
- 2. СИНДРОМ - ? ВПС Особенности фенотипа Аномалии
- 3. СИНДРОМ - ? Неонатальная гипотония Нормальный или
- 4. 1 970,0 млн USD 469,2 млн USD
- 5. 1 970,0 млн USD 469,2 млн USD
- 6. ХРОМОСОМНАЯ ПАТОЛОГИЯ ОПРЕДЕЛЕНИЕ Хромосомная патология – любое нарушение числа и/или структуры хромосом
- 7. ХРОМОСОМНАЯ ПАТОЛОГИЯ КЛИНИЧЕСКОЕ ЗНАЧЕНИЕ числовые и структурные
- 8. ЭВОЛЮЦИЯ ЦИТОГЕНЕТИЧЕСКИХ МЕТОДОВ Термин «кариотип» впервые
- 9. НА ПУТИ К МОЛЕКУЛЯРНОМУ КАРИОТИПИРОВАНИЮ
- 10. НА ПУТИ К МОЛЕКУЛЯРНОМУ КАРИОТИПИРОВАНИЮ
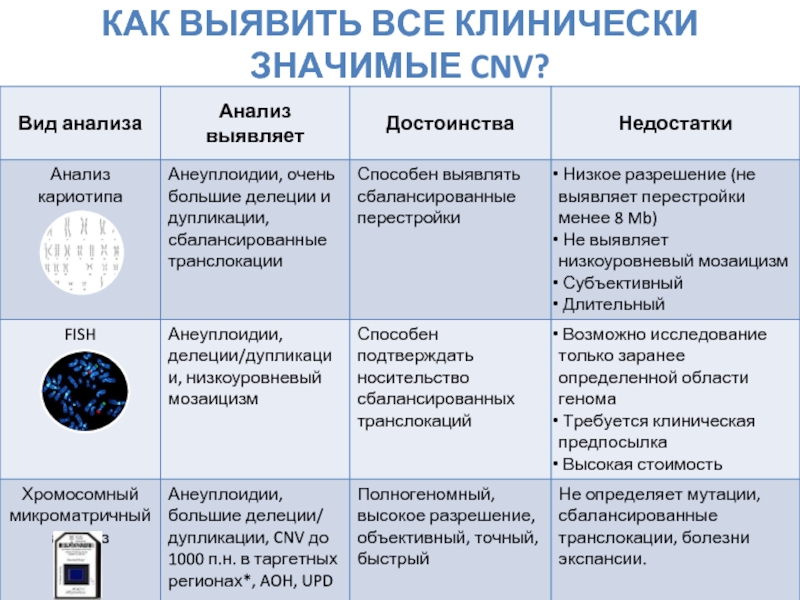
- 11. КАК ВЫЯВИТЬ ВСЕ КЛИНИЧЕСКИ ЗНАЧИМЫЕ CNV?
- 12. Хромосомный микроматричный анализ – молекулярно-цитогенетический метод анализа
- 13. ИСТОРИЯ Первый прототип микроматицы
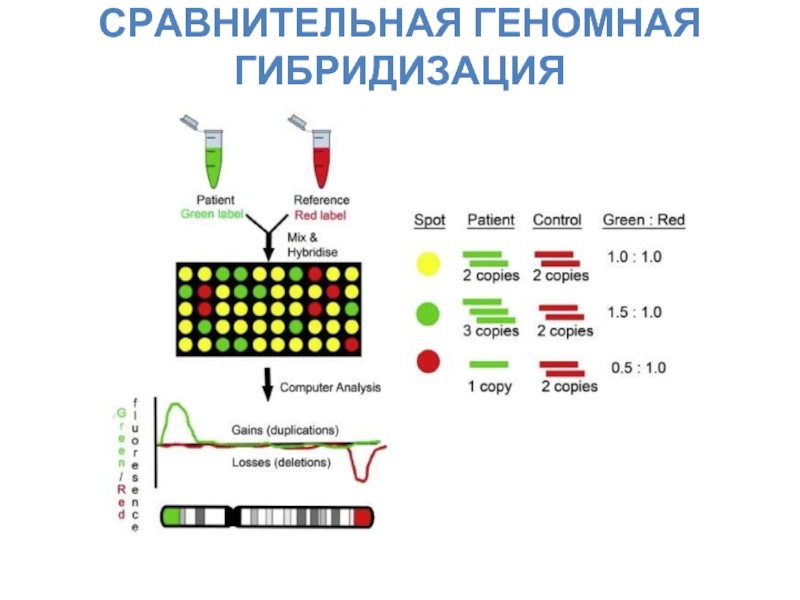
- 14. СРАВНИТЕЛЬНАЯ ГЕНОМНАЯ ГИБРИДИЗАЦИЯ
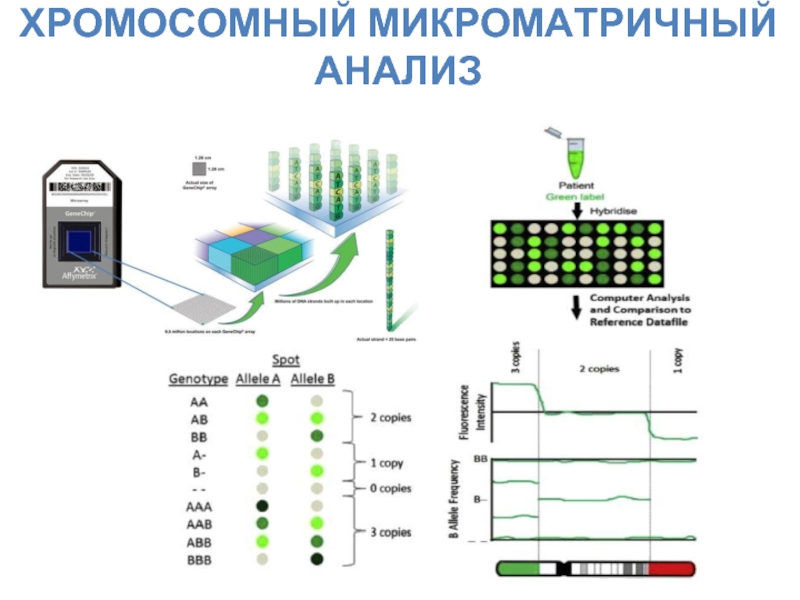
- 15. ХРОМОСОМНЫЙ МИКРОМАТРИЧНЫЙ АНАЛИЗ
- 16. Normal 2N AA:(0.5+0.5)=+1 AB: 0.5-0.5 = 0
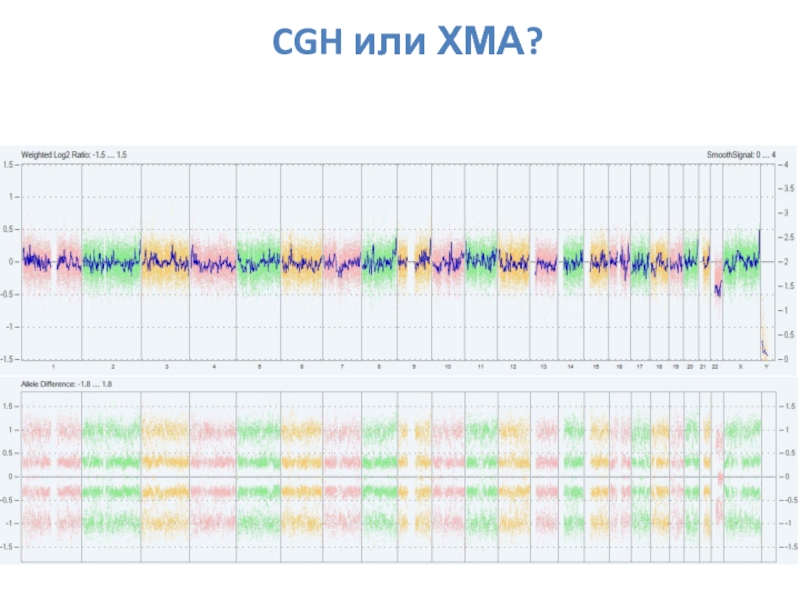
- 17. CGH или ХМА?
- 18. Распределение проб на микроматрицах разной плотности Микроматрица
- 19. МИКРОМАТРИЦЫ ВЫСОКОЙ, СРЕДНЕЙ И НИЗКОЙ ПЛОТНОСТИ 2,7 млн 750 тыс 180 тыс
- 20. ТЕОРЕТИЧЕСКОЕ ОПРЕДЕЛЕНИЕ РАЗРЕШАЮЩЕЙ СПОСОБНОСТИ МИКРОМАТРИЦ Размер определяемых
- 21. ОПРЕДЕЛЕНИЕ РАЗРЕШАЮЩЕЙ СПОСОБНОСТИ МИКРОМАТРИЦЫ В ХОДЕ ИССЛЕДОВАНИЙ
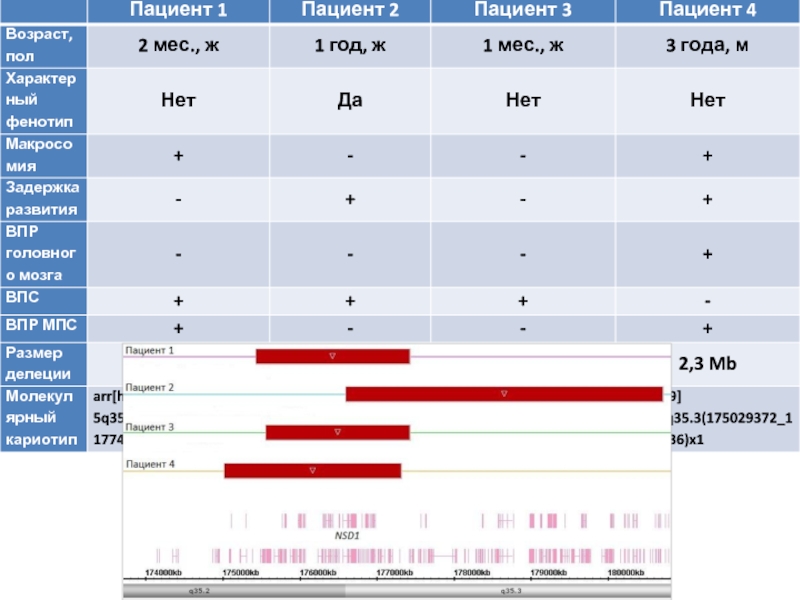
- 22. Пациент Л. Клиническая картина: Задержка развития, фенотип
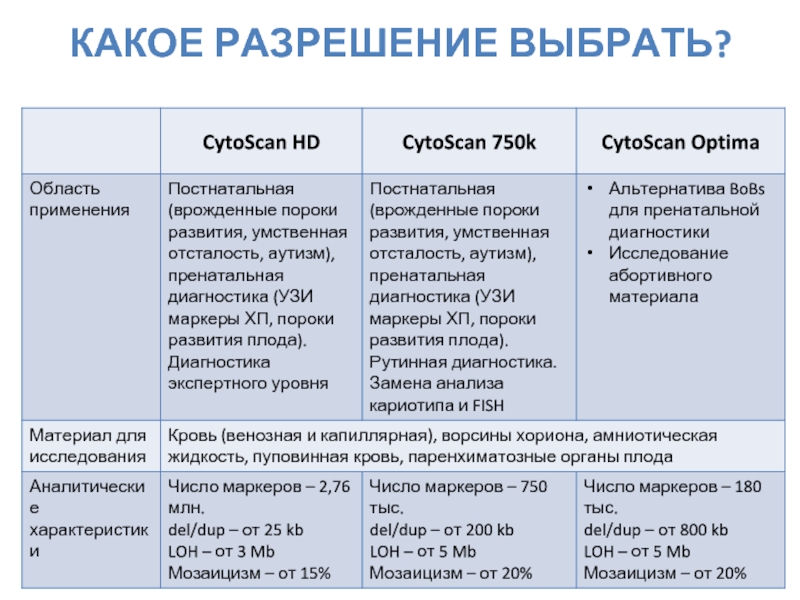
- 23. КАКОЕ РАЗРЕШЕНИЕ ВЫБРАТЬ?
- 24. DNA extraction Digestion Ligation PCR amplification QC
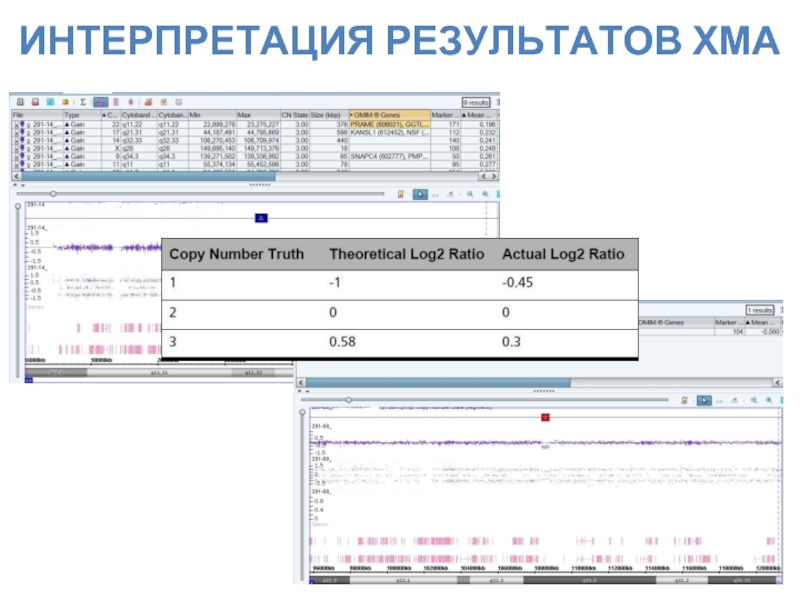
- 25. ИНТЕРПРЕТАЦИЯ РЕЗУЛЬТАТОВ ХМА
- 26. Вариация числа копий (CNV) – это изменение
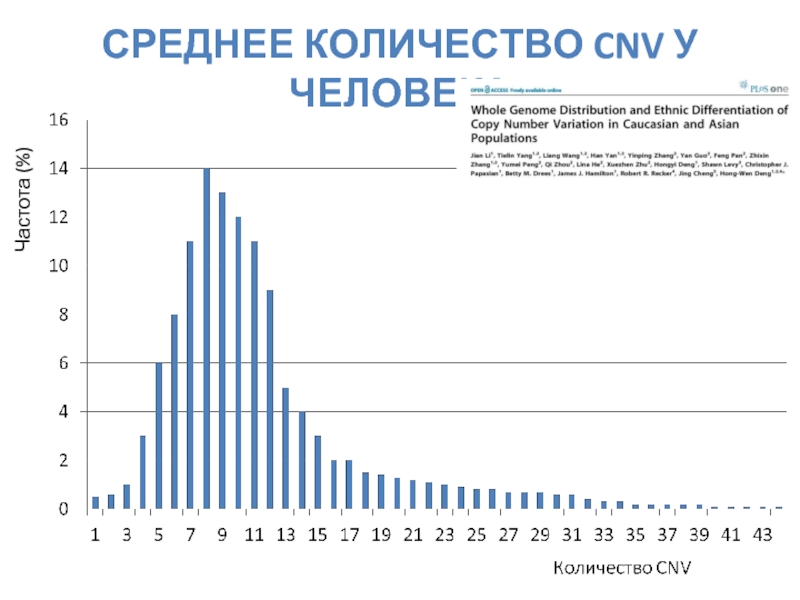
- 27. СРЕДНЕЕ КОЛИЧЕСТВО CNV У ЧЕЛОВЕКА Частота (%)
- 28. ОПРЕДЕЛЕНИЕ КЛИНИЧЕСКОЙ ЗНАЧИМОСТИ (КАУЗАТИВНОСТИ) CNVs Базы данных OMIM, ISCA, DECIPHER, GeneReviews, литературные данные -PubMed
- 29. Включена ли CNV в базы данных нормальных
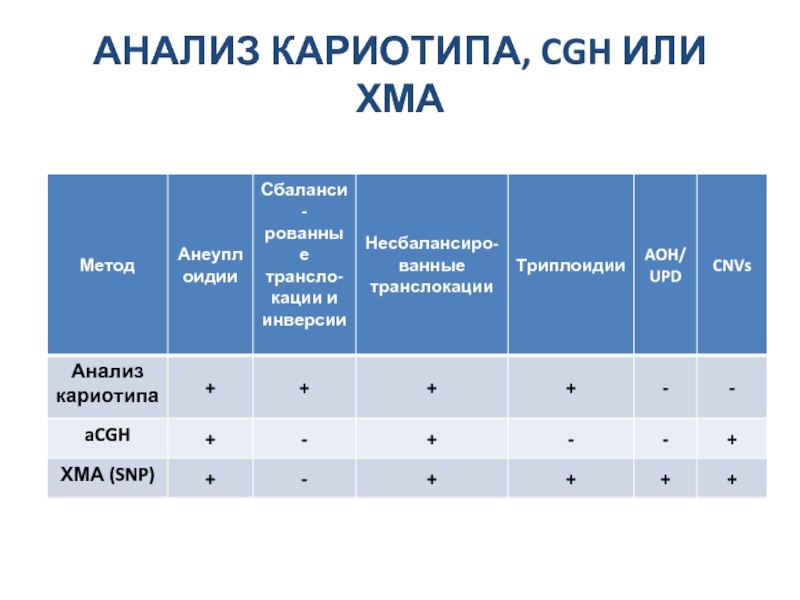
- 30. Включена ли CNV в базы данных нормальных
- 31. ПОПУЛЯЦИОННЫЕ БАЗЫ ДАННЫХ CNVs http://dgv.tcag.ca/dgv/app/home
- 32. Включена ли CNV в базы данных нормальных
- 33. Обнаружены микроделеции участков длинного плеча 17 хромосомы
- 34. Рекомендуется проводить дифференциальную диагностику с 5 синдромами:
- 35. Включена ли CNV в базы данных нормальных
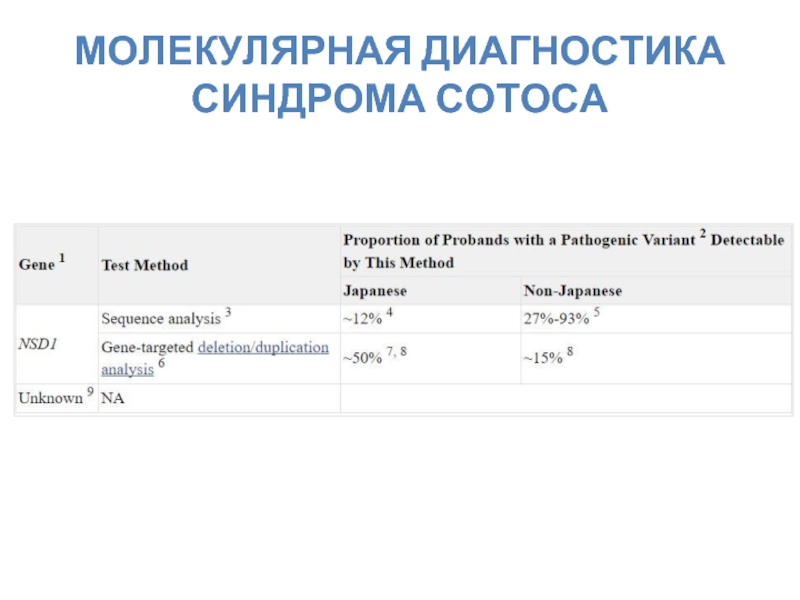
- 36. МОЛЕКУЛЯРНАЯ ДИАГНОСТИКА СИНДРОМА СОТОСА
- 38. ДИФФЕРЕНЦИАЛЬНАЯ ДИАГНОСТИКА СИНДРОМА СОТОСА Синдром Вивера Синдром
- 39. Включена ли CNV в базы данных нормальных
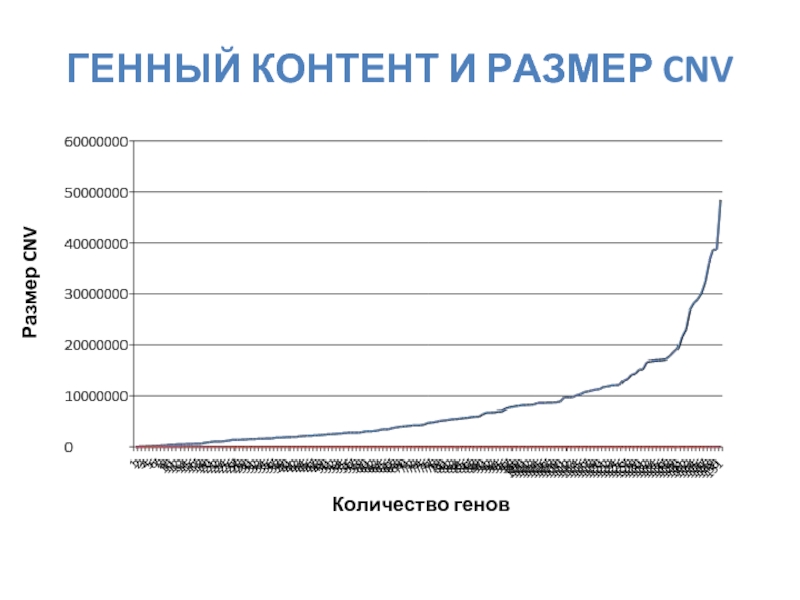
- 40. ГЕННЫЙ КОНТЕНТ И РАЗМЕР CNV Количество генов Размер CNV
- 41. Включена ли CNV в базы данных нормальных
- 42. CNV: НАСЛЕДУЕМАЯ ИЛИ DE NOVO? ХРОМОСОМНЫЙ МИКРОМАТРИЧНЫЙ
- 43. CNV: НАСЛЕДУЕМАЯ ИЛИ DE NOVO?
- 44. КЛАССИФИКАЦИЯ CNVs CNVs
- 45. ПАТОГЕННЫЕ CNVs Описаны в базах OMIM и
- 46. НЕПАТОГЕННЫЕ CNVs CNV была описана во многих
- 47. CNVs С НЕИЗВЕСТНОЙ КЛИНИЧЕСКОЙ ЗНАЧИМОСТЬЮ, ВЕРОЯТНО ПАТОГЕННАЯ
- 48. CNVs С НЕИЗВЕСТНОЙ КЛИНИЧЕСКОЙ ЗНАЧИМОСТЬЮ, ВЕРОЯТНО НЕПАТОГЕННАЯ
- 49. CNVs С НЕИЗВЕСТНОЙ КЛИНИЧЕСКОЙ ЗНАЧИМОСТЬЮ, НЕТ ПОДКЛАССИФИКАЦИИ
- 50. НЕОЖИДАННЫЕ КЛИНИЧЕСКИ ЗНАЧИМЫЕ РЕЗУЛЬТАТЫ, НЕ ОТНОСЯЩИЕСЯ К
- 51. НОСИТЕЛЬСТВО CNVs, СВЯЗАННЫХ С РЕЦЕССИВНЫМИ ЗАБОЛЕВАНИЯМИ Обнаружена
- 52. НОСИТЕЛЬСТВО CNVs, СВЯЗАННЫХ С ЗАБОЛЕВАНИЯМИ С ПОЗДНИМ
- 53. НОСИТЕЛЬСТВО CNVs, СВЯЗАННЫХ С РИСКОМ НЕОПЛАЗИИ Сообщать
- 54. ИНТЕРПРЕТАЦИЯ ВНЗ
- 55. Пациентка Р., 3 года Клинический диагноз: Буллезный
- 56. ФЕНОТИП ПАЦИЕНТА Фенотип синдромов попа-дающих в зону
- 57. Пациентка Р., 3 года Клинический диагноз: Буллезный
- 58. ЗАПИСЬ РЕЗУЛЬТАТОВ ХРОМОСОМНОГО МИКРОМАТРИЧНОГО АНАЛИЗА arr(1-22)x2,(X,Y)x1 –
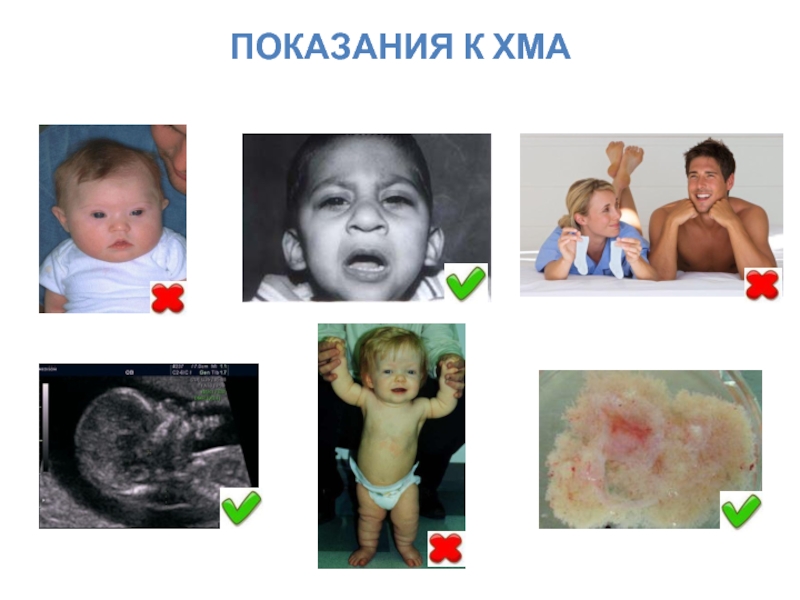
- 59. ПОКАЗАНИЯ К ХМА
- 60. ХМА В ПОСТНАТАЛЬНОЙ ДИАГНОСТИКЕ ХМА является тестом
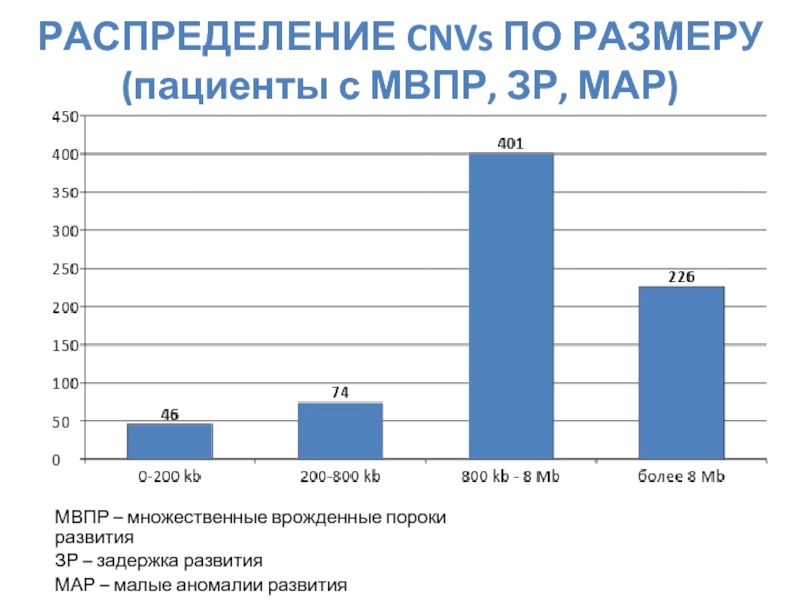
- 61. РАСПРЕДЕЛЕНИЕ CNVs ПО РАЗМЕРУ (пациенты с МВПР,
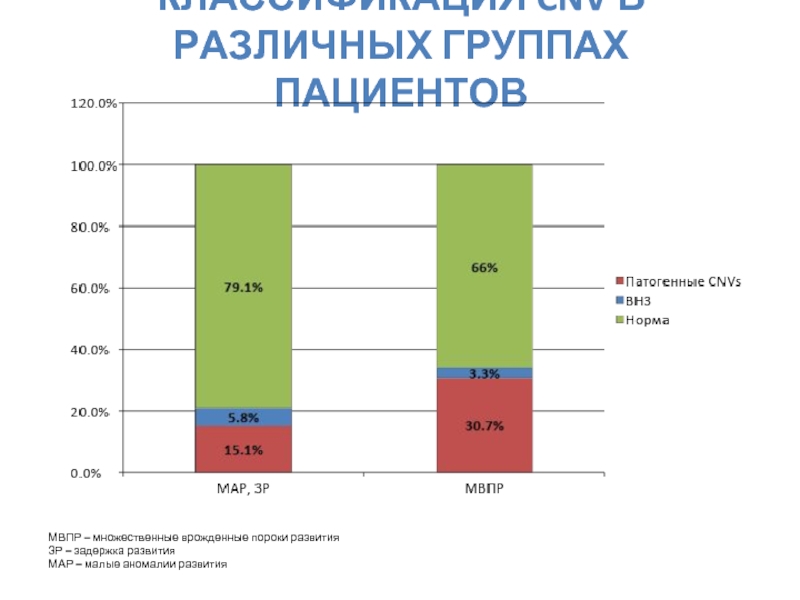
- 62. КЛАССИФИКАЦИЯ CNV В РАЗЛИЧНЫХ ГРУППАХ ПАЦИЕНТОВ МВПР
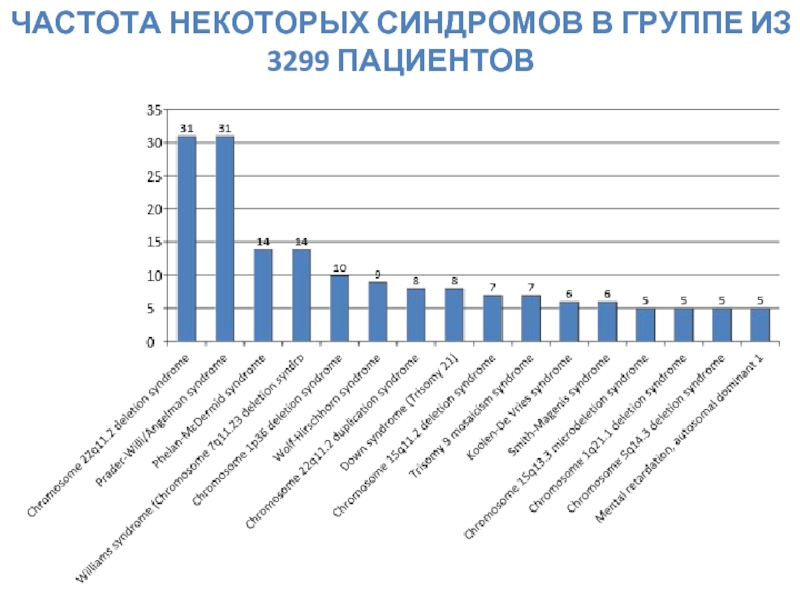
- 63. ЧАСТОТА НЕКОТОРЫХ СИНДРОМОВ В ГРУППЕ ИЗ 3299 ПАЦИЕНТОВ
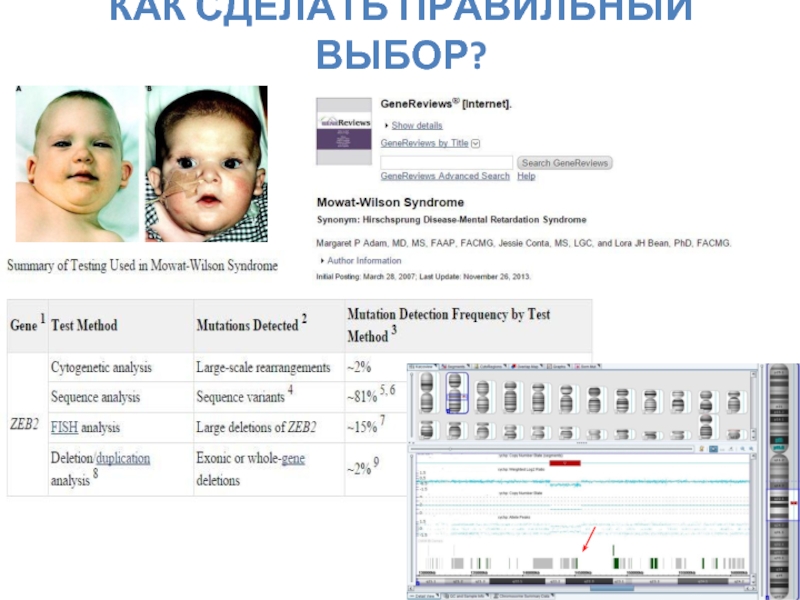
- 64. КАК СДЕЛАТЬ ПРАВИЛЬНЫЙ ВЫБОР?
- 65. Сбалансированные хромосомные перестройки (транслокации, инверсии) Ожидаем ли
- 66. НЕТ! НУЖНО ЛИ ПОДТВЕРЖДАТЬ РЕЗУЛЬТАТЫ ХМА? Система
- 67. Клинический экзом Тщательное прочтение избранных
- 68. Плотность анализируемых участков в клиническом экзоме не
- 69. Эффективность выявления CNV в полноэкзомных данных по
- 70. Секвенирование экзома, хотя уже подтвердило свою высокую
- 71. ГЕНЕТИЧЕСКОЕ КОНСУЛЬТИРОВАНИЕ ПО РЕЗУЛЬТАТАМ ХМА ПРОБЛЕМЫ
- 72. ГЕНЕТИЧЕСКОЕ КОНСУЛЬТИРОВАНИЕ ПО РЕЗУЛЬТАТАМ ХМА ОСОБЕННОСТИ
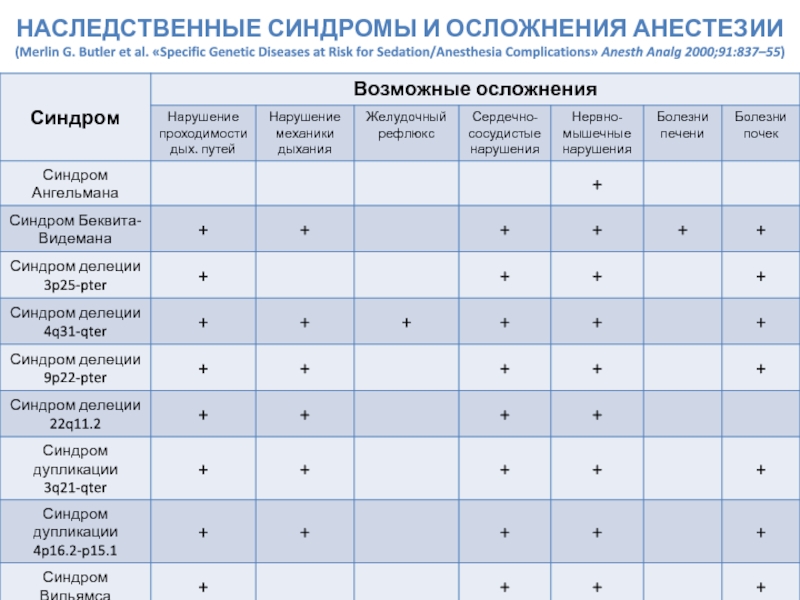
- 73. НАСЛЕДСТВЕННЫЕ СИНДРОМЫ И ОСЛОЖНЕНИЯ АНЕСТЕЗИИ (Merlin
- 74. ХРОМОСОМНЫЙ МИКРОМАТРИЧНЫЙ АНАЛИЗ В ПРЕНАТАЛЬНОЙ ДИАГНОСТИКЕ ПОКАЗАНИЯ
- 75. ХМА в качестве замены анализа кариотипа рекомендован
- 76. ЭФФЕКТИВНОСТЬ ХМА В ПРЕНАТАЛЬНОЙ ДИАГНОСТИКЕ УЗ-аномалии –
- 77. АНАЛИЗ CNV В ТКАНИ ОПУХОЛИ ERBB2
- 78. АНАЛИЗ КАРИОТИПА, CGH ИЛИ ХМА
Слайд 2СИНДРОМ - ?
ВПС
Особенности фенотипа
Аномалии соединительной ткани
Задержка развития
Особенности характера и поведения
Задержка роста
Эндокринные
Слайд 3СИНДРОМ - ?
Неонатальная гипотония
Нормальный или ускоренный рост
Тяжелая задержка развития
Нормальная окружность головы
Множественные
Слайд 51 970,0 млн USD
469,2 млн USD
dup 17p11.2
del 1p36
del 9q34.3
del 3q29
del 22q13.3
dup7q11.23
del
dup 22q11.2
Слайд 6ХРОМОСОМНАЯ ПАТОЛОГИЯ
ОПРЕДЕЛЕНИЕ
Хромосомная патология – любое нарушение числа и/или структуры хромосом
Слайд 7ХРОМОСОМНАЯ ПАТОЛОГИЯ
КЛИНИЧЕСКОЕ ЗНАЧЕНИЕ
числовые и структурные аномалии хромосом (за исключением сбалансированных) патогенны
характеризуются
являются причиной задержки развития/умственной отсталости
часто является причиной потери беременности
многие микроделеционные синдромы не имеют четкого фенотипа и часто остаются недиагностированными
не могут быть вылечены
могут возникать повторно при следующих беременностях
являются показанием для консультации врача-генетика
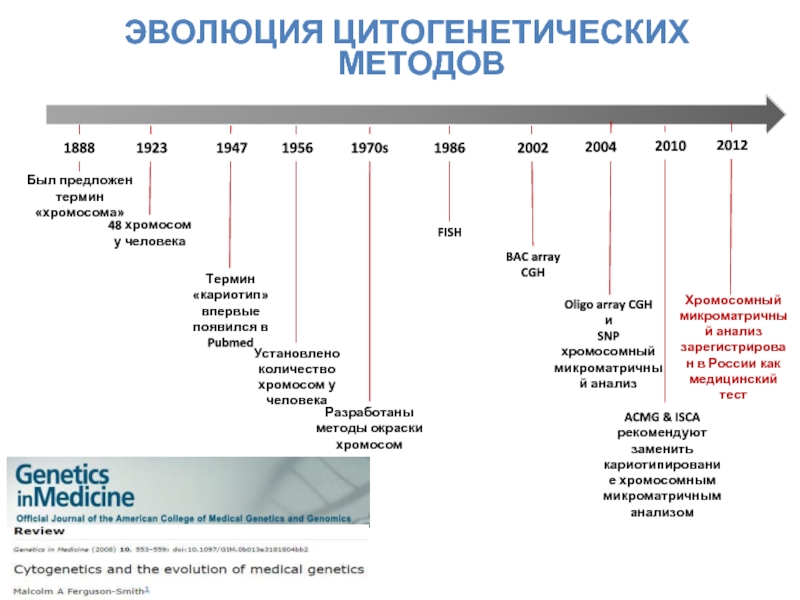
Слайд 8ЭВОЛЮЦИЯ ЦИТОГЕНЕТИЧЕСКИХ МЕТОДОВ
Термин «кариотип» впервые появился в Pubmed
Был предложен
термин «хромосома»
48 хромосом
у
Разработаны методы окраски хромосом
Установлено количество хромосом у человека
FISH
BAC array CGH
Oligo array CGH
и
SNP хромосомный микроматричный анализ
1947
1888
1923
1970s
1956
1986
2002
2004
ACMG & ISCA рекомендуют заменить кариотипирование хромосомным микроматричным анализом
2010
2012
Хромосомный микроматричный анализ зарегистрирован в России как медицинский тест
Слайд 12Хромосомный микроматричный анализ – молекулярно-цитогенетический метод анализа вариаций числа копий ДНК
Синонимы: молекулярное кариотипирование, сравнительная геномная гибридизация на чипах, молекулярно-цитогенетическое исследование
ОПРЕДЕЛЕНИЕ
Слайд 13ИСТОРИЯ
Первый прототип микроматицы на микроскопном стекле
1993
1995 Первая опубликованная научная работа с использованием микроматриц (Shena et al.)
1996 Промышленный выпуск микроматриц (Affymetrix)
1997 Полногеномный экспрессионный анализ S. cerevisiae (De Risi et al.)
1999 Молекулярная классификация опухоли и использованием микроматриц (Goluet al.)
2000 Молекулярный фенотип опухли (Pat Brown et Al.)
2003 Производители микроматриц Affymrtrix, Illumina, Agilent, Nimblegen (Roche) и мн. др.
2004 Полногеномный анализ на одной микроматрице (Affymetrix)
2005 Первая микроматрица одобренная FDA (Amplichip, Roche)
2008 Широкое использование разных типов микроматриц – SNP, CNV, Expression, ChIP, Tilling и пр.
2011 >100,000 исследований с использованием микроматриц в молекулярной цитогенетике.
Выявление причин ВПР и ЗПМР в 20-25% случаев.
Слайд 16Normal 2N
AA:(0.5+0.5)=+1
AB: 0.5-0.5 = 0
BB: 0-(0.5+0.5)=-1
Loss 1N
A:0.5-0 =+0.5
B:0-0.5=-0.5
Mosaic loss
Mosaic gain
Loss of
AA
AB
BB
A
B
Gain 3N
AAA:(0.5+0.5+0.5)=+1.5
AAB: (0.5-0.5 )-0.5 = +0,5
ABB: 0.5-(0.5+0.5)=-0.5
BBB: 0-(0.5+0.5+0,5)=-1.5
AAA
AAB
ABB
BBB
Each point represent a single SNP interrogated by A or B alleles probes and assigned an arbitrary fluorescence uni of haploid locus (single allele) =0,5
A-B=?
АНАЛИЗ СИГНАЛА ОТ SNP
Слайд 18Распределение проб на микроматрицах разной плотности
Микроматрица 400К – 90%
Клиническая достоверность
Слайд 20ТЕОРЕТИЧЕСКОЕ ОПРЕДЕЛЕНИЕ РАЗРЕШАЮЩЕЙ СПОСОБНОСТИ МИКРОМАТРИЦ
Размер определяемых микроделеций и микродупликаций зависит от
1. Волнистость (Waviness SD)
глобальная мера распределения зондов, которая нечувствительна
к вариациям распределения на коротких расстояниях и сфокусирована
на больших расстояниях
2. Различие в интенсивности сигнала между двумя соседними маркерами (MAPD - Median of the Absolute values of all Pairwise Differences)
При Волнистости <=0,12 и MAPD <=0,25 точность определения CNVs 99,5% достигается при включении в анализ 20 маркеров для делеций и 50 маркеров для дупликаций.
Для генов, рекомендованных ISCA плотность маркеров составляет 1 маркер на 384 нуклеотида
Таким образом, теоретически возможная минимальная разрешающая способность ХМА составляет 7680 для делеций и 19200 для дупликаций
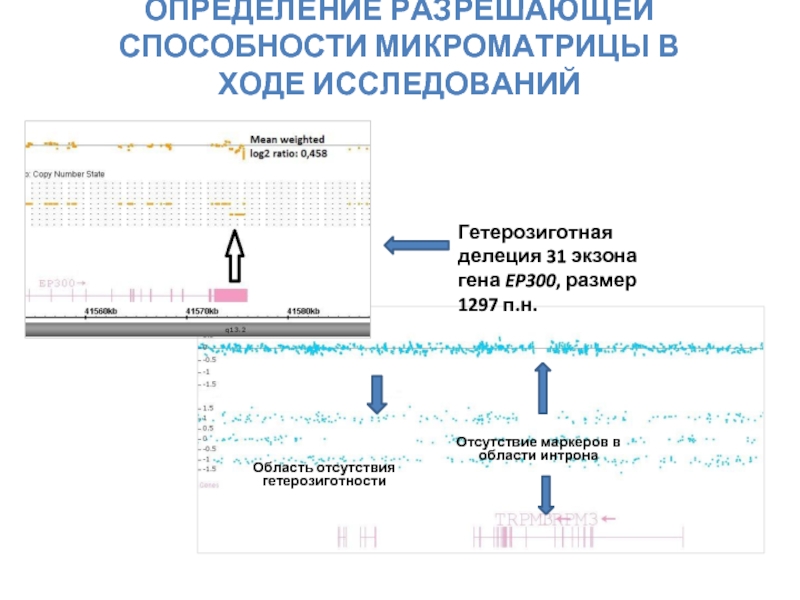
Слайд 21ОПРЕДЕЛЕНИЕ РАЗРЕШАЮЩЕЙ СПОСОБНОСТИ МИКРОМАТРИЦЫ В ХОДЕ ИССЛЕДОВАНИЙ
Отсутствие маркеров в области интрона
Область
Гетерозиготная делеция 31 экзона гена EP300, размер 1297 п.н.
Слайд 22Пациент Л.
Клиническая картина: Задержка развития, фенотип синдрома Рубинштейна-Тейби
Молекулярный кариотип: arr[hg19] 16p13.3(3828749_3833104)x1
Обнаружена делеция двух экзонов гена CREBBP, ассоциированного с синдромом Рубинштейна-Тейби
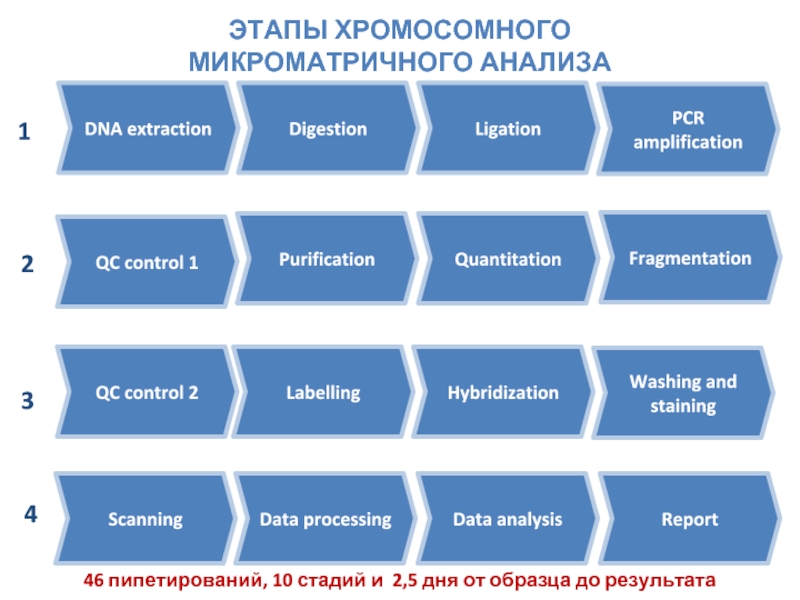
Слайд 24DNA extraction
Digestion
Ligation
PCR amplification
QC control 1
Purification
Fragmentation
Quantitation
QC control 2
Labelling
Hybridization
Washing and staining
Scanning
Data processing
Data analysis
Report
1
2
3
4
ЭТАПЫ
46 пипетирований, 10 стадий и 2,5 дня от образца до результата
Слайд 26Вариация числа копий (CNV) – это изменение числа копий определенного сегмента
Термин «CNV» не подразумевает исходно обязательной клинической значимости, поэтому CNVs классифицируются на патогенные, непатогенные и CNVs с неизвестной значимостью.
ВАРИАЦИИ ЧИСЛА КОПИЙ ДНК (CNV)
Слайд 28ОПРЕДЕЛЕНИЕ КЛИНИЧЕСКОЙ ЗНАЧИМОСТИ (КАУЗАТИВНОСТИ) CNVs
Базы данных OMIM, ISCA, DECIPHER, GeneReviews, литературные
Слайд 29Включена ли CNV в базы данных нормальных вариаций?
Содержит ли CNV область,
Содержит ли CNV ген, делеция или дупликация которого являются причиной известных синдромов?
Каков общий генный контент или размер CNV?
Является ли обнаруженная CNV вариацией de novo?
Является ли CNV наследуемой?
ЧТО ОПРЕДЕЛЯЕТ КЛИНИЧЕСКУЮ ЗНАЧИМОСТЬ CNV?
Слайд 30Включена ли CNV в базы данных нормальных вариаций?
Содержит ли CNV область,
Содержит ли CNV ген, делеция или дупликация которого являются причиной известных синдромов?
Каков общий генный контент или размер CNV?
Является ли обнаруженная CNV вариацией de novo?
Является ли CNV наследуемой?
ЧТО ОПРЕДЕЛЯЕТ КЛИНИЧЕСКУЮ ЗНАЧИМОСТЬ CNV?
Слайд 32Включена ли CNV в базы данных нормальных вариаций?
Содержит ли CNV область,
Содержит ли CNV ген, делеция или дупликация которого являются причиной известных синдромов?
Каков общий генный контент или размер CNV?
Является ли обнаруженная CNV вариацией de novo?
Является ли CNV наследуемой?
ЧТО ОПРЕДЕЛЯЕТ КЛИНИЧЕСКУЮ ЗНАЧИМОСТЬ CNV?
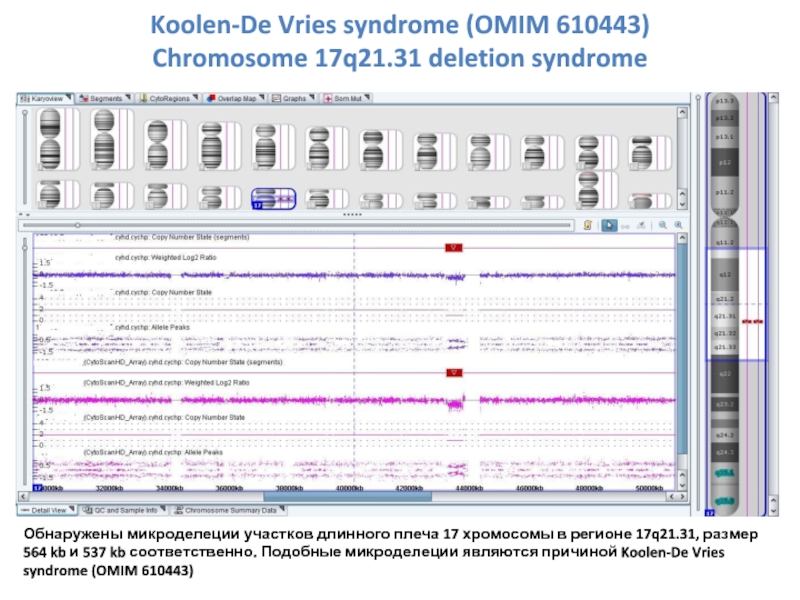
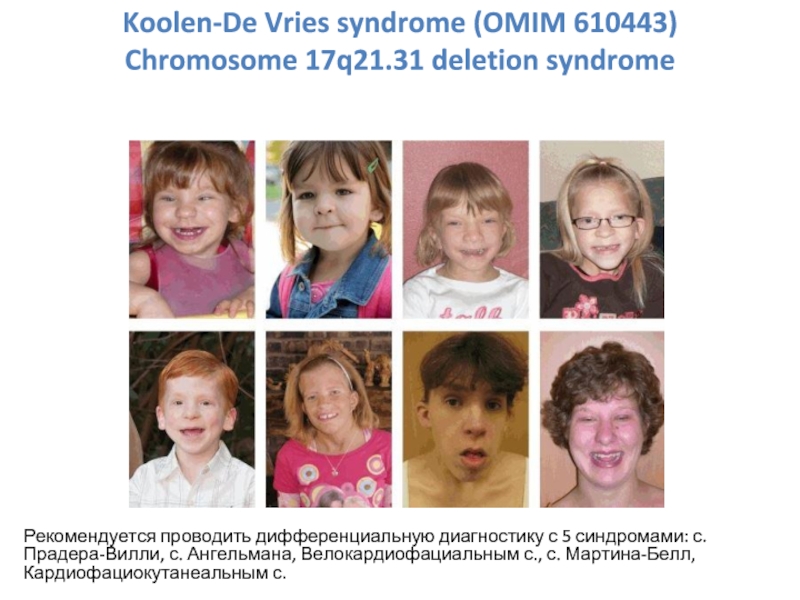
Слайд 33Обнаружены микроделеции участков длинного плеча 17 хромосомы в регионе 17q21.31, размер
Koolen-De Vries syndrome (OMIM 610443)
Chromosome 17q21.31 deletion syndrome
Слайд 34Рекомендуется проводить дифференциальную диагностику с 5 синдромами: с. Прадера-Вилли, с. Ангельмана,
Koolen-De Vries syndrome (OMIM 610443)
Chromosome 17q21.31 deletion syndrome
Слайд 35Включена ли CNV в базы данных нормальных вариаций?
Содержит ли CNV область,
Содержит ли CNV ген, делеция или дупликация которого являются причиной известных синдромов?
Каков общий генный контент или размер CNV?
Является ли обнаруженная CNV вариацией de novo?
Является ли CNV наследуемой?
ЧТО ОПРЕДЕЛЯЕТ КЛИНИЧЕСКУЮ ЗНАЧИМОСТЬ CNV?
Слайд 38ДИФФЕРЕНЦИАЛЬНАЯ ДИАГНОСТИКА СИНДРОМА СОТОСА
Синдром Вивера
Синдром Беквита-Видемана
Синдром Банаян-Райли-Рувалькаба
Дупликации 4p
Мозаичная трисомия 20p11.2-р12.1
Синдром делеции
Слайд 39Включена ли CNV в базы данных нормальных вариаций?
Содержит ли CNV область,
Содержит ли CNV ген, делеция или дупликация которого являются причиной известных синдромов?
Каков общий генный контент или размер CNV?
Является ли обнаруженная CNV вариацией de novo?
Является ли CNV наследуемой?
ЧТО ОПРЕДЕЛЯЕТ КЛИНИЧЕСКУЮ ЗНАЧИМОСТЬ CNV?
Слайд 41Включена ли CNV в базы данных нормальных вариаций?
Содержит ли CNV область,
Содержит ли CNV ген, делеция или дупликация которого являются причиной известных синдромов?
Каков общий генный контент или размер CNV?
Является ли обнаруженная CNV вариацией de novo?
Является ли CNV наследуемой?
ЧТО ОПРЕДЕЛЯЕТ КЛИНИЧЕСКУЮ ЗНАЧИМОСТЬ CNV?
Слайд 42CNV: НАСЛЕДУЕМАЯ ИЛИ DE NOVO?
ХРОМОСОМНЫЙ МИКРОМАТРИЧНЫЙ АНАЛИЗ ПОЗВОЛЯЕТ ВЫЯВЛЯТЬ МИКРОДЕЛЕЦИИ…
Пациент К.,
Беременность 21 неделя. ВПС: тетрада Фалло у плода.
Материал: амниотическая жидкость
Обнаружена микроделеция 3p26.3. Размер: 1,4 Mb
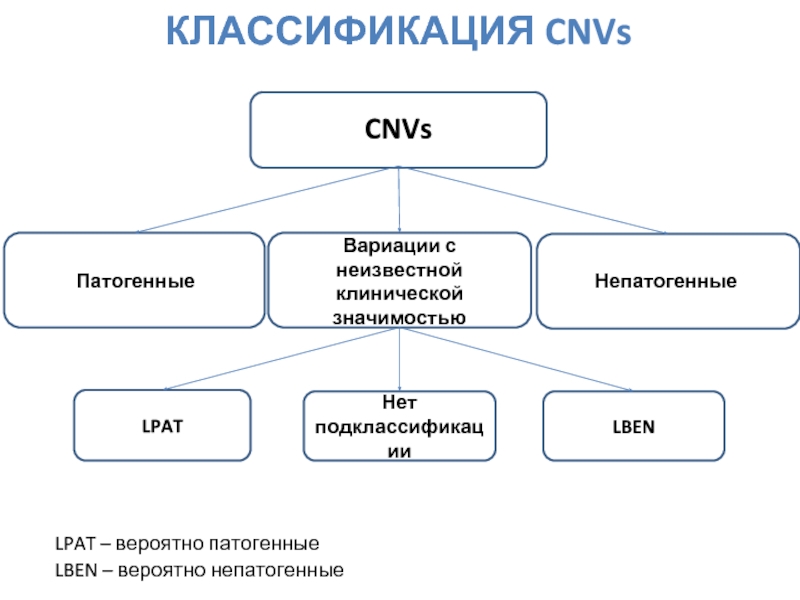
Слайд 44КЛАССИФИКАЦИЯ CNVs
CNVs
Патогенные
Непатогенные
Вариации с неизвестной клинической значимостью
LPAT
LBEN
Нет подклассификации
LPAT – вероятно патогенные
LBEN –
Слайд 45ПАТОГЕННЫЕ CNVs
Описаны в базах OMIM и ORPHANET как делеционный/дупликационный синдром
Описаны в
Слайд 46НЕПАТОГЕННЫЕ CNVs
CNV была описана во многих рецензируемых публикациях или курируемых базах
Слайд 47CNVs С НЕИЗВЕСТНОЙ КЛИНИЧЕСКОЙ ЗНАЧИМОСТЬЮ, ВЕРОЯТНО ПАТОГЕННАЯ
CNV описана в единственной рецензируемой
CNV описана как вероятно патогенная в рецензируемых публикациях и базах данных у пациентов со сходными клиническими проявлениями
В регионе CNV имеется ген с известной функцией, нарушение которой может привести к клиническим проявлениям, сходным с наблюдаемыми у пациента.
Слайд 48CNVs С НЕИЗВЕСТНОЙ КЛИНИЧЕСКОЙ ЗНАЧИМОСТЬЮ, ВЕРОЯТНО НЕПАТОГЕННАЯ
В районе CNV отсутствуют гены
CNV присутствует у небольшого количества индивидов в общепопуляционных базах данных, но не является распространённым полиморфизмом.
Слайд 49CNVs С НЕИЗВЕСТНОЙ КЛИНИЧЕСКОЙ ЗНАЧИМОСТЬЮ, НЕТ ПОДКЛАССИФИКАЦИИ
CNV содержит гены, однако неизвестно,
CNV представлена во многих противоречащих друг другу публикациях и/или базах данных, из-за чего невозможно сделать вывод о ее клинической значимости.
Слайд 50НЕОЖИДАННЫЕ КЛИНИЧЕСКИ ЗНАЧИМЫЕ РЕЗУЛЬТАТЫ, НЕ ОТНОСЯЩИЕСЯ К ПРИЧИНЕ НАПРАВЛЕНИЯ
Носительство CNVs, связанных
Носительство CNVs, связанных с заболеваниями с поздним началом или не диагностированными состояниями
Носительство вариантов, ассоциированных с риском неоплазии
Слайд 51НОСИТЕЛЬСТВО CNVs, СВЯЗАННЫХ С РЕЦЕССИВНЫМИ ЗАБОЛЕВАНИЯМИ
Обнаружена мутация в гене, связанном с
Обнаружена мутация в гене, связанном с рецессивным заболеванием с симптоматикой, совпадающей с имеющейся у пациента.
Слайд 52НОСИТЕЛЬСТВО CNVs, СВЯЗАННЫХ С ЗАБОЛЕВАНИЯМИ С ПОЗДНИМ НАЧАЛОМ
Сообщать или нет?
Может
Если сообщать, то о каких именно состояниях?
Слайд 53НОСИТЕЛЬСТВО CNVs, СВЯЗАННЫХ С РИСКОМ НЕОПЛАЗИИ
Сообщать или нет?
Какие варианты могут быть
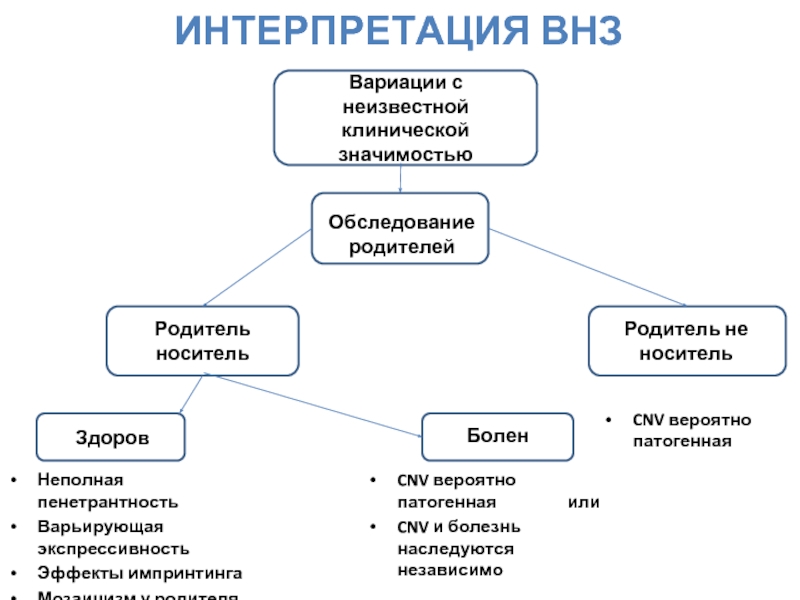
Слайд 54ИНТЕРПРЕТАЦИЯ ВНЗ
Вариации с неизвестной клинической значимостью
Обследование родителей
Родитель не носитель
Родитель носитель
Здоров
Болен
Неполная пенетрантность
Варьирующая
Эффекты импринтинга
Мозаицизм у родителя
CNV вероятно патогенная или
CNV и болезнь наследуются независимо
CNV вероятно патогенная
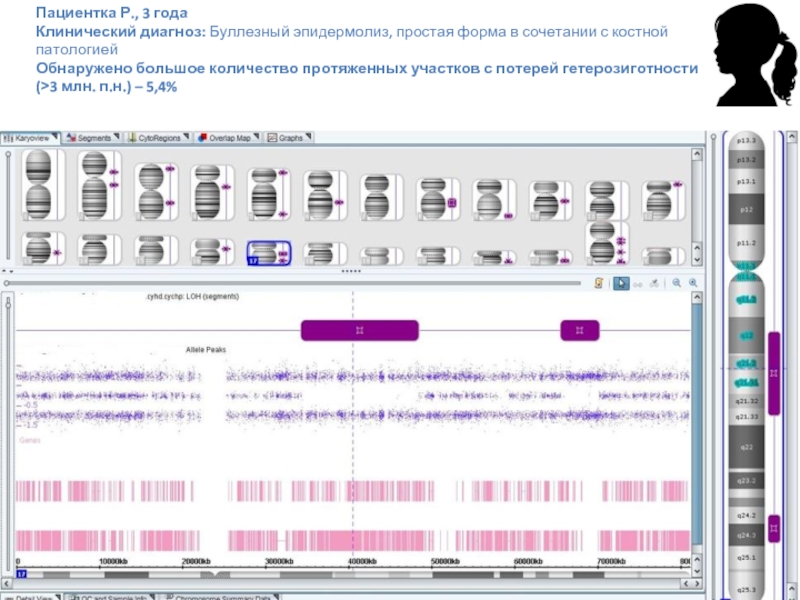
Слайд 55Пациентка Р., 3 года Клинический диагноз: Буллезный эпидермолиз, простая форма в сочетании
Слайд 56ФЕНОТИП ПАЦИЕНТА
Фенотип синдромов попа-дающих в зону потери гетерозиготности
=
=>
Анализ мутаций связанных с
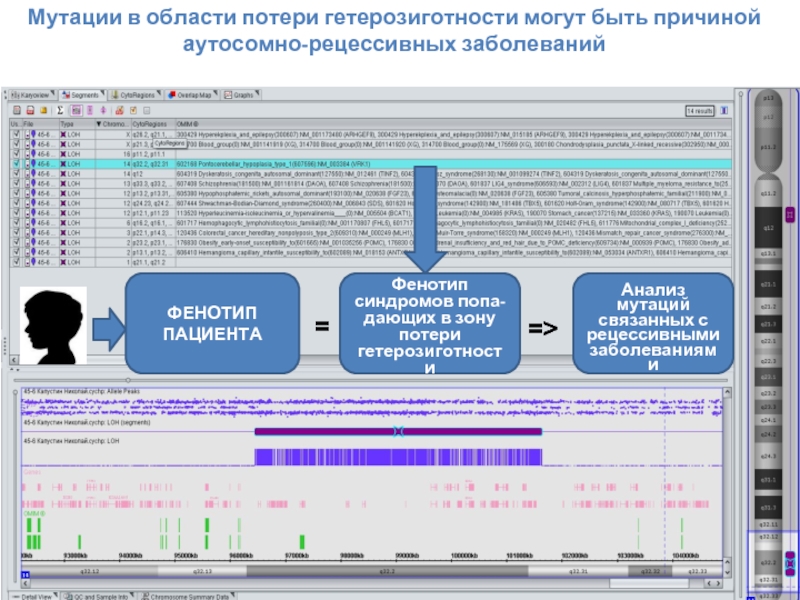
Мутации в области потери гетерозиготности могут быть причиной аутосомно-рецессивных заболеваний
Слайд 57Пациентка Р., 3 года Клинический диагноз: Буллезный эпидермолиз, простая форма в сочетании
Слайд 58ЗАПИСЬ РЕЗУЛЬТАТОВ ХРОМОСОМНОГО МИКРОМАТРИЧНОГО АНАЛИЗА
arr(1-22)x2,(X,Y)x1 – норма, мужской пол
arr(1-22,X)x2 – норма,
arr[hg19] 7p21.1p14.1(16817164_40301877)x3
arr – array, указывает на то, каким методом был выполнен анализ
[hg19] – версия референсного генома
7p21.1p14.1 – локализация точек разрывов
(16817164_40301877) – геномные координаты
х3 – число копий в анализируемой области
Слайд 60ХМА В ПОСТНАТАЛЬНОЙ ДИАГНОСТИКЕ
ХМА является тестом первой линии в постнатальной диагностике
Слайд 61РАСПРЕДЕЛЕНИЕ CNVs ПО РАЗМЕРУ
(пациенты с МВПР, ЗР, МАР)
МВПР – множественные врожденные
ЗР – задержка развития
МАР – малые аномалии развития
Слайд 62КЛАССИФИКАЦИЯ CNV В РАЗЛИЧНЫХ ГРУППАХ ПАЦИЕНТОВ
МВПР – множественные врожденные пороки развития
ЗР
МАР – малые аномалии развития
Слайд 65Сбалансированные хромосомные перестройки (транслокации, инверсии)
Ожидаем ли мы увидеть сбалансированную перестройку у
Точковые мутации
Болезни экспансии тринуклеотидных повторов
Микроделеции/микродупликации, размер которых меньше разрешающей способности микроматрицы
ОГРАНИЧЕНИЯ МЕТОДА
Слайд 66НЕТ!
НУЖНО ЛИ ПОДТВЕРЖДАТЬ РЕЗУЛЬТАТЫ ХМА?
Система Геноскан 3000 является единственной апробированной полногеномной
Слайд 67
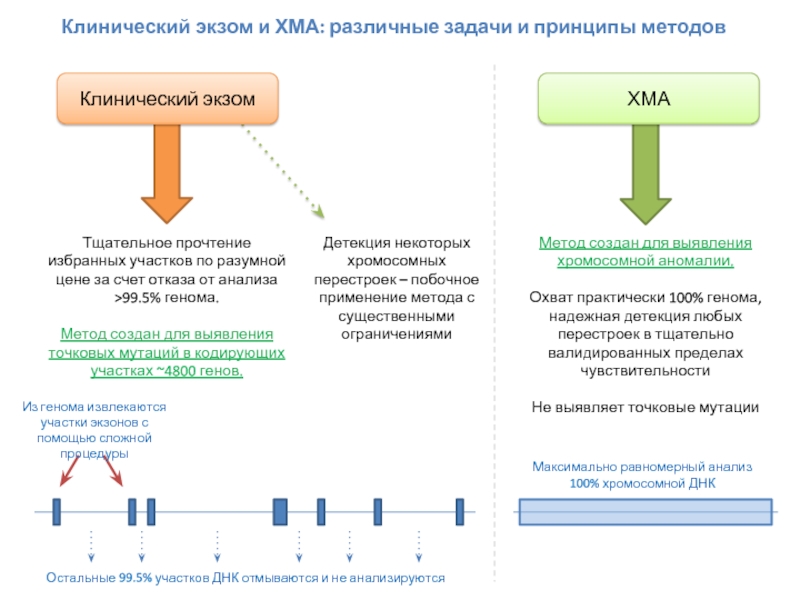
Клинический экзом
Тщательное прочтение избранных участков по разумной цене за счет отказа
Метод создан для выявления точковых мутаций в кодирующих участках ~4800 генов.
Детекция некоторых хромосомных перестроек – побочное применение метода с существенными ограничениями
ХМА
Метод создан для выявления хромосомной аномалии.
Охват практически 100% генома, надежная детекция любых перестроек в тщательно валидированных пределах чувствительности
Не выявляет точковые мутации
Из генома извлекаются участки экзонов с помощью сложной процедуры
Остальные 99.5% участков ДНК отмываются и не анализируются
Максимально равномерный анализ
100% хромосомной ДНК
Клинический экзом и ХМА: различные задачи и принципы методов
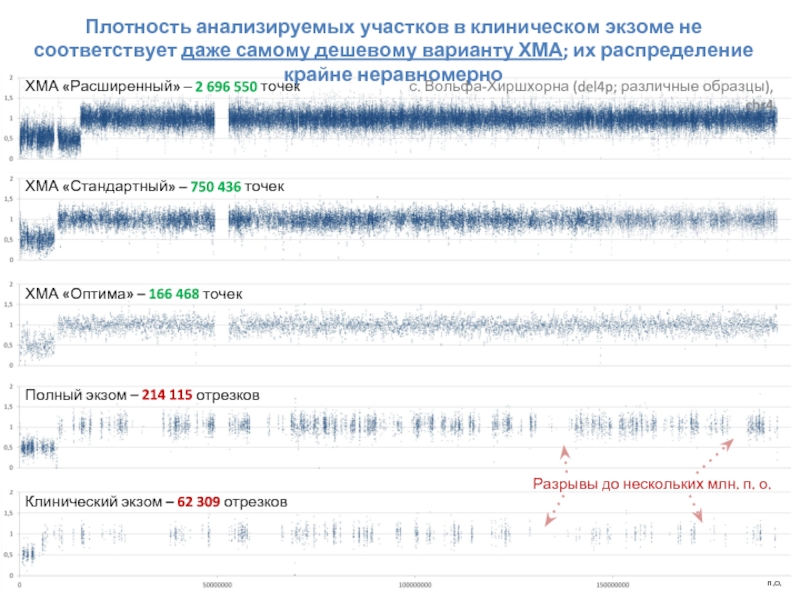
Слайд 68Плотность анализируемых участков в клиническом экзоме не соответствует даже самому дешевому
Клинический экзом – 62 309 отрезков
Полный экзом – 214 115 отрезков
ХМА «Оптима» – 166 468 точек
ХМА «Стандартный» – 750 436 точек
ХМА «Расширенный» – 2 696 550 точек
Разрывы до нескольких млн. п. о.
с. Вольфа-Хиршхорна (del4p; различные образцы), chr4
п.о.
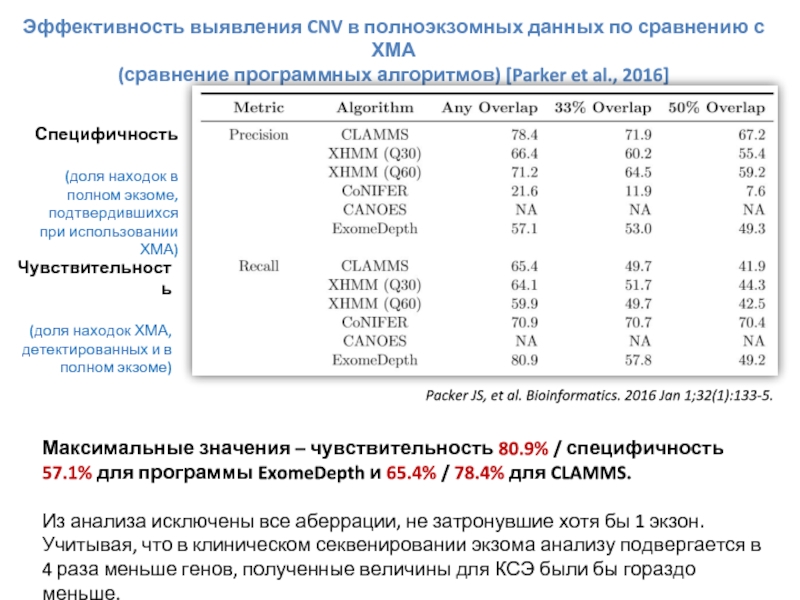
Слайд 69Эффективность выявления CNV в полноэкзомных данных по сравнению с ХМА
(сравнение программных
Специфичность
(доля находок в полном экзоме, подтвердившихся при использовании ХМА)
Чувствительность
(доля находок ХМА, детектированных и в полном экзоме)
Максимальные значения – чувствительность 80.9% / специфичность 57.1% для программы ExomeDepth и 65.4% / 78.4% для CLAMMS.
Из анализа исключены все аберрации, не затронувшие хотя бы 1 экзон.
Учитывая, что в клиническом секвенировании экзома анализу подвергается в 4 раза меньше генов, полученные величины для КСЭ были бы гораздо меньше.
Packer JS, et al. Bioinformatics. 2016 Jan 1;32(1):133-5.
Слайд 70 Секвенирование экзома, хотя уже подтвердило свою высокую эффективность в выявлении точковых
ХМА, напротив, давно признан «золотым стандартом» молекулярной цитогенетики, как за рубежом, так и в России; в частности, МГНЦ РАМН в 2015 г. опубликовано описание медицинской технологии «Использование хромосомного микроматричного анализа для повышения эффективности медико-генетического консультирования».
Это значит, что даже при успешной детекции аберраций с помощью NGS их необходимо обязательно подтверждать референсным методом для постановки диагноза – в случае совсем крупных делеций или дупликаций таковым может послужить кариотип, но в большинстве случаев подтверждающим методом будет выступать ХМА.
Слайд 71ГЕНЕТИЧЕСКОЕ КОНСУЛЬТИРОВАНИЕ ПО РЕЗУЛЬТАТАМ ХМА
ПРОБЛЕМЫ
Назначения врача не выполняются
Завышенные ожидания от результатов
Пациенты получают результаты исследования до того, как с ними может ознакомиться врач
Нарушение требований к подготовке заключений
Незнание или непонимание содержимого заключения
Слайд 72ГЕНЕТИЧЕСКОЕ КОНСУЛЬТИРОВАНИЕ ПО РЕЗУЛЬТАТАМ ХМА
ОСОБЕННОСТИ
Некоторые патогенные CNV наследуются
Могут иметь неполную пенетрантность
Выявление
Выявление патогенных CNV у пробанда – основание для рекомендации пренатальной диагностики
Ложное отцовство, ВРТ с донорскими клетками
Слайд 73НАСЛЕДСТВЕННЫЕ СИНДРОМЫ И ОСЛОЖНЕНИЯ АНЕСТЕЗИИ (Merlin G. Butler et al. «Specific
Слайд 74ХРОМОСОМНЫЙ МИКРОМАТРИЧНЫЙ АНАЛИЗ В ПРЕНАТАЛЬНОЙ ДИАГНОСТИКЕ
ПОКАЗАНИЯ
Высокий риск ХП по результатам НИПТ
Ребенок с хромосомной патологией в семье
Носительство сбалансированной хромосомной перестройки у одного из родителей
Высокий риск ХП по результатам скрининга
УЗ-маркеры ХП
ВПР плода
ВОЗМОЖНОСТИ
Анеуплоидии, триплоидии
Микроделеции/микродупликации
Несбалансированные транслокации
Потеря гетерозиготности, ОРД
Слайд 75ХМА в качестве замены анализа кариотипа рекомендован при выявлении пороков развития
врач должен обсудить преимущества и ограничения анализа кариотипа и ХМА с пациентами, которым показана инвазивная процедура
проведение пре- и посттестового консультирования должен проводить врач, имеющий опыт в интерпретации результатов ХМА
пациенты должны быть информированы об ограничениях ХМА, а также о возможности выявлять признаки родства родителей
при выявлении ВНЗ рекомендована консультация эксперта имеющего доступ к базам данных для выявления генотип-фенотипических корреляций
не рекомендуется использование ХМА в качестве теста первой линии для диагностики причин потери беременности в первом триместре
Слайд 76ЭФФЕКТИВНОСТЬ ХМА В ПРЕНАТАЛЬНОЙ ДИАГНОСТИКЕ
УЗ-аномалии – 6%
Другие причины – 1,7%
от общего
УЗ-аномалии – 6,5%
Возраст старше 35 лет – 1%
Другие причины – 1,1%
от общего числа случаев с нормальным кариотипом плода














![Пациент Л.Клиническая картина: Задержка развития, фенотип синдрома Рубинштейна-ТейбиМолекулярный кариотип: arr[hg19] 16p13.3(3828749_3833104)x1 Размер 4355 пн.Обнаружена делеция](/img/tmb/2/151525/b7214c2ae77960b8eb3d735701864f4c-800x.jpg)






















![ЗАПИСЬ РЕЗУЛЬТАТОВ ХРОМОСОМНОГО МИКРОМАТРИЧНОГО АНАЛИЗАarr(1-22)x2,(X,Y)x1 – норма, мужской полarr(1-22,X)x2 – норма, женский полarr[hg19] 7p21.1p14.1(16817164_40301877)x3arr –](/img/tmb/2/151525/e767c21fc07a0cf5d185adcea7f3bb6b-800x.jpg)