- Главная
- Разное
- Дизайн
- Бизнес и предпринимательство
- Аналитика
- Образование
- Развлечения
- Красота и здоровье
- Финансы
- Государство
- Путешествия
- Спорт
- Недвижимость
- Армия
- Графика
- Культурология
- Еда и кулинария
- Лингвистика
- Английский язык
- Астрономия
- Алгебра
- Биология
- География
- Детские презентации
- Информатика
- История
- Литература
- Маркетинг
- Математика
- Медицина
- Менеджмент
- Музыка
- МХК
- Немецкий язык
- ОБЖ
- Обществознание
- Окружающий мир
- Педагогика
- Русский язык
- Технология
- Физика
- Философия
- Химия
- Шаблоны, картинки для презентаций
- Экология
- Экономика
- Юриспруденция
Хроматин, гетерохроматин, упаковка генома презентация
Содержание
- 1. Хроматин, гетерохроматин, упаковка генома
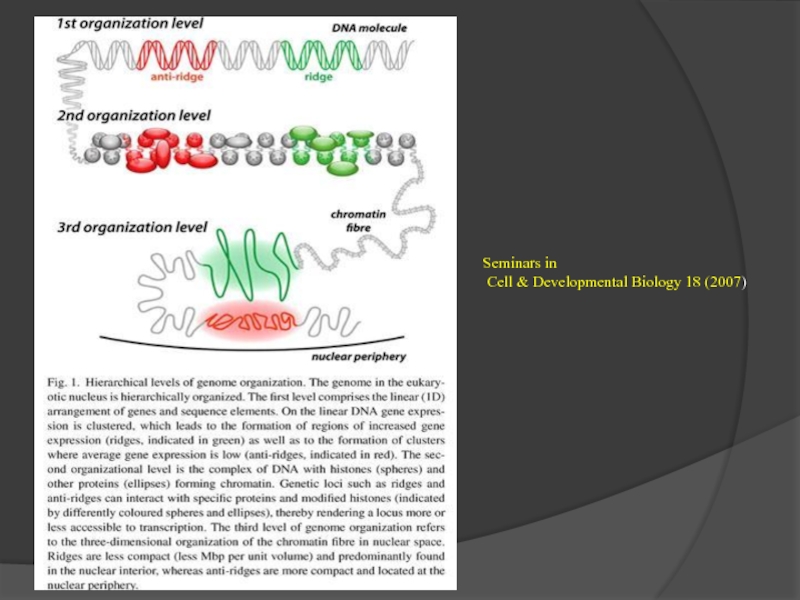
- 2. Seminars in Cell & Developmental Biology 18 (2007)
- 3. 4 2
- 5. Уровни упаковки генома Двойная спираль 10 нм
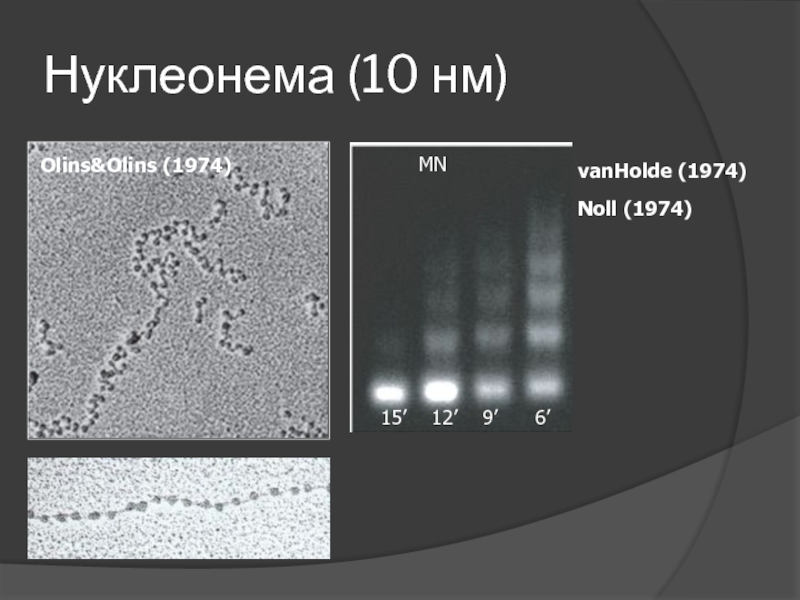
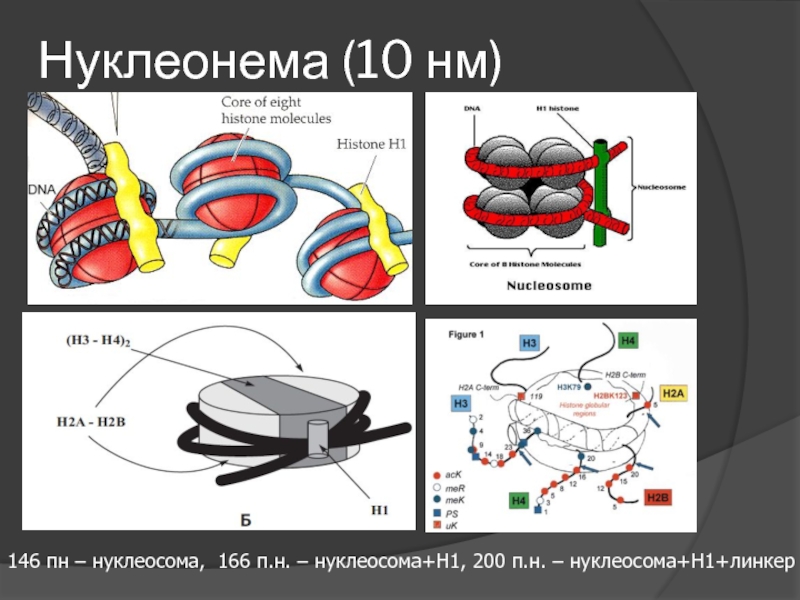
- 6. Нуклеонема (10 нм) 15’ 12’
- 7. Нуклеонема (10 нм) 146 пн – нуклеосома, 166 п.н. – нуклеосома+Н1, 200 п.н. – нуклеосома+Н1+линкер
- 8. Фазирование нуклеосом Фиксированное расположение нуклеосом Вирус
- 9. Гистоны Основные типы H1 (H5 у
- 10. Вариантные формы гистонов H1, H2A, H2B, H3
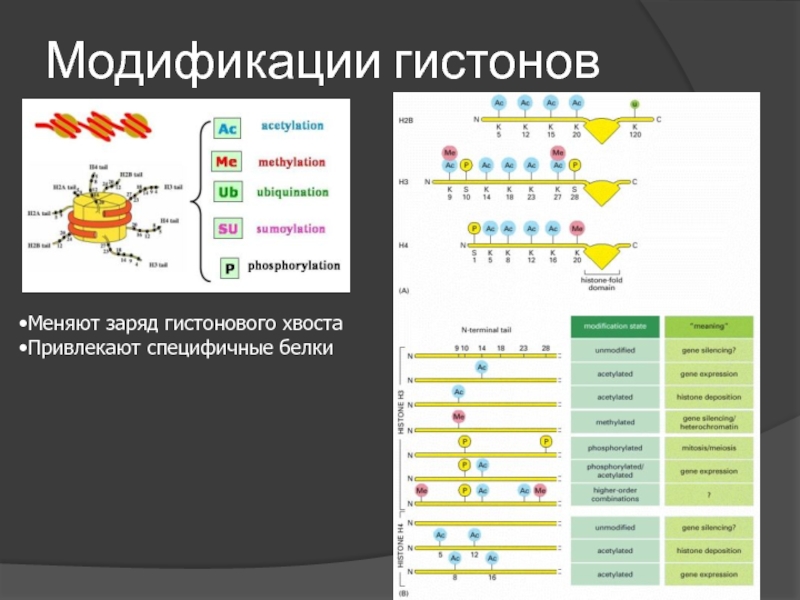
- 11. Модификации гистонов Меняют заряд гистонового хвоста Привлекают специфичные белки
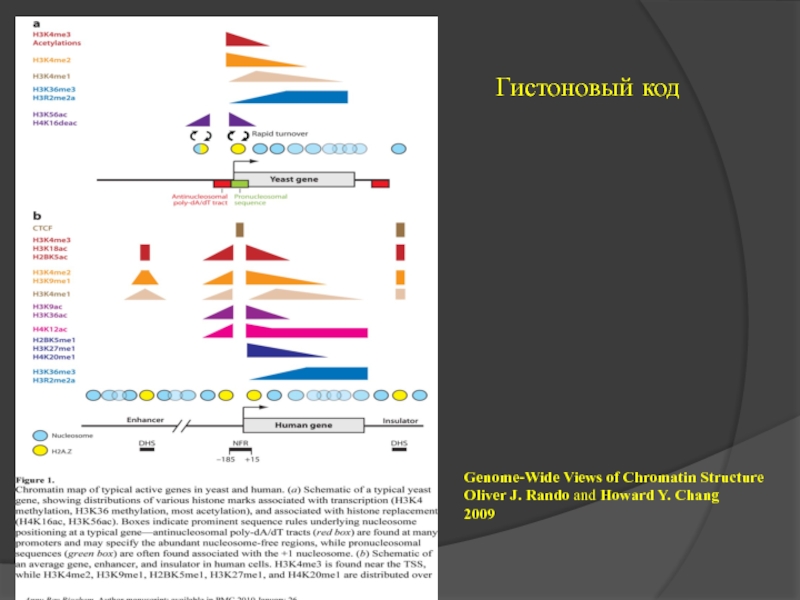
- 12. «Гистоновый код» Совокупность сигналов, экспонированных на поверхности
- 13. Гистоновый код Genome-Wide Views of Chromatin Structure Oliver J. Rando and Howard Y. Chang 2009
- 14. Histone 3 barcode (Hake, Allis; 2006)
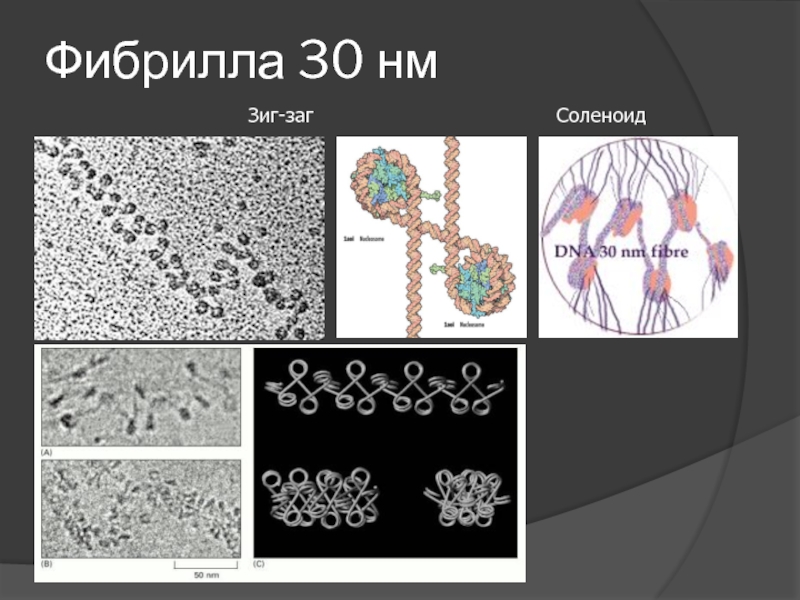
- 15. Фибрилла 30 нм Зиг-заг
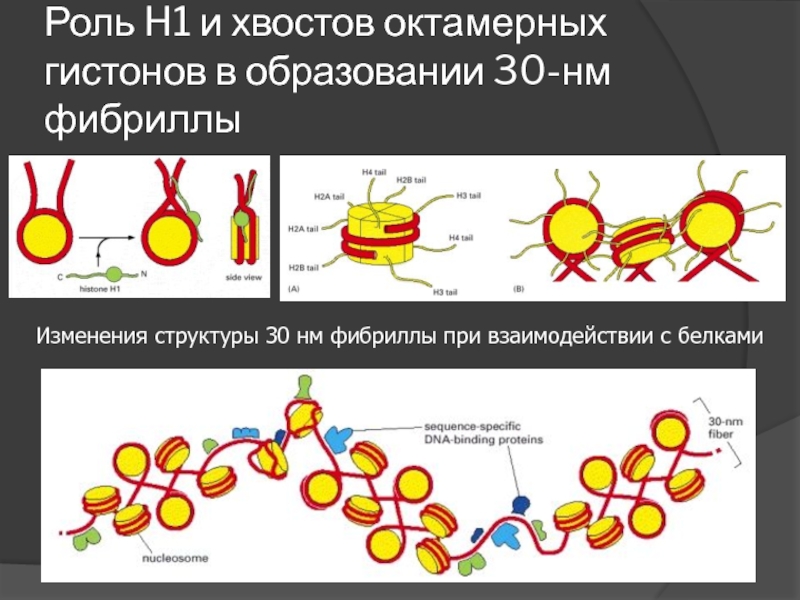
- 16. Роль H1 и хвостов октамерных гистонов в
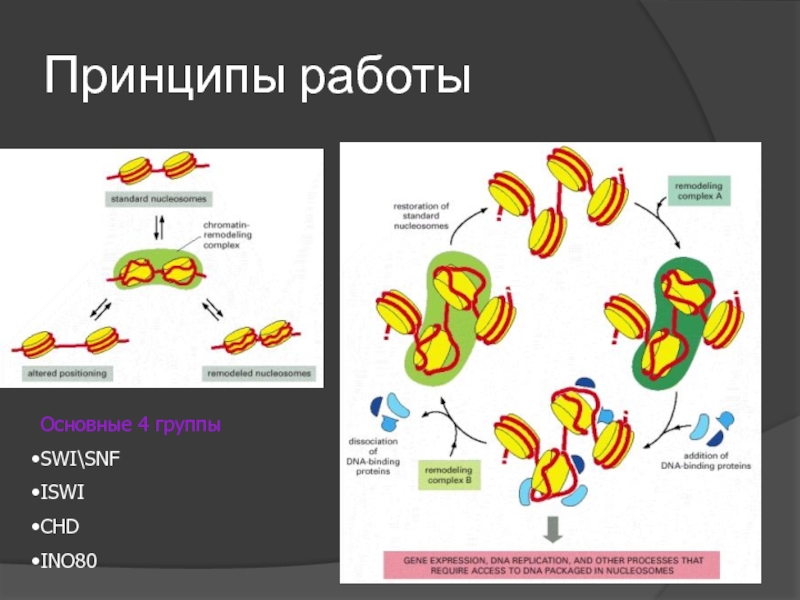
- 17. АТФ-зависимые комплексы ремоделирования хроматина Перемещение нуклеосом вдоль
- 18. Принципы работы Основные 4 группы SWI\SNF ISWI CHD INO80
- 19. Высшие уровни упаковки хроматина Соленоид? Латеральное взаимодействие соседних фибрилл? Радиально-петлевая модель ?
- 20. Радиально-петлевая модель Двойная спираль 10 нм фибрилла
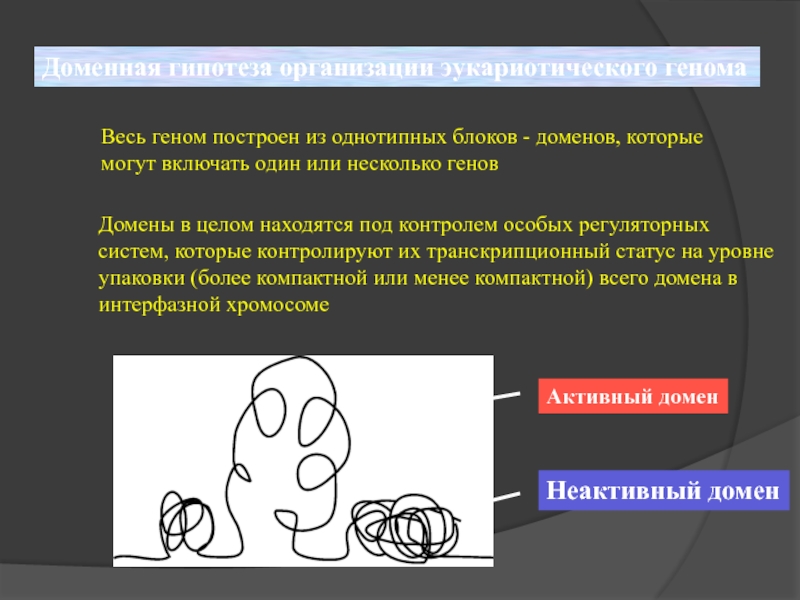
- 21. Доменная гипотеза организации эукариотического генома Весь геном
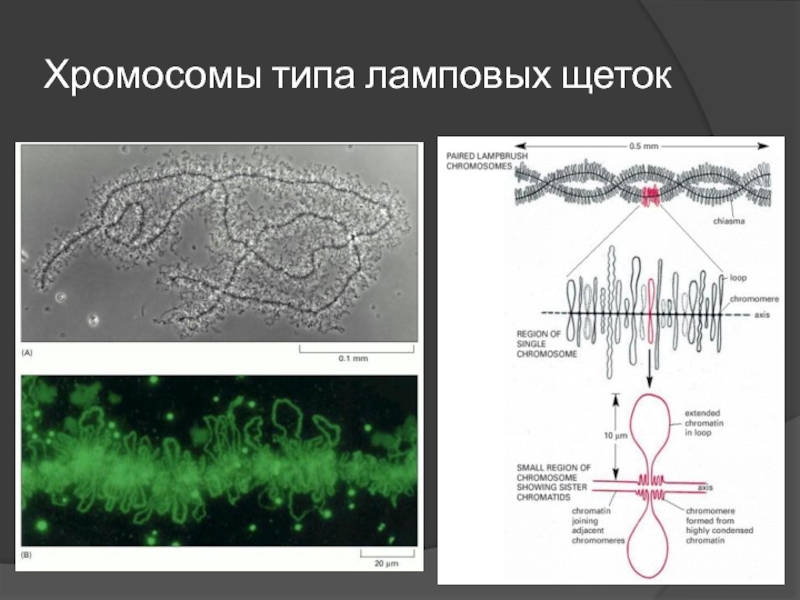
- 22. Хромосомы типа ламповых щеток
- 23. Хромосомный скэффолд
- 24. 1947. Mirsky, Ris. Фракция остаточных белков при
- 25. ЯМ – это…. Компоненты ЯМ ламина,
- 26. Препарат ядерного матрикса Фиброгранулярная сеть эдектронноплотные
- 29. Непрерывный клеточный скелет Figure 1 Transmission
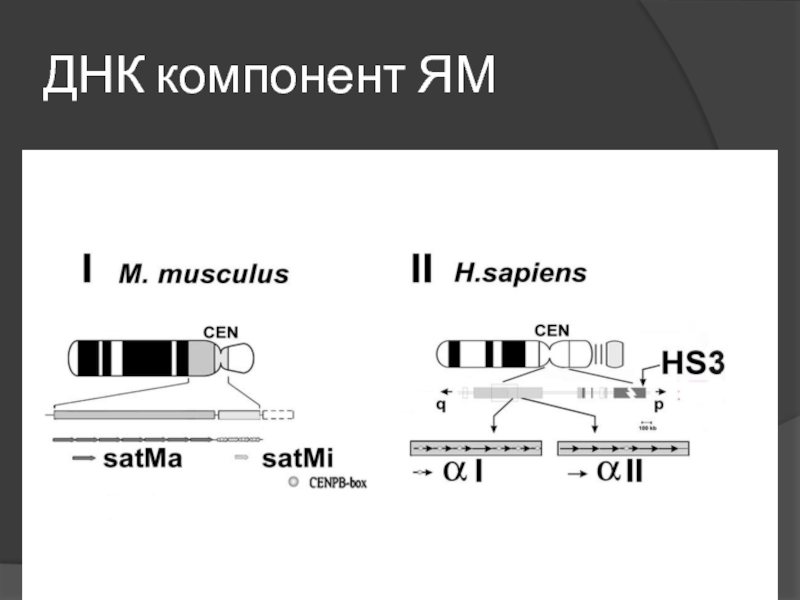
- 30. ДНК компонент ЯМ
- 32. Regulating the mammalian genome: the role of
- 33. Alexey Gavrilov, Sergey V. Razin, and
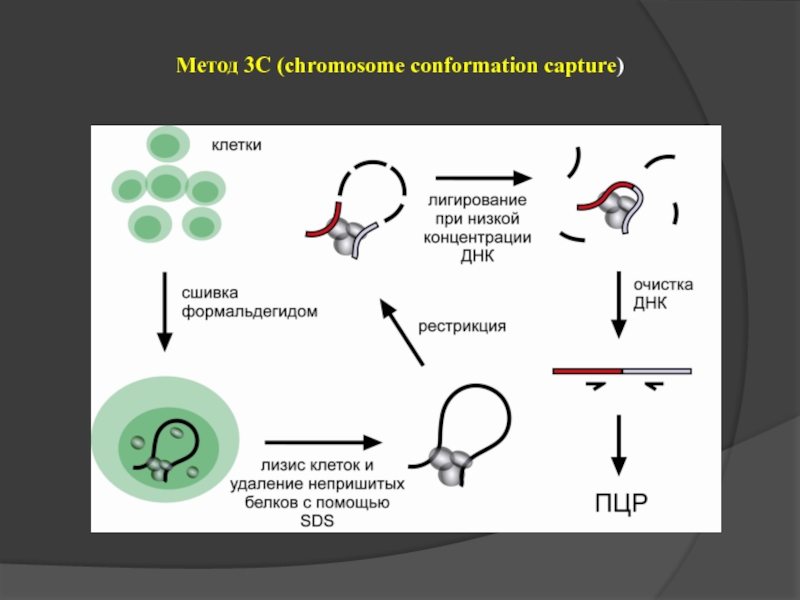
- 34. Метод 3С (chromosome conformation capture)
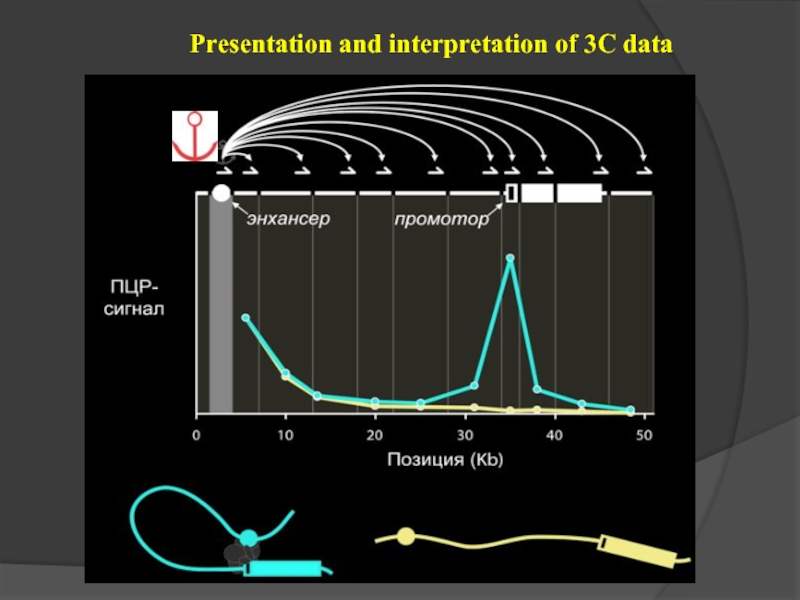
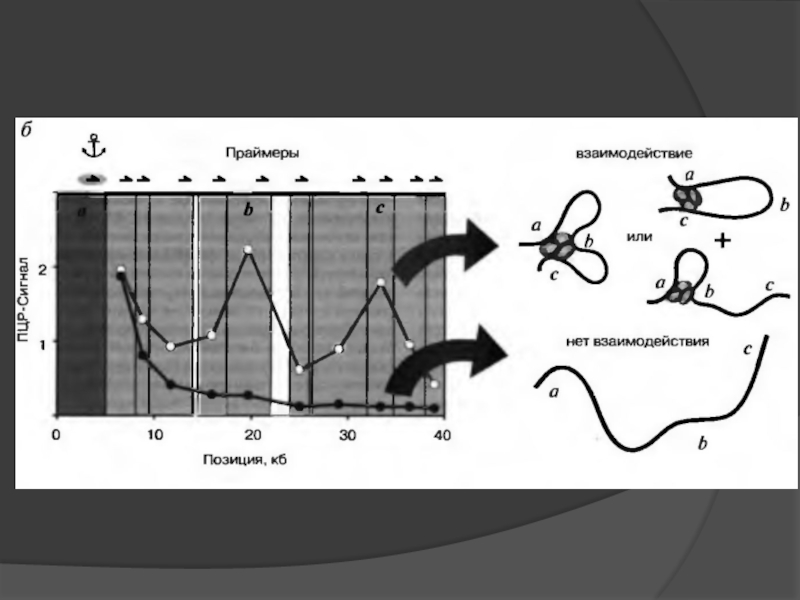
- 35. Presentation and interpretation of 3C data
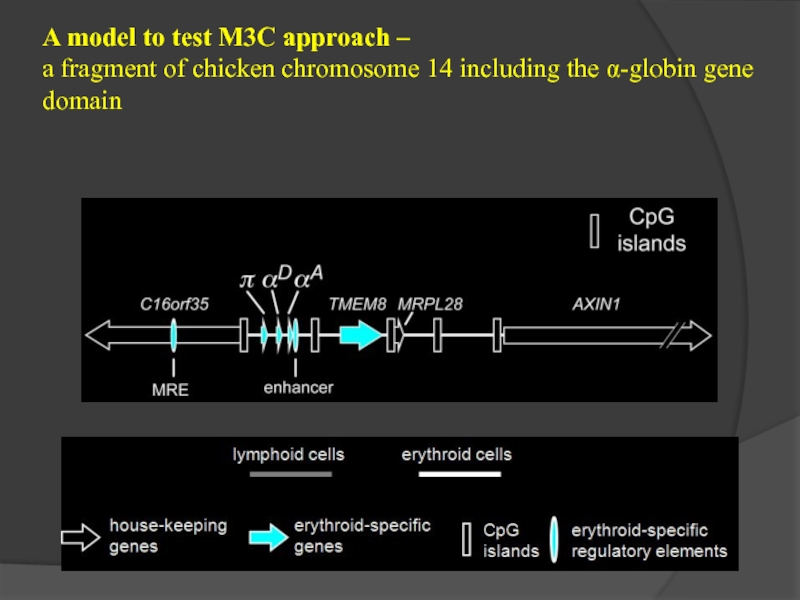
- 37. A model to test M3C approach –
- 38. In chicken genome
- 39. Matrix 3C (M3C) – an experimental procedure
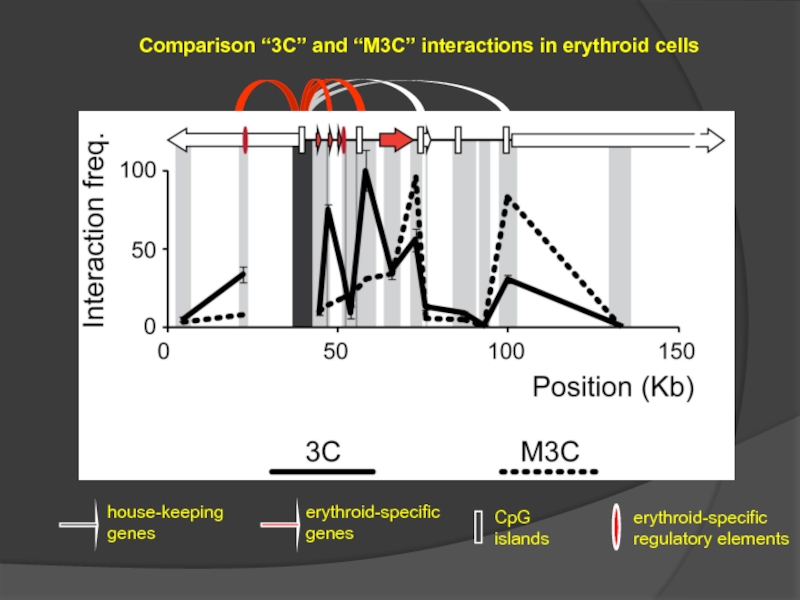
- 40. Comparison “3C” and “M3C” interactions in erythroid cells
- 41. Chromatin hub conception The chromatin hub
- 43. Белки ЯМ
- 44. Белки ЯМ
- 45. Nuclear matrins : Identification of
- 46. The Functional Levels of Higher Order Chromatin Organization Are Associated With the Nuclear Matrix
- 47. 1. The nuclear matrix is composed of
- 48. 4. Matrin Cyp (cyclophilin) a ~88 kDa
- 49. Nuclear Matrix Proteins (cont.)
- 50. Saf-a/hnRNP U a b c d Lobov et al., 2000 Lobov et al., 2000
- 51. Saf-a/hnRNP U specifically interacts with β-actin
- 52. Actin binds Saf-a/hnRNP U in vivo 0.5
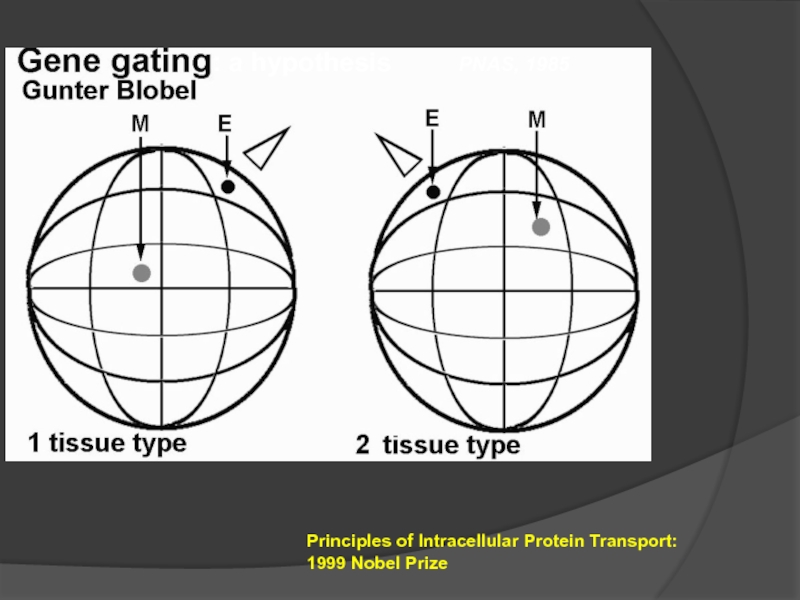
- 53. : a hypothesis
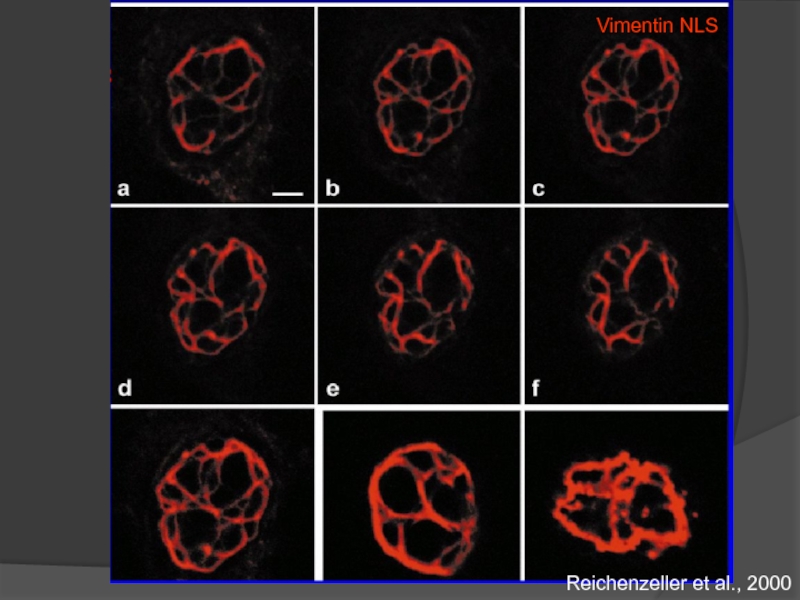
- 54. Vimentin NLS Reichenzeller et al., 2000
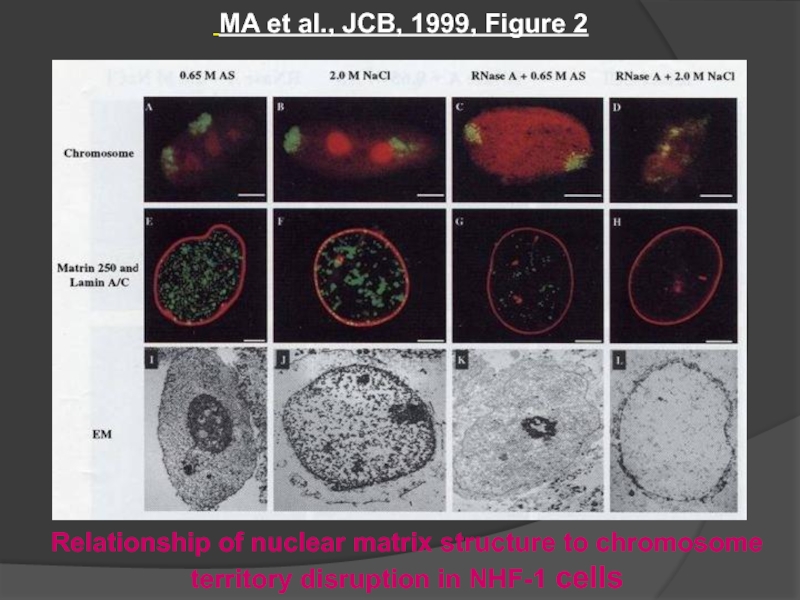
- 56. MA et al., JCB, 1999, Figure
- 57. MA et al., JCB, 1999, Figure
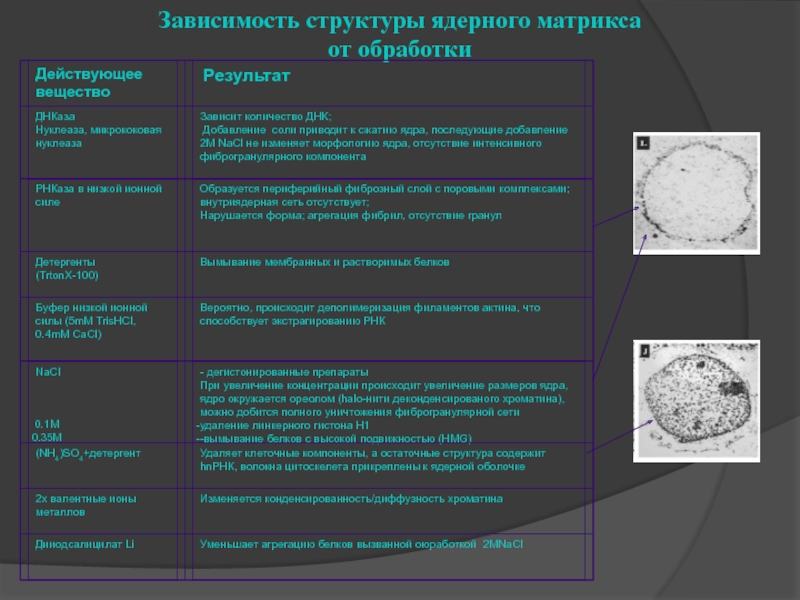
- 58. MA et al., JCB, 1999, Figure
- 59. Зависимость структуры ядерного матрикса от обработки 0.1M 0.35M
- 60. MAJOR CONCLUSIONS OF MA et al., 1999
- 61. Hendzel et al (1999) – в интерхроматиновом
- 62. Однако, существование одного из компонентов ядерного
- 63. Структурные белки цитоплазмы Поддержание формы клетки Локомоция
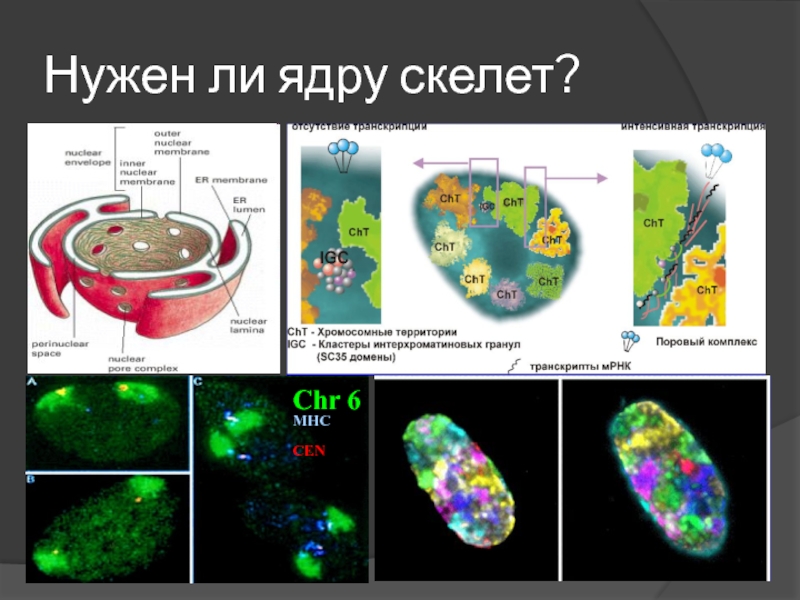
- 64. Какие функции нужны в ядре? Поддержание формы
- 65. Нужен ли ядру скелет? Chr 6 MHC CEN
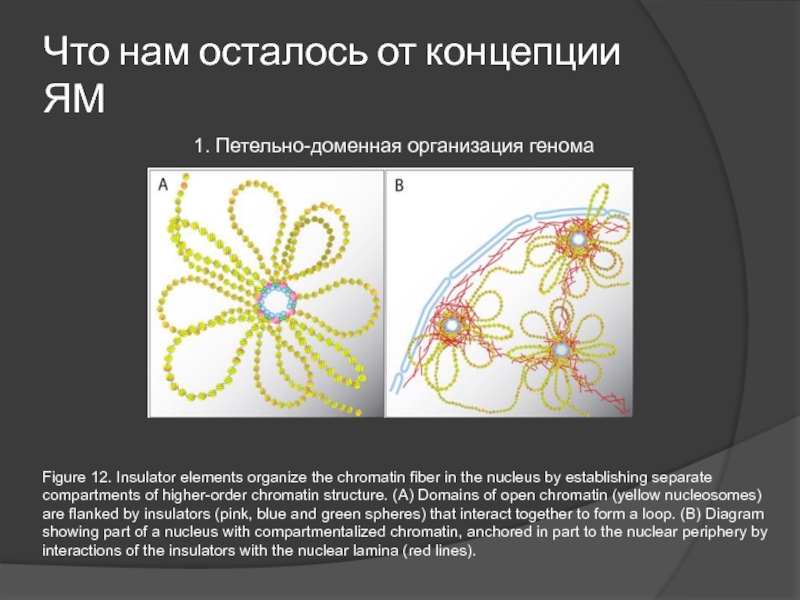
- 66. Что нам осталось от концепции ЯМ 1.
- 67. Schumaker et al., 2003 Что нам
- 68. Dahl K N , and Kalinowski
- 69. ЯМ – как сеть опорных промежуточных
- 70. Схема организации высших уровней упаковки хроматина 40%
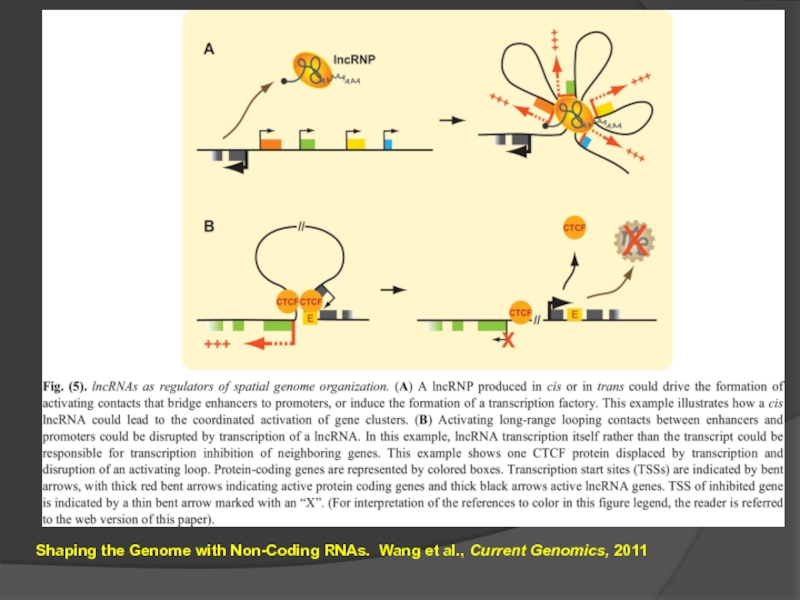
- 71. Петельные домены несомненно есть Основные белки,
- 72. Shaping the Genome with Non-Coding RNAs. Wang et al., Current Genomics, 2011
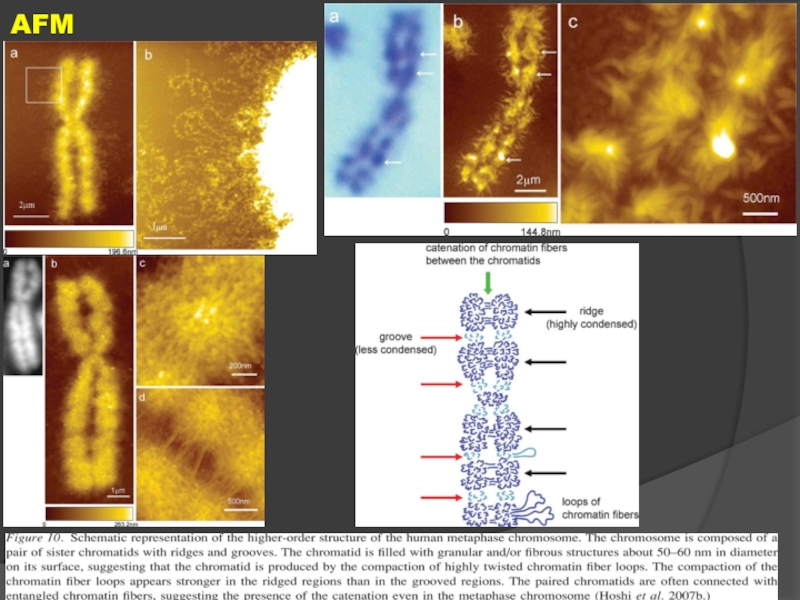
- 73. Модели высших уровней упаковки хроматина
- 74. AFM
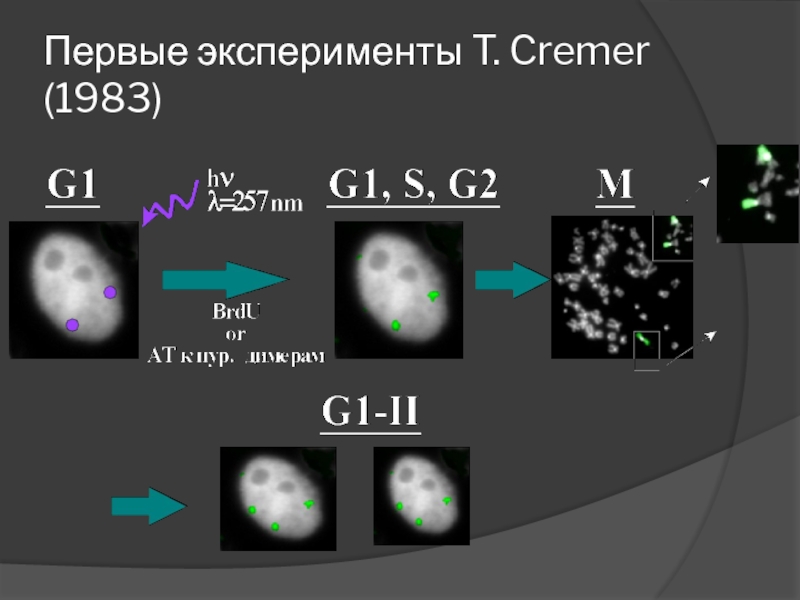
- 75. Хромосомные территории “Вермишель на тарелке” – хроматиновые
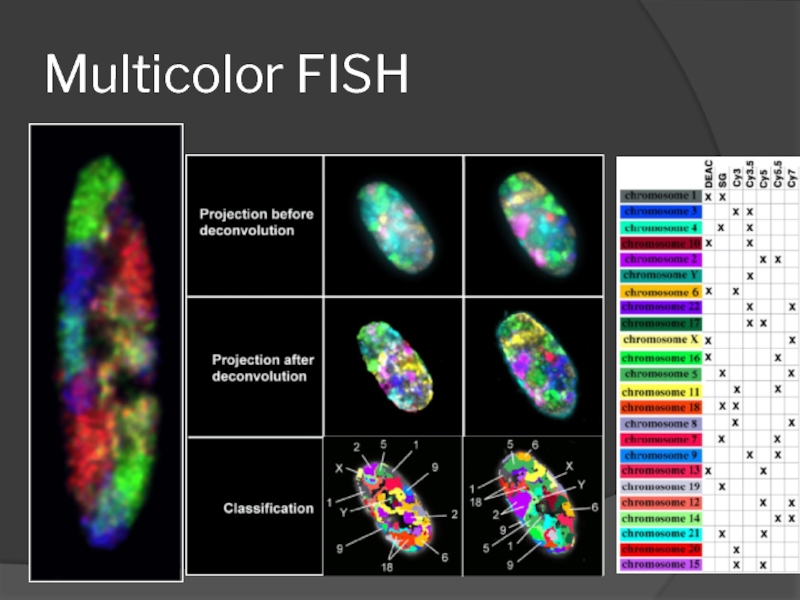
- 76. Первые эксперименты T. Cremer (1983)
- 77. Multicolor FISH
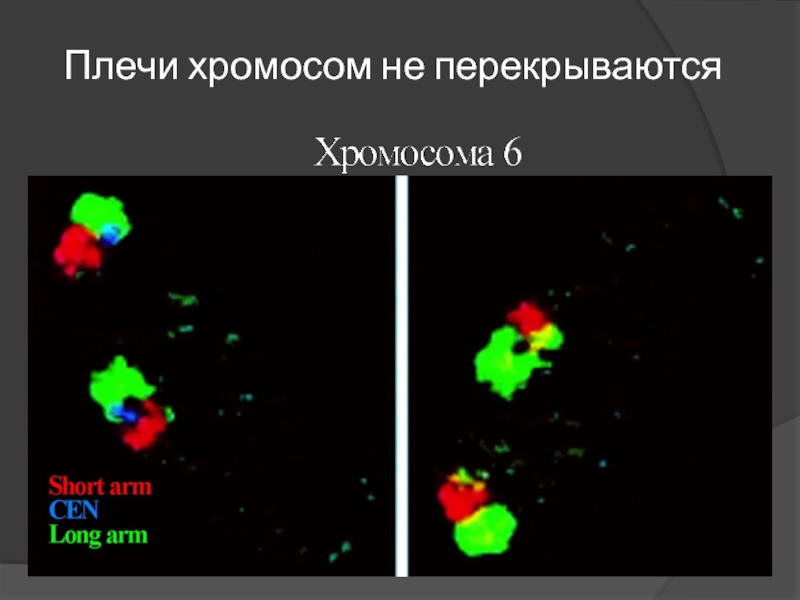
- 78. Плечи хромосом не перекрываются
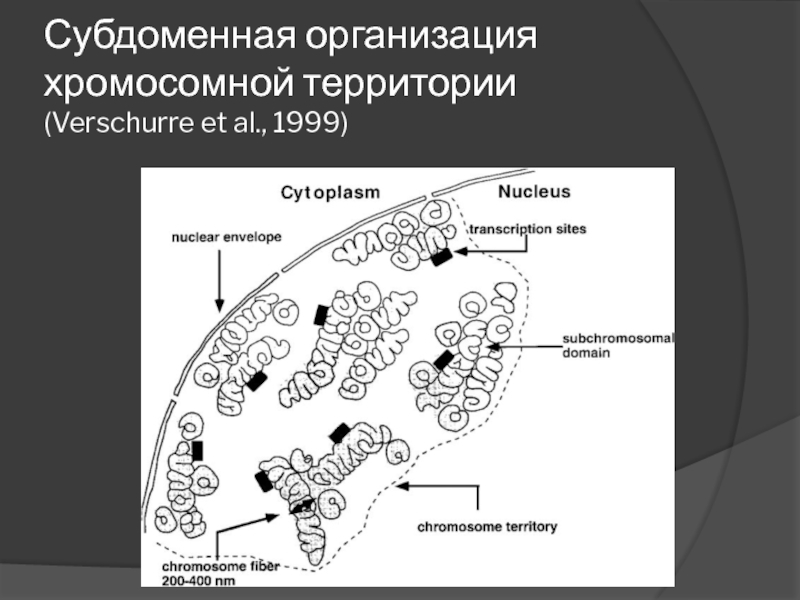
- 80. Субдоменная организация хромосомной территории (Verschurre et al., 1999)
- 81. Активные гены могут быть удалены от основной
- 82. Сателлитная ДНК также может проникать в соседник ХрТ
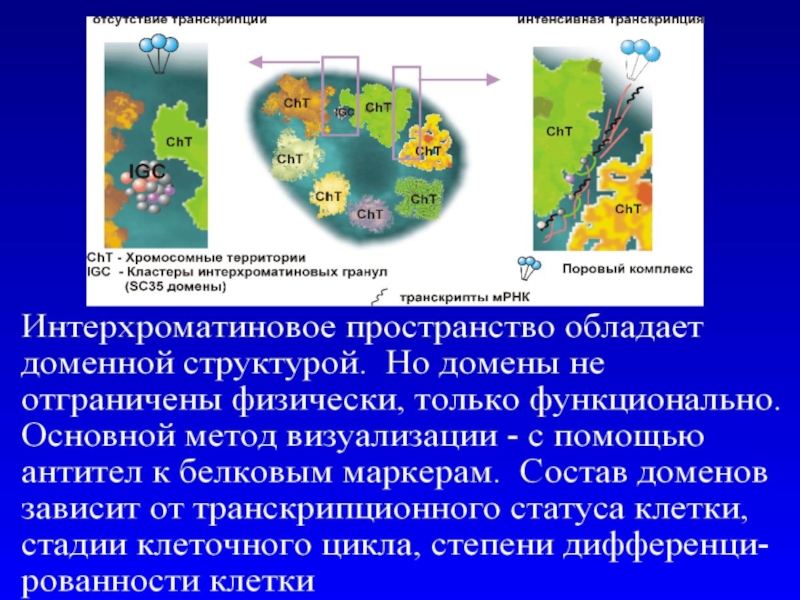
- 84. Интерхроматиновое пространство
- 85. Есть ли упорядоченность в 3-d организации интерфазного хроматина?
- 86. Distribution of large and small chromosomes in interphase nuclei of human diploid fibroblasts
- 87. Distribution of large and small chromosomes in spherical nuclei of human lymphocytes
- 88. There is a reproducible size-correlated pattern
- 89. How mobile are the chromosome territories
- 90. 1st cycle 3d cycle after at least
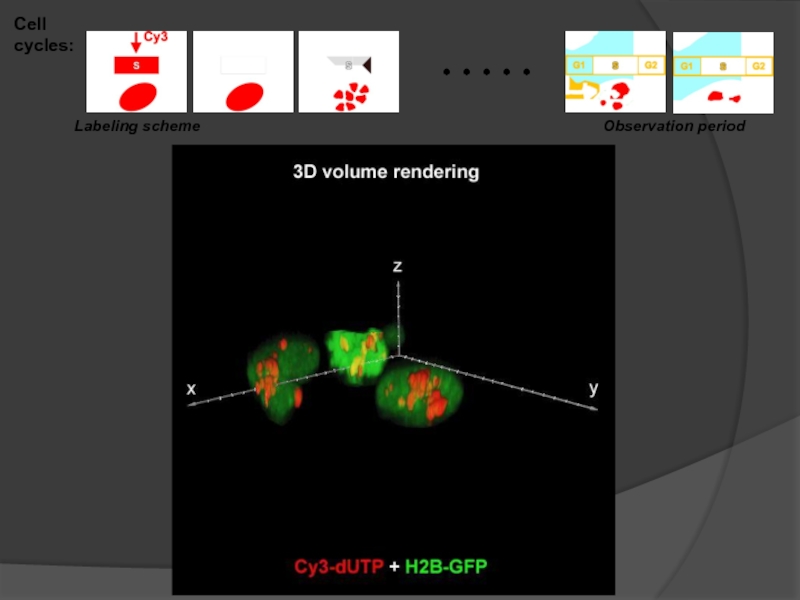
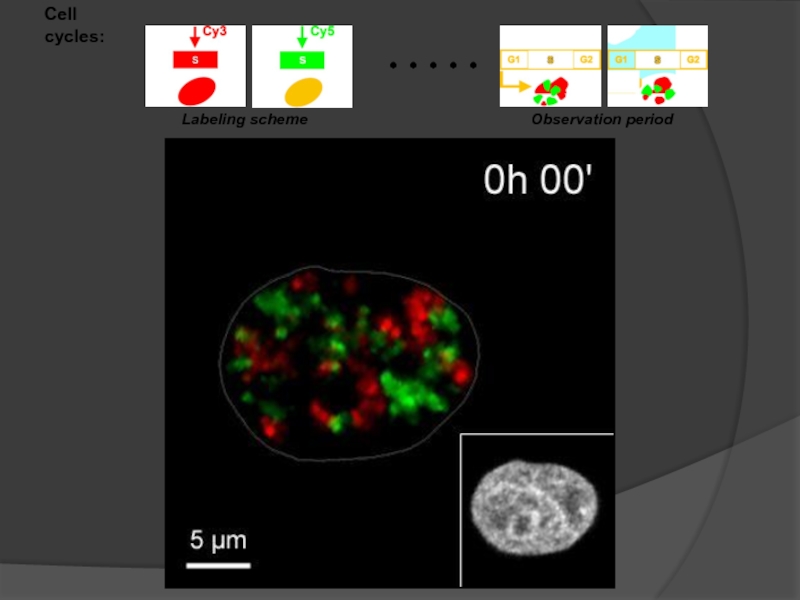
- 91. Cell cycles: . . . . . Labeling scheme Observation period
- 92. Chromosome territories move little, if
- 93. What determines the size-correlated distribution
- 94. prometaphase rosette of human fibroblast with labelled
- 95. We suggest that the difference in the
- 96. One more important aspect of order
- 97. Distribution of gene-poor chromosomes in spherical
- 98. Gene content affects chromosome distribution in spherical
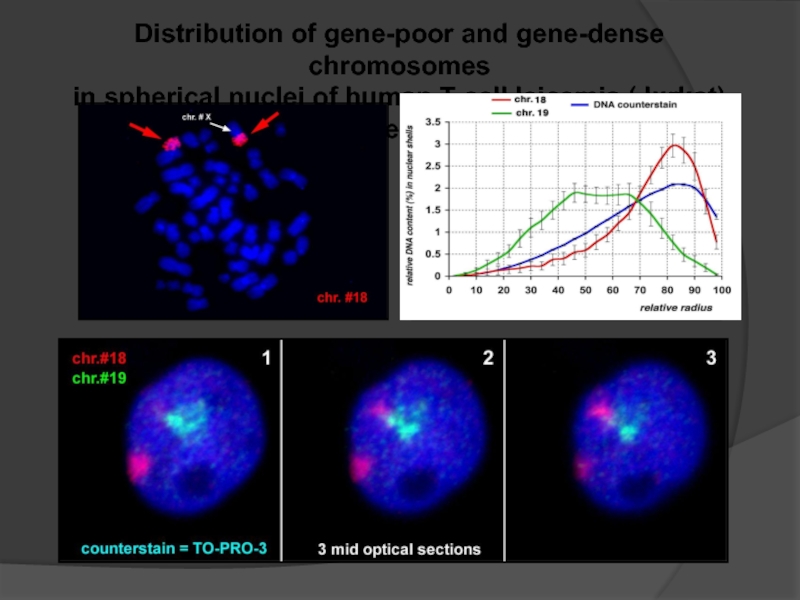
- 99. Distribution of gene-poor and gene-dense chromosomes
- 100. The distribution of chromatin homologous to human
- 101. Gene density affects the distribution of interphase
- 102. The differential positioning of gene-poor and
- 103. Nuclei are characterized by a
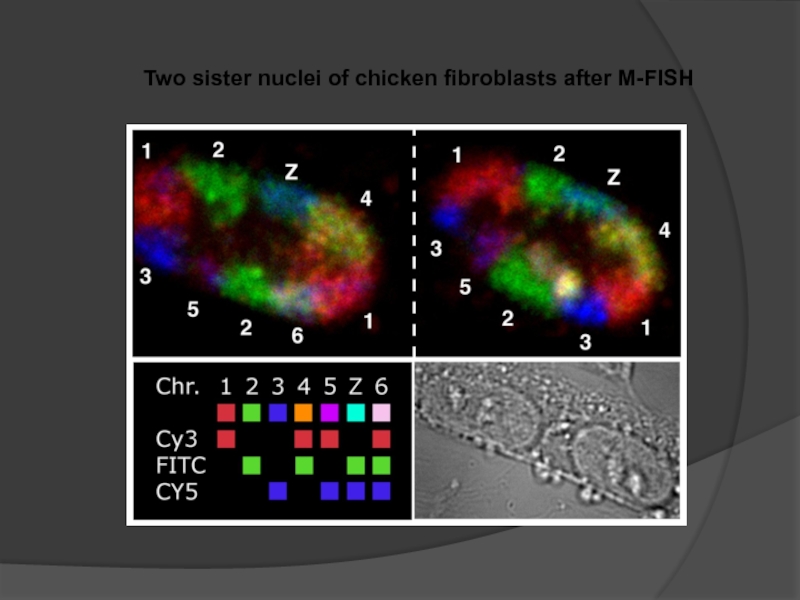
- 104. Seven chromosome territories (1- 6 and Z)
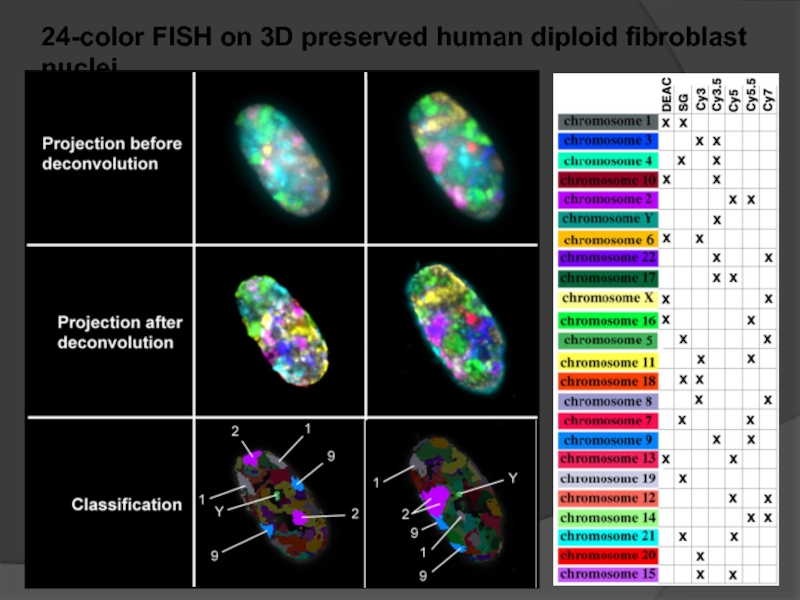
- 105. 24-color FISH on 3D preserved human diploid fibroblast nuclei
- 106. When and how do chromosomes change their
- 107. Cell cycles: Labeling scheme Observation period . . . . .
- 108. When and how chromosomes change their neigborhood
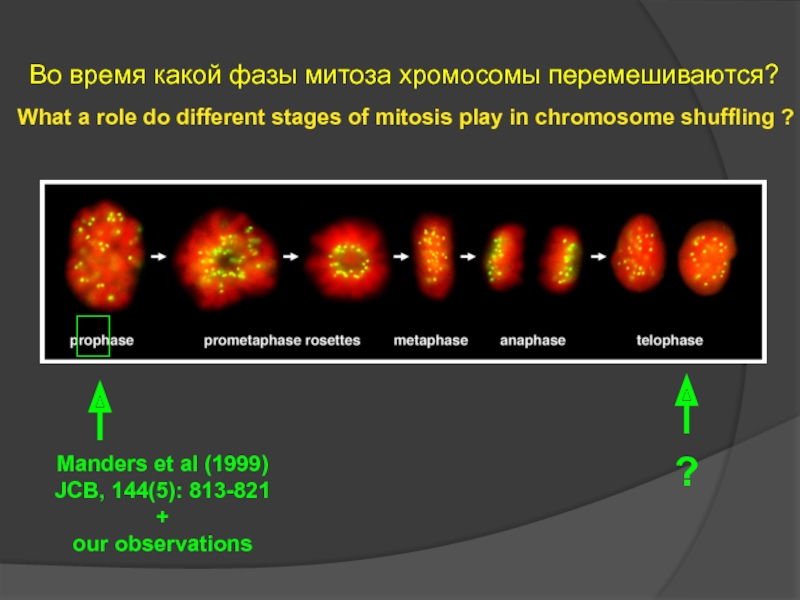
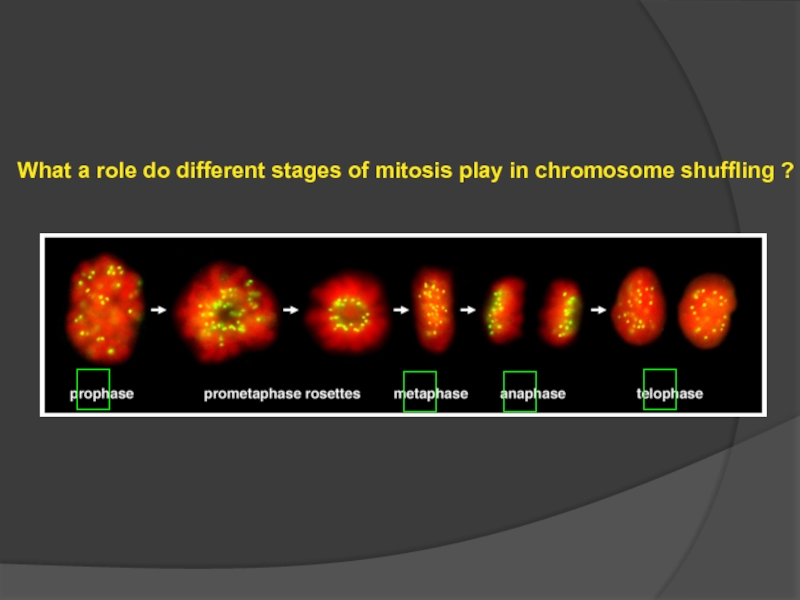
- 109. What a role do different stages
- 110. Two sister nuclei of chicken fibroblasts after M-FISH
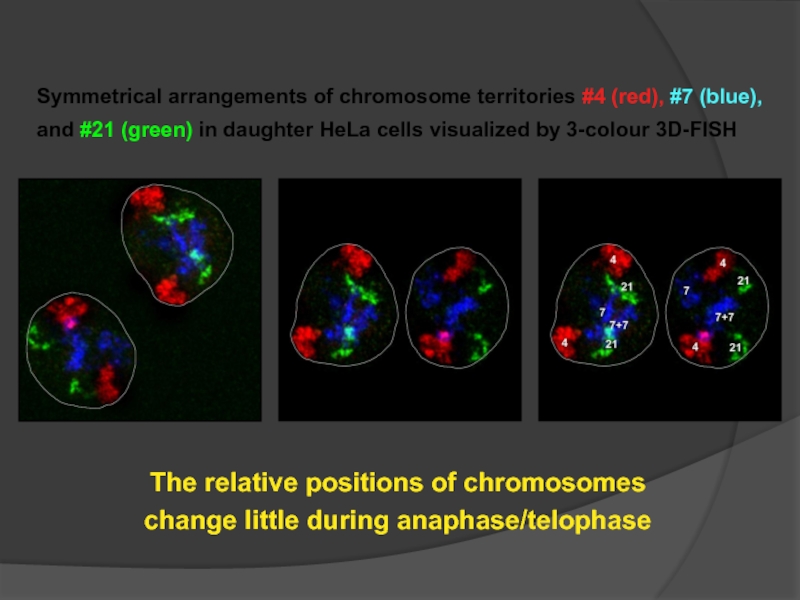
- 111. Symmetrical arrangements of chromosome territories #4 (red),
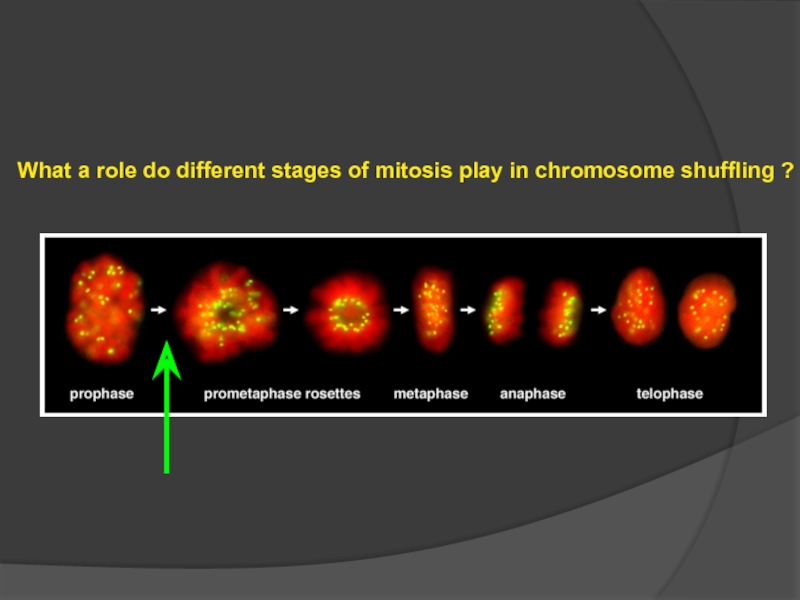
- 112. What a role do different stages
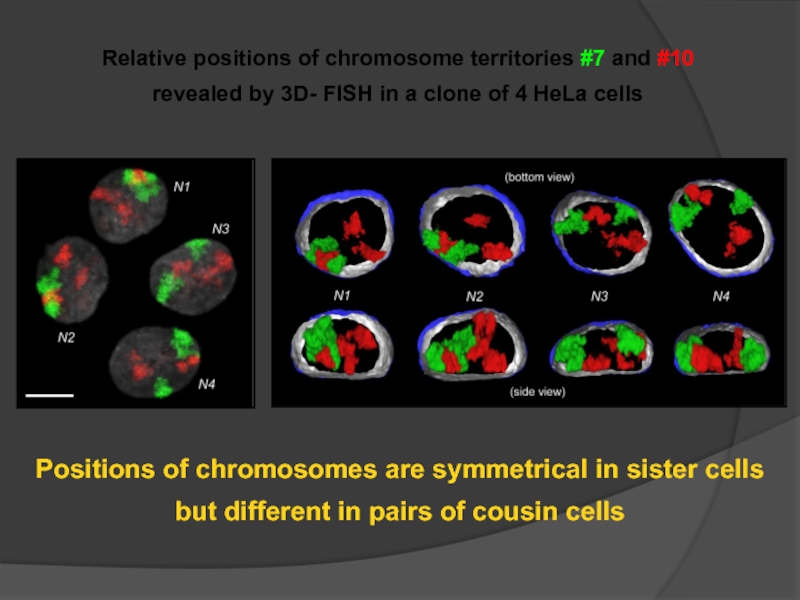
- 113. Relative positions of chromosome territories #7 and
- 114. What a role do different stages of mitosis play in chromosome shuffling ?
- 115. Shuffling of chromosomes takes place
- 116. Conclusions The arrangement of
- 117. University of Munich Department of Biology
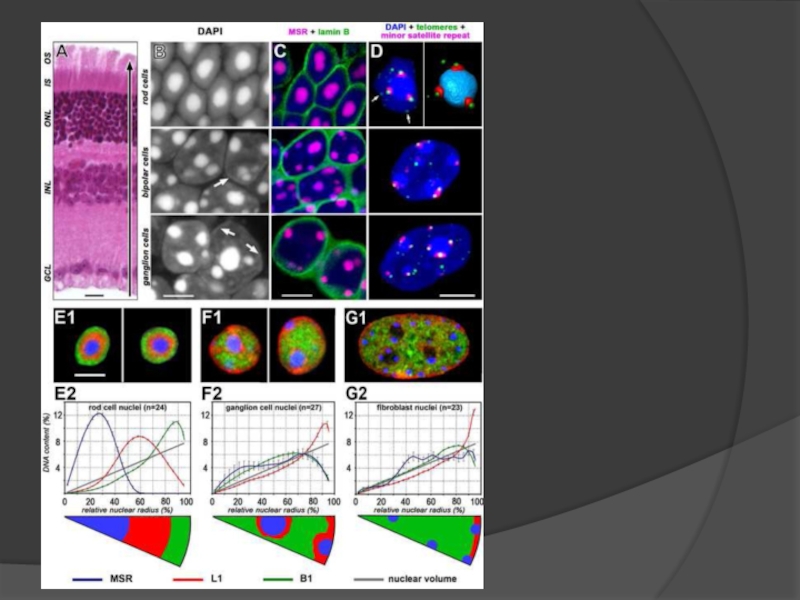
- 119. Cell. 2009 137(2):356-68. Nuclear architecture of rod
Слайд 1ХРОМАТИН. ГЕТЕРОХРОМАТИН. УПАКОВКА ГЕНОМА.
Наука это род человеческой деятельности
по поиску порядка
Слайд 5Уровни упаковки генома
Двойная спираль
10 нм фибрилла
30 нм фибрилла
Петельные домены
Фрагмент
конденсированной
хромосомы
Метафазная
хромосома
2 нм 1х
10
30 нм фибрилла 40х
Петельные домены 700х
Интерфаза
?
Из статьи:
Controlling the double helix
Gary Felsenfeld and Mark Groudine
Nature 421, 448-453
Слайд 7Нуклеонема (10 нм)
146 пн – нуклеосома, 166 п.н. – нуклеосома+Н1, 200
Слайд 8Фазирование нуклеосом
Фиксированное расположение нуклеосом
Вирус MMTV,
Участок начала репликации SV40,
βmayor-глобиновый
альфоидная ДНК АЗМ
Возникает
при наличии в определенном районе генома барьера для распространения нуклеосом (напр. места связывания белковых факторов)
При наличии последовательности, на которой нуклеосома располагается с большим предпочтением (напр. Изогнутые, на которых фазированно расположены каждые 10 пар блоки из 2-3 АТ)
Слайд 9Гистоны
Основные типы H1 (H5 у птиц), H2A, H2B, H3, H4
Обогащены
Сходная вторичная структура
Слайд 10Вариантные формы гистонов
H1, H2A, H2B, H3
Кодируются отдельным однокопийным геном (кроме H3.3)
CENP-A
H3.3 – вариант транкрипционно активного хроматина
H3.lt – вариант специфичный для семенников
hTSH2b, H2BFWT – формы H2B. Известны только в сперматозоидах
macroH2A – гистон неактивной X-хромосомы. 57% негомологично ни одному из гистонов
H2ABbd – компонент активного хроматина
H2A.Z – компонент активного хроматина
H2A.X – в местах репарации ДНК
Слайд 12«Гистоновый код»
Совокупность сигналов, экспонированных на поверхности нуклеосом (включая как присутсвие вариантов
Слайд 13Гистоновый код
Genome-Wide Views of Chromatin Structure
Oliver J. Rando and Howard Y.
2009
Слайд 16Роль H1 и хвостов октамерных гистонов в образовании 30-нм фибриллы
Изменения структуры
Слайд 17АТФ-зависимые комплексы ремоделирования хроматина
Перемещение нуклеосом вдоль молекулы ДНК и размещение нуклеосом
Частичная декомпактизация нуклеосом, сопряженная с изменением спектра контактов ДНК и гистонов
Перемещение нуклеосомных глобул с одной молекулы ДНК на другую
Замена канонических гистонов на вариантные
Решают проблемы, связанные с нуклеосомной организацией
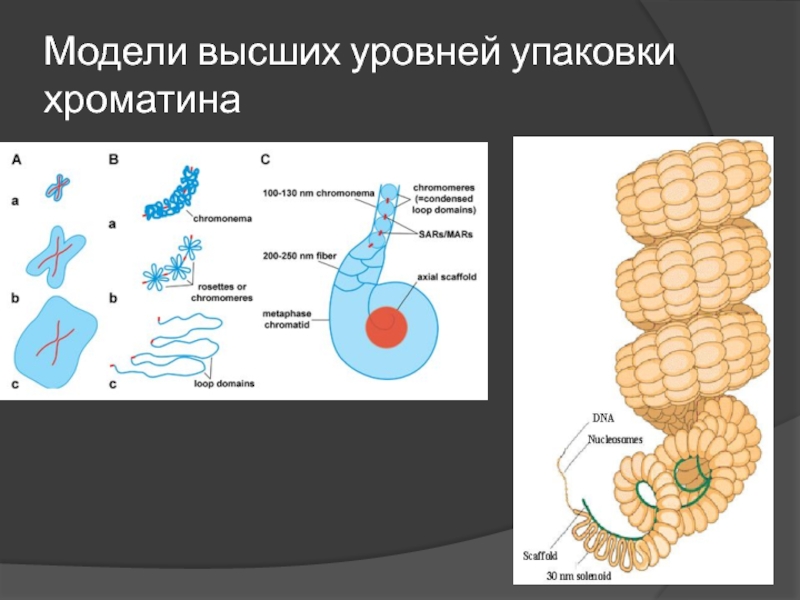
Слайд 19Высшие уровни упаковки хроматина
Соленоид?
Латеральное взаимодействие соседних фибрилл?
Радиально-петлевая модель ?
Слайд 20Радиально-петлевая модель
Двойная спираль
10 нм фибрилла
30 нм фибрилла
Петельные домены
Фрагмент
конденсированной
хромосомы
Метафазная
хромосома
2 нм 1х
10 нм
30 нм фибрилла 40х
Петельные домены 700х
Интерфаза
?
Слайд 21Доменная гипотеза организации эукариотического генома
Весь геном построен из однотипных блоков -
могут включать один или несколько генов
Домены в целом находятся под контролем особых регуляторных
систем, которые контролируют их транскрипционный статус на уровне
упаковки (более компактной или менее компактной) всего домена в
интерфазной хромосоме
Активный домен
Неактивный домен
Слайд 241947. Mirsky, Ris. Фракция остаточных белков при солевой экстракции хромосом
1948. Zbarsky,
1962. Zbarsky et al, 1962; Georgiev and Chentsov, 1962 - препараты ядер, обработанные сходным образом, сохраняют очертания ядра и в них выявляются четко различимые остаточное ядрышко и участки гетерохроматина
1974. . Berezney and Coffey опубликовали статью о биохимическом получении фракции остаточных белков и предложили термин “ядерный матрикс”.
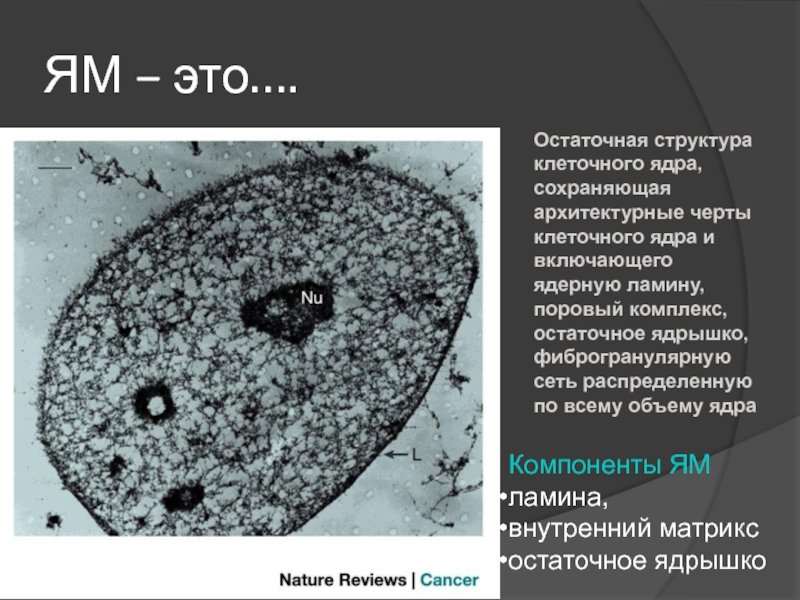
Слайд 25ЯМ – это….
Компоненты ЯМ
ламина,
внутренний матрикс
остаточное ядрышко
Остаточная структура клеточного ядра,
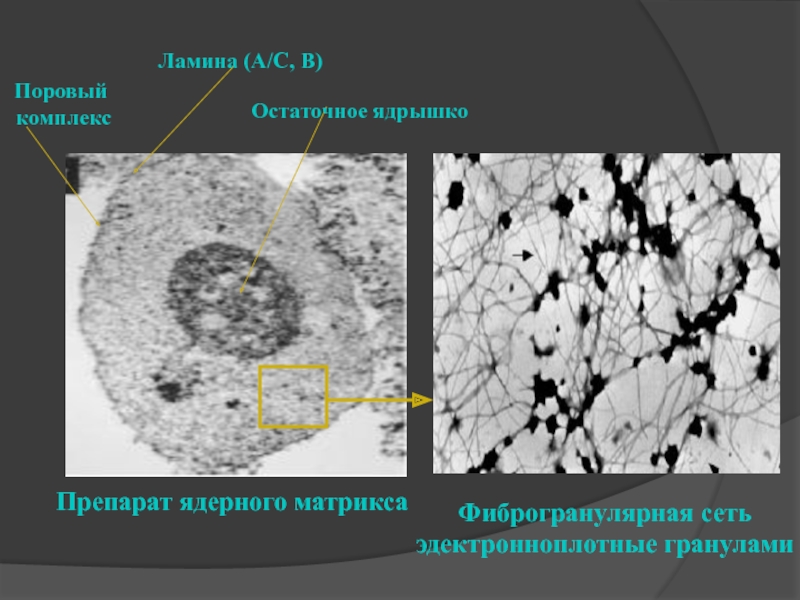
Слайд 26
Препарат ядерного матрикса
Фиброгранулярная сеть
эдектронноплотные гранулами
Остаточное ядрышко
Ламина (A/C, B)
Поровый
комплекс
Слайд 29Непрерывный клеточный скелет
Figure 1 Transmission electron micrograph (X47,000) of a
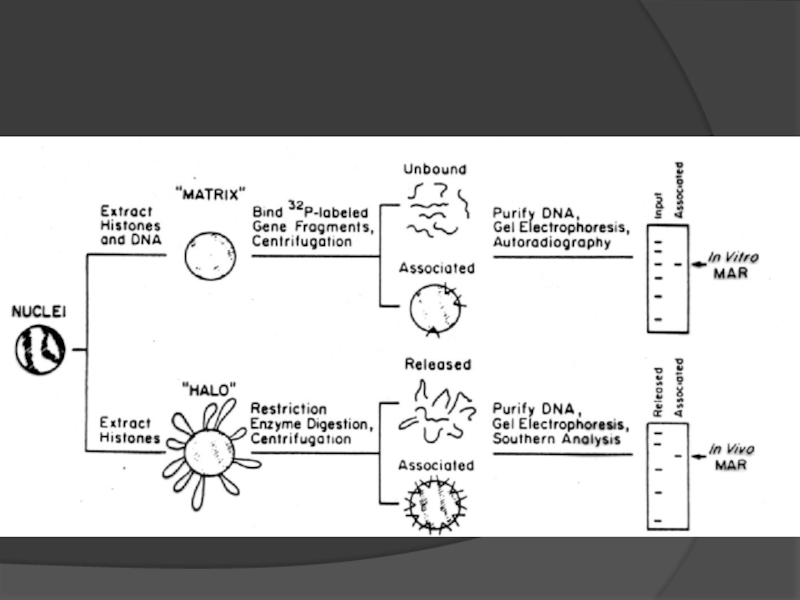
Слайд 32Regulating the mammalian genome: the role of
nuclear architecture
Ronald Berezney (2002)
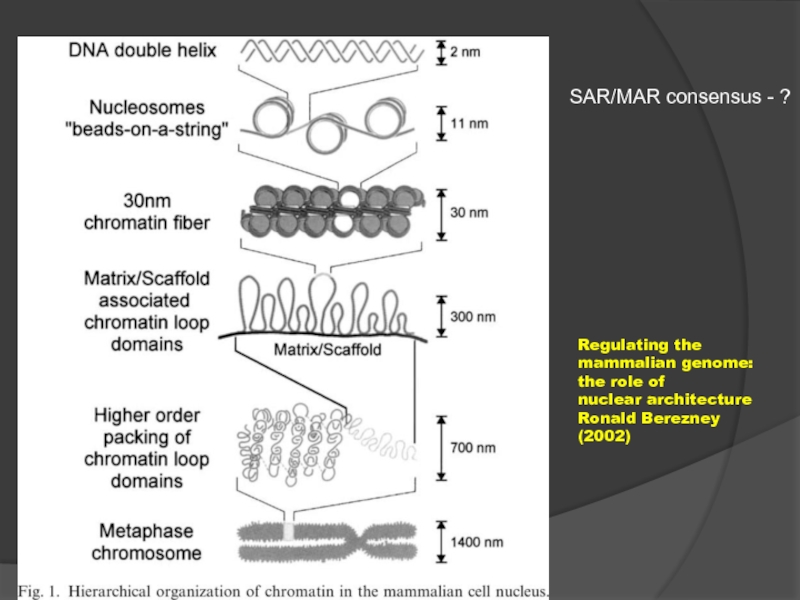
SAR/MAR consensus
Слайд 33
Alexey Gavrilov, Sergey V. Razin, and Olga V. Iarovaia
Department of Structural
Institute of Gene Biology
Moscow
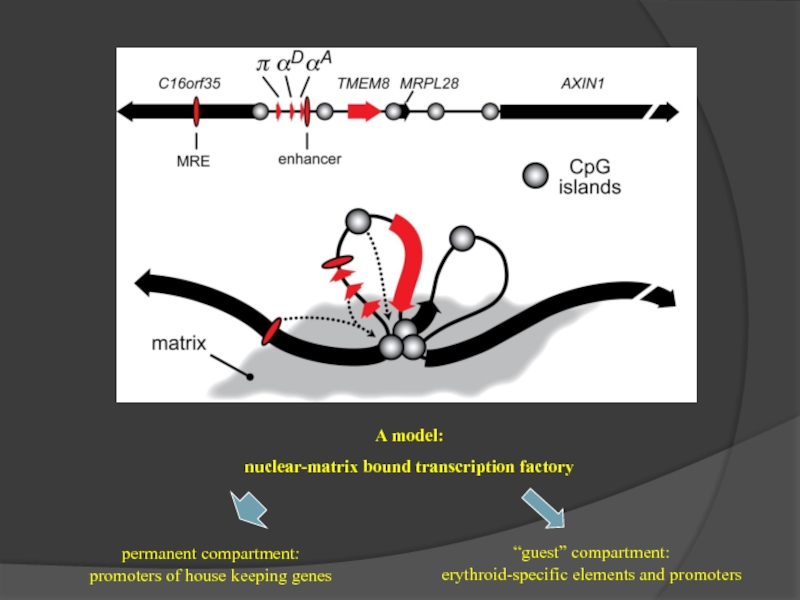
Mapping of the nuclear matrix-bound chromatin hubs
by a new M3C experimental procedure
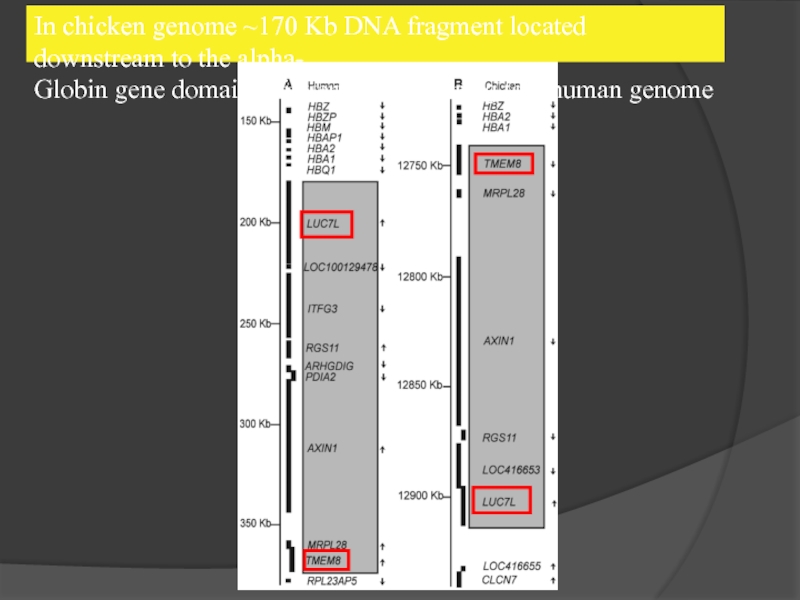
Слайд 37A model to test M3C approach –
a fragment of chicken chromosome
Слайд 38
In chicken genome ~170 Kb DNA fragment located downstream to the
Globin gene domain is inverted, comparing to a human genome
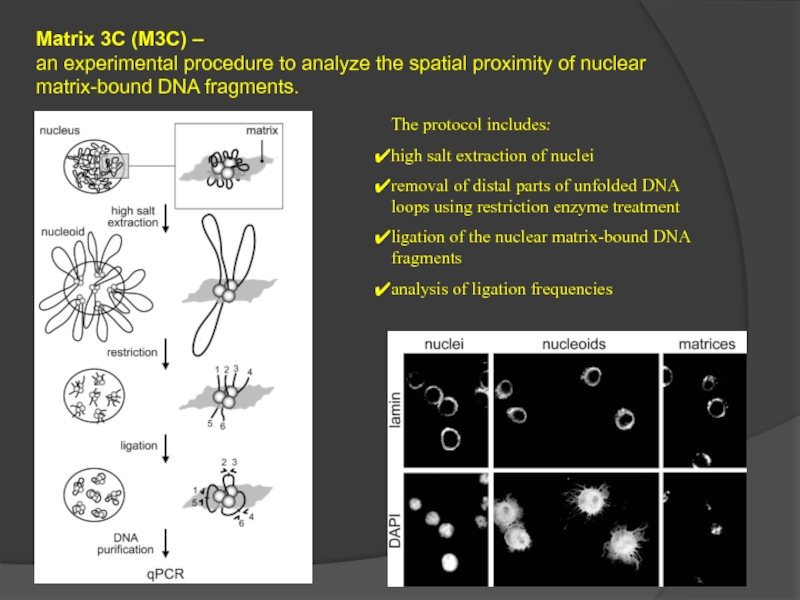
Слайд 39Matrix 3C (M3C) –
an experimental procedure to analyze the spatial proximity
The protocol includes:
high salt extraction of nuclei
removal of distal parts of unfolded DNA loops using restriction enzyme treatment
ligation of the nuclear matrix-bound DNA fragments
analysis of ligation frequencies
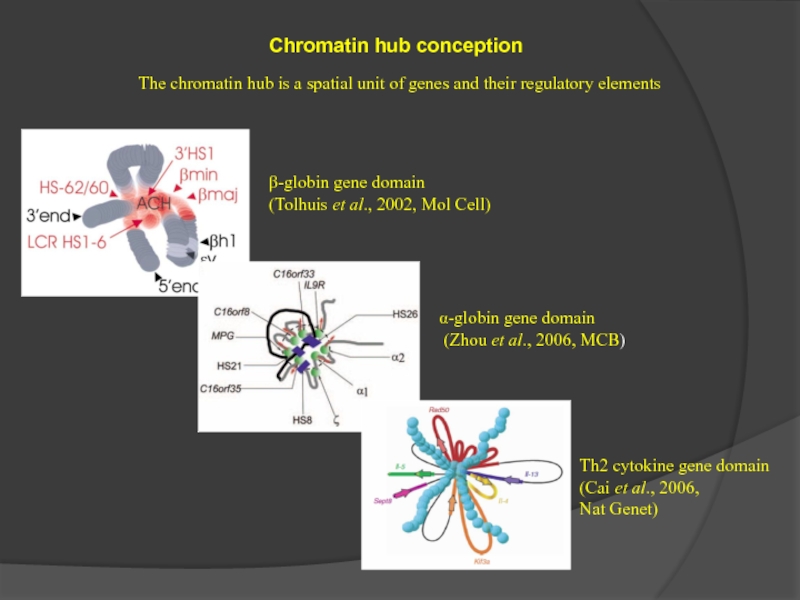
Слайд 41Chromatin hub conception
The chromatin hub is a spatial unit of
β-globin gene domain
(Tolhuis et al., 2002, Mol Cell)
α-globin gene domain
(Zhou et al., 2006, MCB)
Th2 cytokine gene domain
(Cai et al., 2006,
Nat Genet)
Слайд 45Nuclear
matrins
:
Identification of the major
nuclear matrix proteins
HIROSHI NAKAYASU*
AND
BEREZNEYt
PNAS, 1991
Matrin
3 Binds and Stabilizes
mRNA
Salton et al.,
2011
Белки ЯМ (Nakayasu, Berezney, 1991)
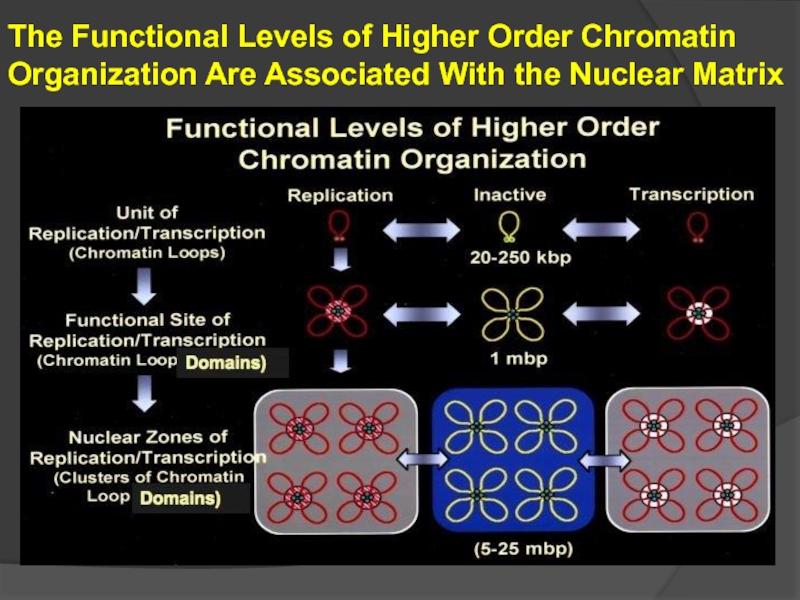
Слайд 46The Functional Levels of Higher Order Chromatin
Organization Are Associated With
Слайд 471. The nuclear matrix is composed of a major group of
2. Many of the nuclear matrins are pre-mRNP, RNA splicing or transcriptional factors but many have yet to be identified.
3. Matrin 3 is an ~ 96 kDa protein that contains 2 Zn finger motifs, RNA Recognition Motifs (RRM’s) and an acidic rich domain at the C-T common among transcriptional activators (J Biol Chem 266, 1991: 9893)
Nuclear Matrix Proteins
Слайд 484. Matrin Cyp (cyclophilin) a ~88 kDa protein that contains the
5. Matrin SRm 160 (~160 kDa protein) is an exon junction splicing factor (Mol. Cell. Biol. 22, 2002: 148).
6. Matrin 250 (~250 kDa) is the hyperphosphorylated form of RNA pol II LS (PNAS 93, 1996: 8253). 7. Matrin SCAF 8 (140 kDa) contains SR-rich motifs and a binding domain specific for hyperphosphorylated CTD of RNA pol II LS (Mol. Cell Biol. 18, 1998, 2406)
Nuclear Matrix Proteins (cont.)
Слайд 49Nuclear Matrix Proteins (cont.)
DNA (Chromatin) Loop Anchoring Proteins
or MAR/SAR Binding Proteins
DNA Topoisomerase
SAF-A and SAF-B (Bind both DNA and RNA)
SAT B1 – MAR protein specific for lymphocytes
What else ???? More Research is needed at the levels of DNA (chromatin loops), multi-loop chromatin domains ( ~1 mbp domains) and whole chromosome territories.
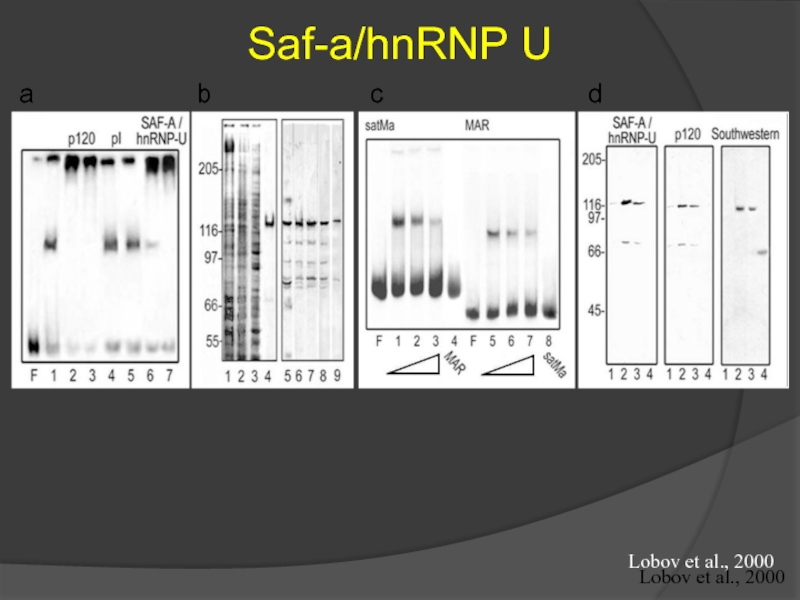
Слайд 51Saf-a/hnRNP U specifically interacts with
β-actin in the nucleus
Actin and hnRNP
Kukalev A et al., Nature, 2005
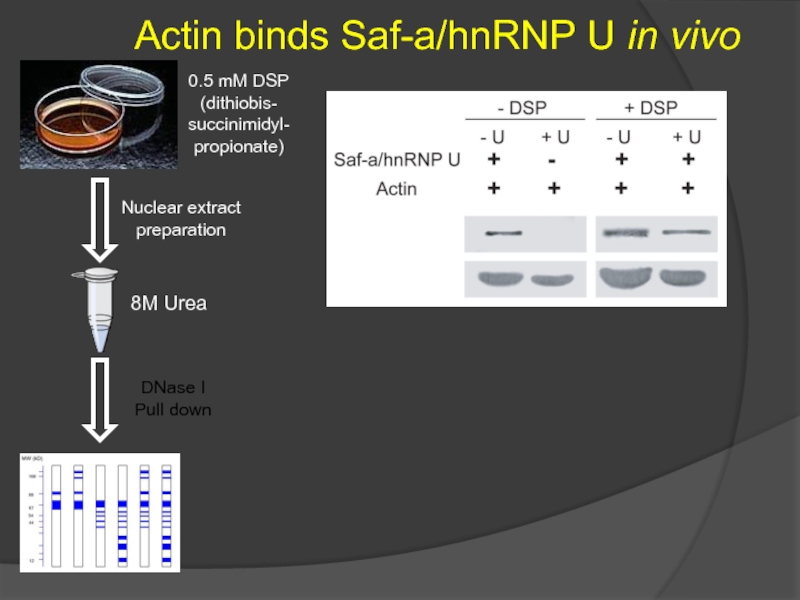
Слайд 52Actin binds Saf-a/hnRNP U in vivo
0.5 mM DSP
(dithiobis-
succinimidyl-
propionate)
Nuclear extract
preparation
8M Urea
DNase I
Pull
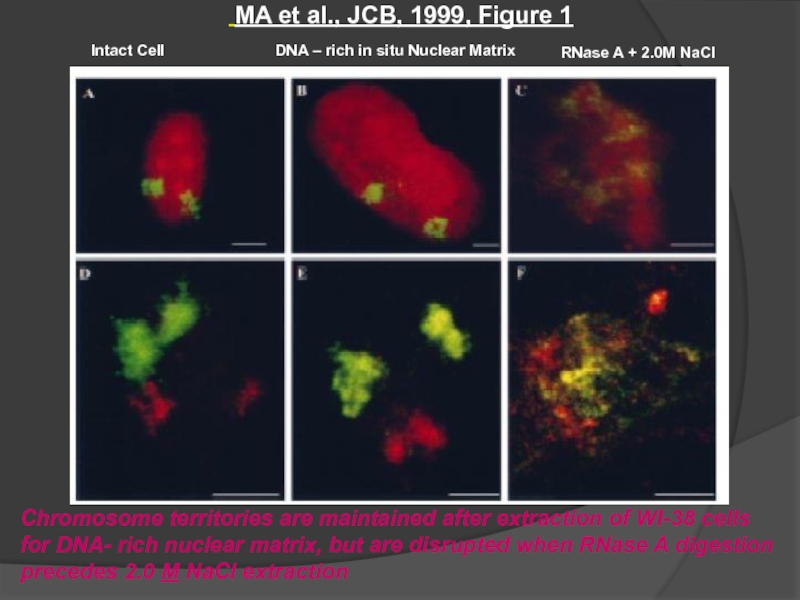
Слайд 56 MA et al., JCB, 1999, Figure 1
Chromosome territories are maintained
Intact Cell
DNA – rich in situ Nuclear Matrix
RNase A + 2.0M NaCl
Слайд 57 MA et al., JCB, 1999, Figure 2
Relationship of nuclear matrix
Слайд 58 MA et al., JCB, 1999, Figure 4
Protocol for releasing nuclear
Слайд 60MAJOR CONCLUSIONS OF MA et al., 1999
Chromosome territory organization is maintained
Disruption of nuclear matrix organization by pre-treatment with RNase A before 2M NaCl extraction leads to a corresponding disruption of territorial organization (Fig 1 & 2)
The finding that extraction with ammonium sulfate at similar ionic strength (0.65 M) as 2M NaCl following RNase does not lead to territorial disruption (Fig 2) has led to a procedure to isolate proteins that are released in association with disruption of territories (Fig 4)
These released proteins comprise a distinct subset of proteins in nuclear matrix preparations (Fig 5) and are termed CTAPs (Chromosome Territory Anchoring Proteins)
Слайд 61Hendzel et al (1999) – в интерхроматиновом пространстве не выявляется нуклеопротеиновая
Pederson (2000) – в ядре нет места для волокон ЯМ
Misteli etal (1997) - Стационарное положение интерхроматиновых доменов объясняется высокой концентрацией белков и нуклеопротеинов в интерхроматиновом пространстве.
Hancock (1999) - Анализ методик получения ЯМ показывает, что каждый из шагов получения ЯМ (инкубация при 37оС, обработка соединениями меди, дийодсалицилатом лития, NaCl или (NH4)2SO4 и т.п.) приводит к образованию артефактных белок-белковых комплексов. Филаментная сеть, наблюдавшаяся первыми исследователями, есть как результат артефактной агрегации.
Tan et al. (2000) - белки рибонуклеопротеиновых комплексов при инкубации их при 37оС и обработке их 2-меркаптоэтанолом и высокими концентрациями солей образуют филаменты 7-10 нм.
Слайд 62
Однако, существование одного из компонентов ядерного матрикса – промежуточнофиламентной ламины in
Известно, что во внутреннем пространстве ядра располагаются белки, способные к полимеризации в филаментноподобные структуры – ламины, ядерный актин, NuMa, белок порового комплекса Tpr (Moir et al., 1994; Parry, 1994; Hozak et al., 1995; Jagatheesan et al., 1999; Pedersen, 2000).
Показано, что эти белки не принимают участия в формировании внутриядерных филаментов in vivo (Parry, 1994; Moir et al., 1994; Hozak et al., 1995; Nickerson, 1995; Jagatheesan et al., 1999).
Однако обсуждается возможность формирования небольших локальных филаментных сетей (gene expression matrices) в местах активной транскрипции (Pedersen, 2000
Слайд 63Структурные белки цитоплазмы
Поддержание формы клетки
Локомоция
Участие в межклеточных контактах
Регуляторные функции
Опосредование вне-
Слайд 64Какие функции нужны в ядре?
Поддержание формы клетки
Локомоция
Участие в межклеточных контактах
Регуляторные функции
Опосредование вне- и внутриклеточных сигналов
Отделение ядерного пространства
Контроль за транспортом внутри ядра
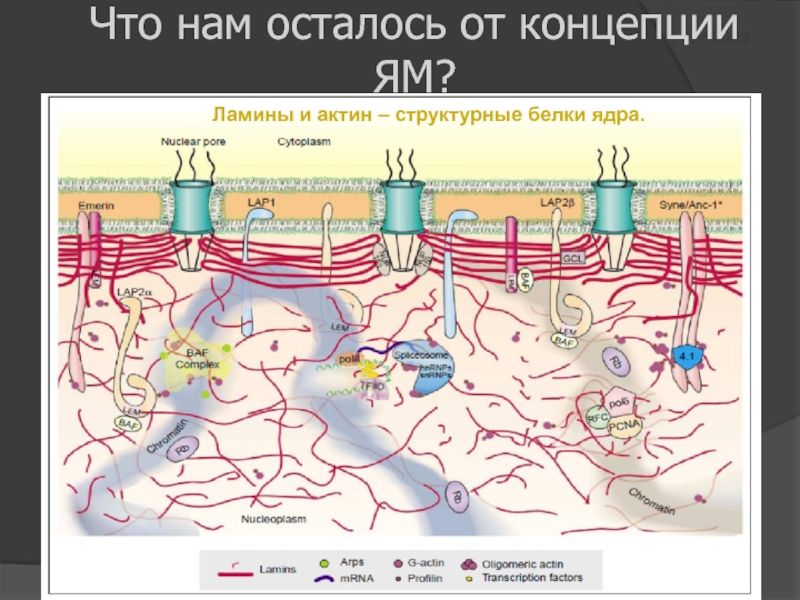
Слайд 66Что нам осталось от концепции ЯМ
1. Петельно-доменная организация генома
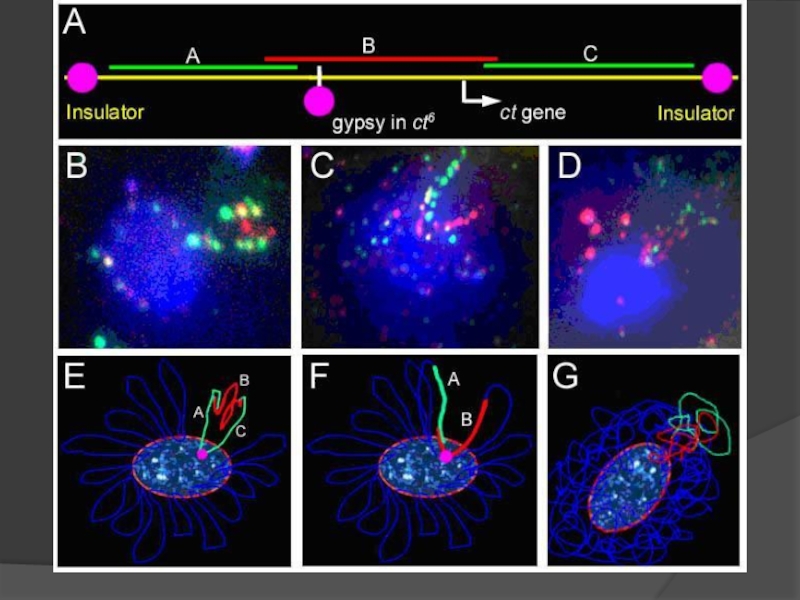
Figure 12. Insulator
Слайд 67
Schumaker et al., 2003
Что нам осталось от концепции ЯМ?
Ламины и актин
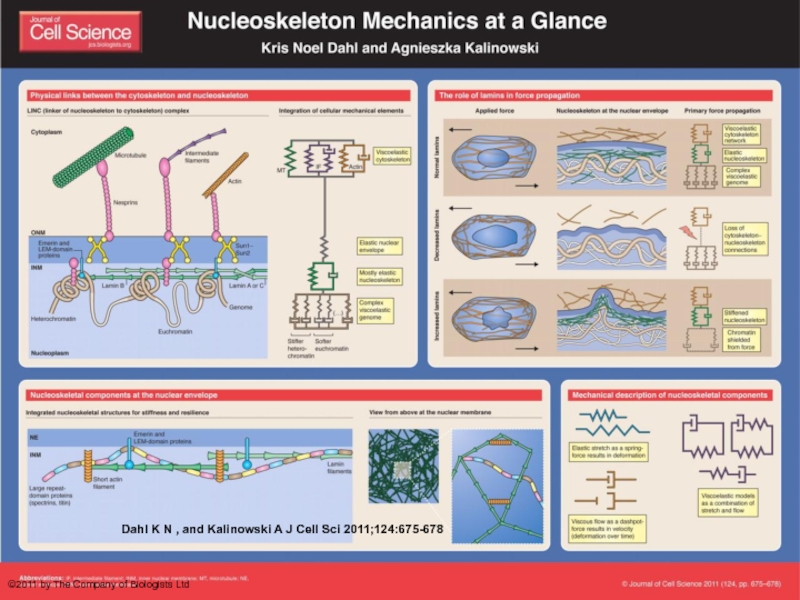
Слайд 68
Dahl K N , and Kalinowski A J Cell Sci 2011;124:675-678
©2011
Слайд 69
ЯМ – как сеть опорных промежуточных филаментов – неверная гипотеза
НО его
Мы также получили удобный метод для изучения белков, взаимодействующих с определенной фракцией генома – некодирующей ДНК, активно транскрибируемой ДНК и реплицирующейся ДНК
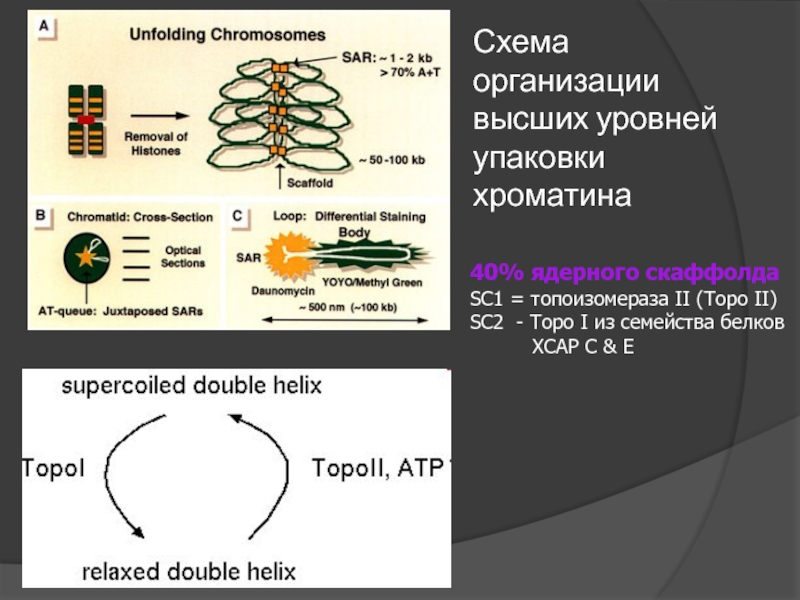
Слайд 70Схема организации высших уровней упаковки хроматина
40% ядерного скаффолда
SC1 = топоизомераза II
SC2 - Topo I из семейства белков
XCAP C & E
Слайд 71
Петельные домены несомненно есть
Основные белки, участвующие в формировании петель – Топоизомеразы,
Некоторые из них ассоциированы с локальными скелетными (напр. актиновыми) филаментами
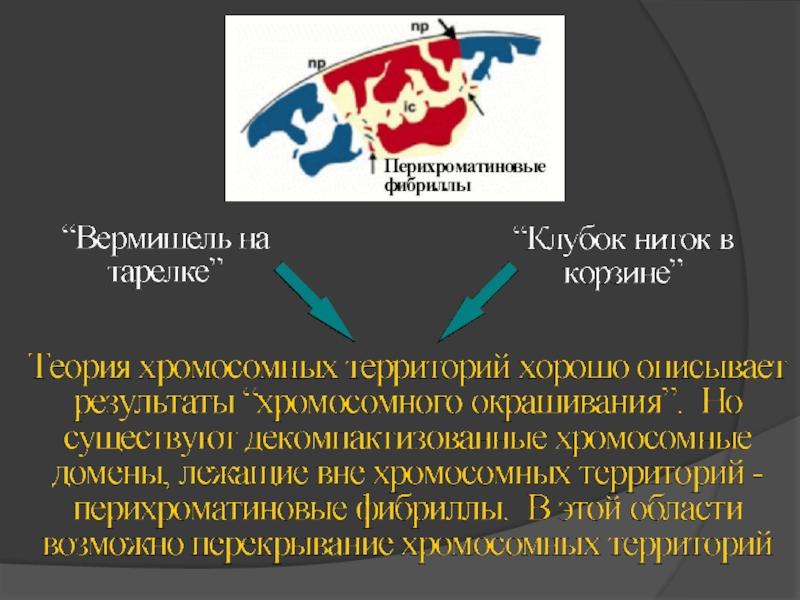
Слайд 75Хромосомные территории
“Вермишель на тарелке” – хроматиновые нити, беспорядочно распределенные по ядру
“Клубки
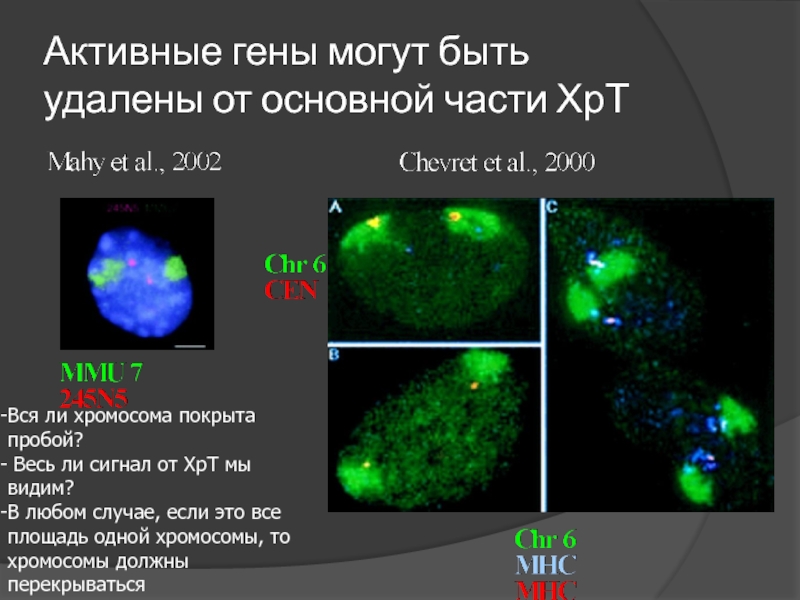
Слайд 81Активные гены могут быть удалены от основной части ХрТ
Вся ли хромосома
Весь ли сигнал от ХрТ мы видим?
В любом случае, если это все площадь одной хромосомы, то хромосомы должны перекрываться
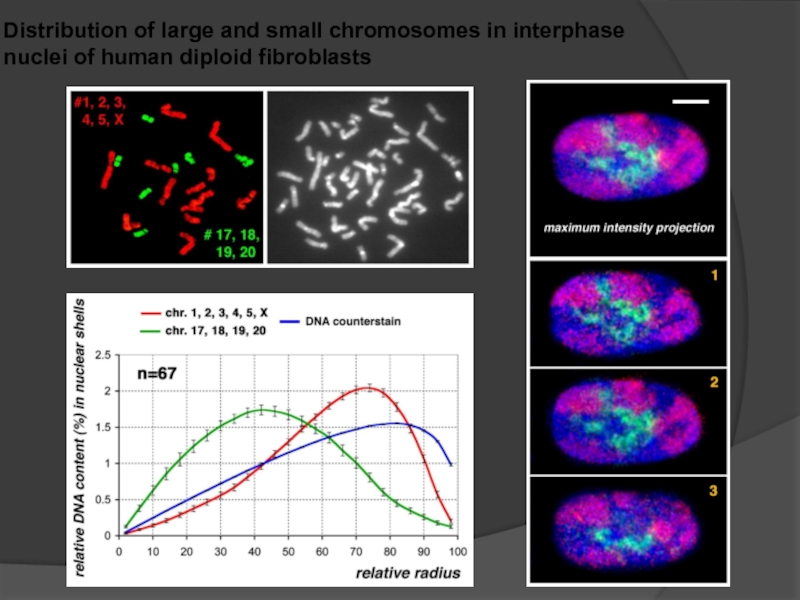
Слайд 86Distribution of large and small chromosomes in interphase nuclei of human
Слайд 88There is a reproducible size-correlated pattern
of radial chromosome arrangement:
towards the periphery of the nucleus
small chromosomes have
a central position
Большие хромосомы тяготеют к периферии
ядра
Маленькие хромосомы тяготеют к центру
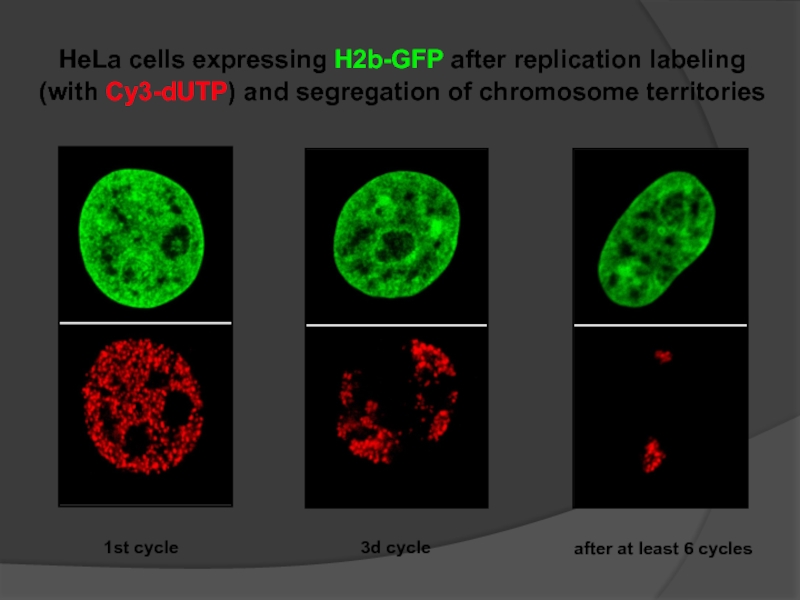
Слайд 89How mobile are the chromosome territories
in the interphase nucleus ?
What determines
distribution of chromosomes ?
Какой механизм расставляет хромосомы по размеру?
Насколько подвижны хромосомные
территории в интерфазе
Слайд 901st cycle
3d cycle
after at least 6 cycles
HeLa cells expressing H2b-GFP after
(with Cy3-dUTP) and segregation of chromosome territories
Слайд 92Chromosome territories move little,
if at all, during interphase
СТ почти не
Слайд 93What determines the size-correlated
distribution of chromosomes ?
We suggest that
in the positioning of chromosomes in the mitotic cells.
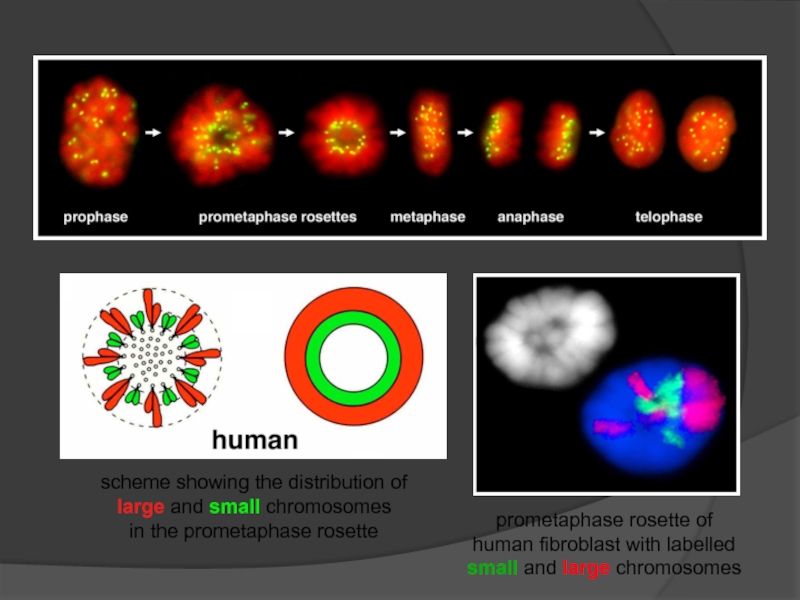
Слайд 94prometaphase rosette of
human fibroblast with labelled
small and large chromosomes
scheme showing the
large and small chromosomes
in the prometaphase rosette
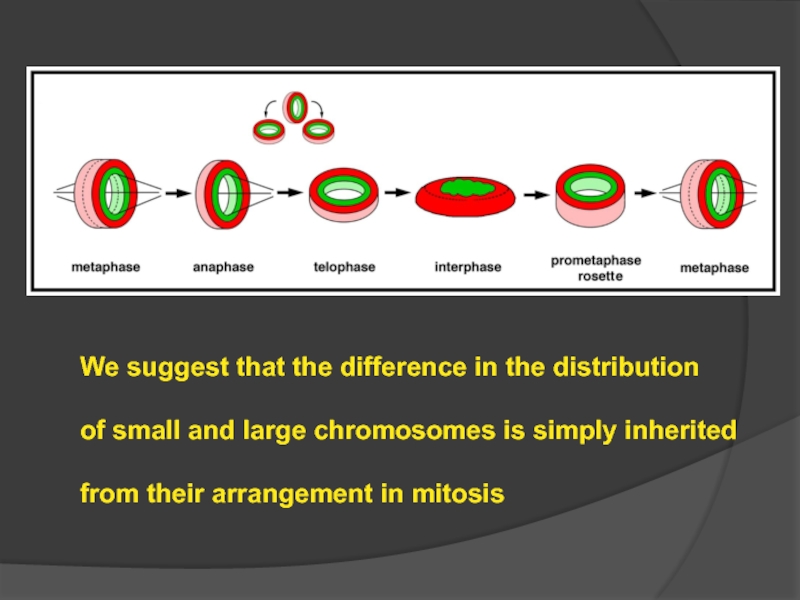
Слайд 95We suggest that the difference in the distribution
of small and
from their arrangement in mitosis
Слайд 96One more important aspect of order
in interphase chromosomes arrangement
Однако, это не
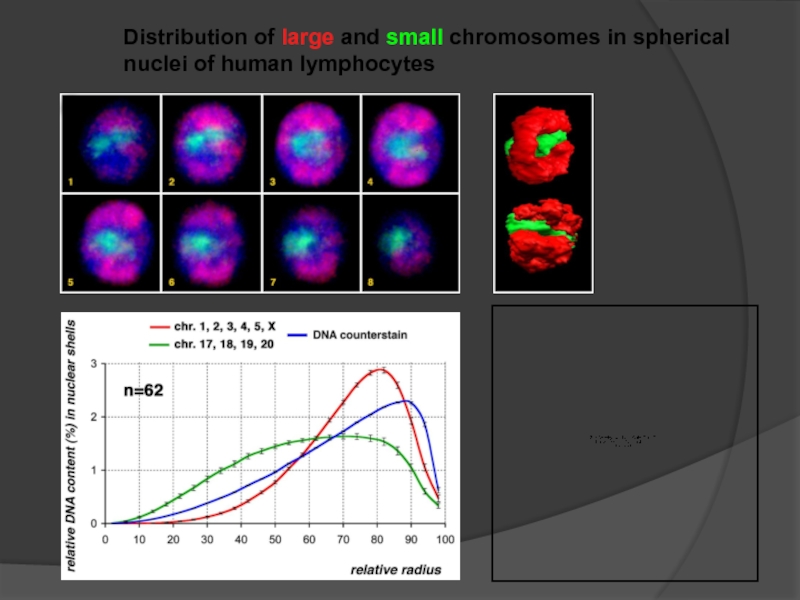
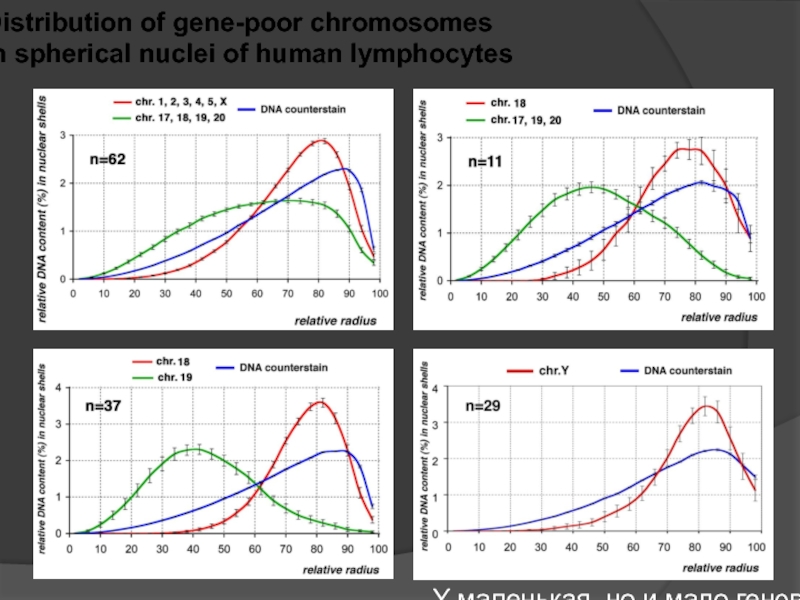
Слайд 97Distribution of gene-poor chromosomes
in spherical nuclei of human lymphocytes
Y маленькая,
Слайд 98Gene content affects chromosome distribution
in spherical nuclei of lymphocytes
Обогащение\обедненность генами влияет
позицию хромосом в круглом ядре лимфоцитов
Слайд 99Distribution of gene-poor and gene-dense chromosomes in spherical nuclei of human
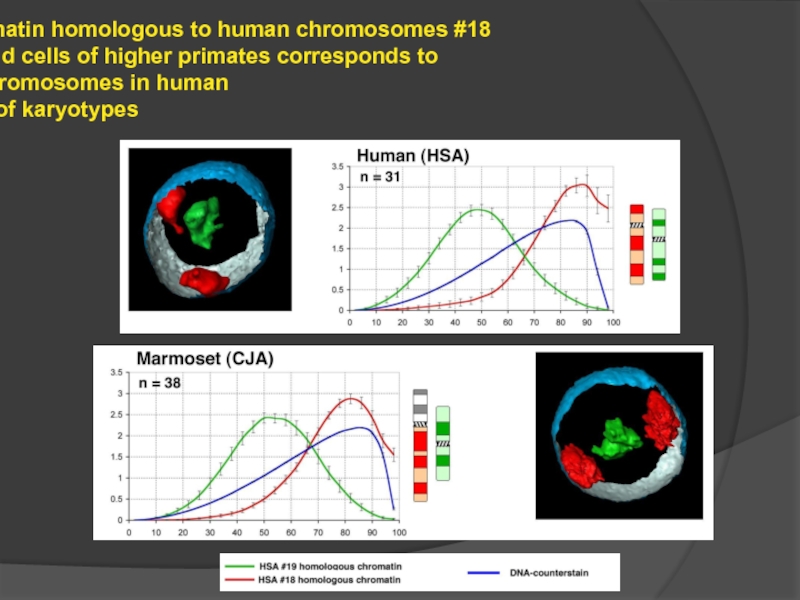
Слайд 100The distribution of chromatin homologous to human chromosomes #18 and #19
Слайд 101Gene density affects the distribution of interphase
chromosomes:
a peripheral position, gene-dense chromosomes
are located in the interior of spherical lymphocyte nuclei
Observations on tumor cells and primates imply that DNA sequences affect the positions of chromosomes containing these specific sequences
Слайд 102The differential positioning of gene-poor and
gene-dense chromosomes necessitates selective
movements during
The forces which drive gene-poor and
gene-dense chromosomes to their specific
positions in interphase nuclei and
timing of these movements remain unknown
Различные позиции ген-бедных и ген-богатых хромосом
требует направленного движения в телофазе\начале G1
Механизм и время распределения хромосом неизвестны
Слайд 103 Nuclei are characterized by a reproducible pattern
Side-by-side chromosome arrangement varies
between nuclei of individual cells
Для ядер характерен воспроизводимый рисунок
радиального распределения хромосом
В ядрах отдельных клеток распределение хромосом
варьирует
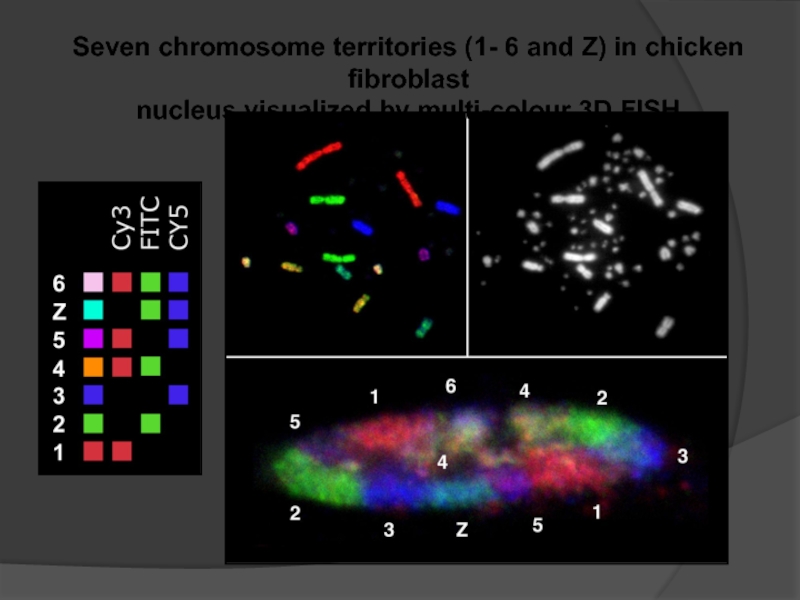
Слайд 104Seven chromosome territories (1- 6 and Z) in chicken fibroblast
nucleus
Слайд 106When and how do chromosomes
change their neigborhood ?
Does this change
Когда и как хромосомы меняют соседей?
Бывают ли изменения в интерфазе?
Слайд 108When and how chromosomes
change their neigborhood ?
Does this change take
NO !
Does it take place during mitosis ?
Хромосомы меняют соседей во время митоза ?
СТ не перемещаются в интерфазе
Слайд 109 What a role do different stages of mitosis play in
Manders et al (1999)
JCB, 144(5): 813-821
+
our observations
?
Во время какой фазы митоза хромосомы перемешиваются?
Слайд 111Symmetrical arrangements of chromosome territories #4 (red), #7 (blue), and #21
The relative positions of chromosomes
change little during anaphase/telophase
Слайд 113Relative positions of chromosome territories #7 and #10
revealed by 3D-
Positions of chromosomes are symmetrical in sister cells
but different in pairs of cousin cells
Слайд 115 Shuffling of chromosomes takes place in prometaphase
During
in metaphase is mainly retained
In telophase / early G1, chromosomal arrangement may be
modified by specific movements of gene-dense and
gene-poor chromosomes
Хромосомы перемещаются друг относительно друга в прометафазе
Порядок хромосом в метафазе сохраняется в телофазе
Позиции хромосом могут модифицироваться в соответствии
с обогащенностью генами в телофазе\G0
Слайд 116Conclusions
The arrangement of chromosome territories is ordered
Chromosome territories do not move significantly
during the interphase
The positions of chromosomes in the interphase nucleus are
basically predetermined by their positions in mitotic cells
Neighborhoods of chromosomes are different
between individual cells. Reshuffling of chromosomes
takes place during the prophase - prometaphase transition
Расположение СТ зависит от величины хромосомы и ее насыщенностью генами
СТ не двигаются в интерфазе
Позиция хромосомы в метафазной пластине определяет позицию СТ в ядре
Слайд 117University of Munich
Department of Biology II
Chair of Anthropology
and Human Genetics
Thomas
Andreas Bolzer
Alessandro Brero
Felix Habermann
Claudia Weierich
Marion Cremer
Hide Tanabe
Michaela Neusser
Stefan Müller
Lothar Schermelleh
Joachim Walter
Daniela Köhler
Kirchhof-Institute of Physics,
University of Heidelberg
Christoph Cremer Laboratory:
Johann von Hase
Enzo Calcagno
Gregor Kreth
J.Wienberg, University of Munich (LMU)
M.Ferguson-Smith, University of Cambridge
M.Speicher, Technical University of Munich (TU)
A.Jauch, University of Heidelberg
K.Sullivan, Scripps Research Institute
W.C.Earnshaw, University of Edinburgh
Слайд 119Cell. 2009 137(2):356-68.
Nuclear architecture of rod photoreceptor cells adapts to vision
Solovei ISolovei I, Kreysing MSolovei I, Kreysing M, Lanctôt CSolovei I, Kreysing M, Lanctôt C, Kösem SSolovei I, Kreysing M, Lanctôt C, Kösem S, Peichl LSolovei I, Kreysing M, Lanctôt C, Kösem S, Peichl L, Cremer TSolovei I, Kreysing M, Lanctôt C, Kösem S, Peichl L, Cremer T, Guck JSolovei I, Kreysing M, Lanctôt C, Kösem S, Peichl L, Cremer T, Guck J, Joffe B