- Главная
- Разное
- Дизайн
- Бизнес и предпринимательство
- Аналитика
- Образование
- Развлечения
- Красота и здоровье
- Финансы
- Государство
- Путешествия
- Спорт
- Недвижимость
- Армия
- Графика
- Культурология
- Еда и кулинария
- Лингвистика
- Английский язык
- Астрономия
- Алгебра
- Биология
- География
- Детские презентации
- Информатика
- История
- Литература
- Маркетинг
- Математика
- Медицина
- Менеджмент
- Музыка
- МХК
- Немецкий язык
- ОБЖ
- Обществознание
- Окружающий мир
- Педагогика
- Русский язык
- Технология
- Физика
- Философия
- Химия
- Шаблоны, картинки для презентаций
- Экология
- Экономика
- Юриспруденция
Next-generation sequencing RNA-Seq. Анализ презентация
Содержание
- 1. Next-generation sequencing RNA-Seq. Анализ
- 2. Шаг 5: Анализ данных
- 3. Особенности программного обеспечения для анализа данных
- 4. ДНК A Приготовление библиотеки B Секвенирование D Анализ данных
- 5. Анализ данных Ion Torrent Определение положений на
- 6. Ошибки случаются… Эксперимент. Ошибки
- 7. Контроль и улучшение качества результатов секвенирования
- 8. 1) Димеры адаптеров. Адаптеры соединяются друг с
- 9. Возможные ошибки Barry Merriman, Ion Torrent R&D
- 10. Тримминг - удаление ошибок секвенирования Две задачи
- 11. P – вероятность ошибки Q – параметр
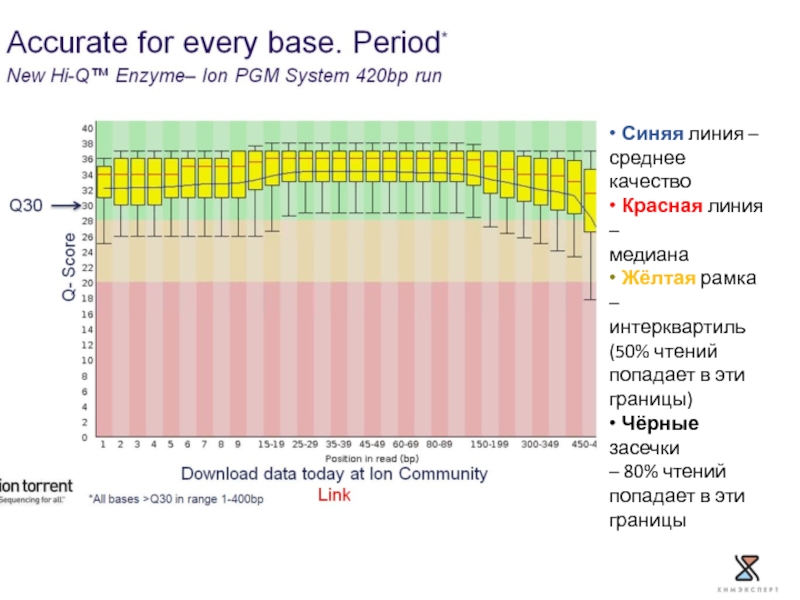
- 12. • Синяя линия – среднее качество •
- 13. FASTQ – общепринятый формат записи чтений. FASTQ
- 14. Качество в формате FASTQ закодировано в ASCII
- 15. Первичный анализ Børsting, C., Fordyce, S. L.,
- 16. Первичный анализ Børsting, C., Fordyce, S. L.,
- 17. Фильтры для ридов Варианты фильтрации прочтений:
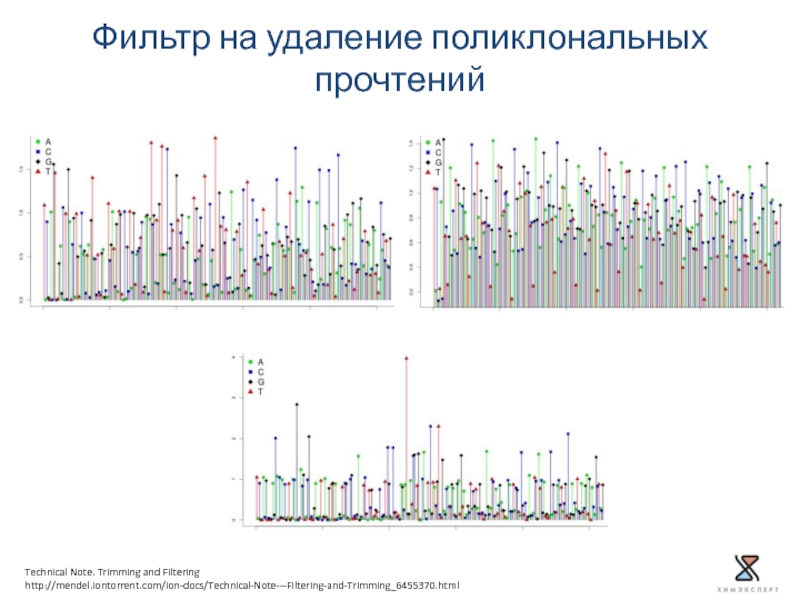
- 18. Фильтр на удаление поликлональных прочтений Technical Note. Trimming and Filtering http://mendel.iontorrent.com/ion-docs/Technical-Note---Filtering-and-Trimming_6455370.html
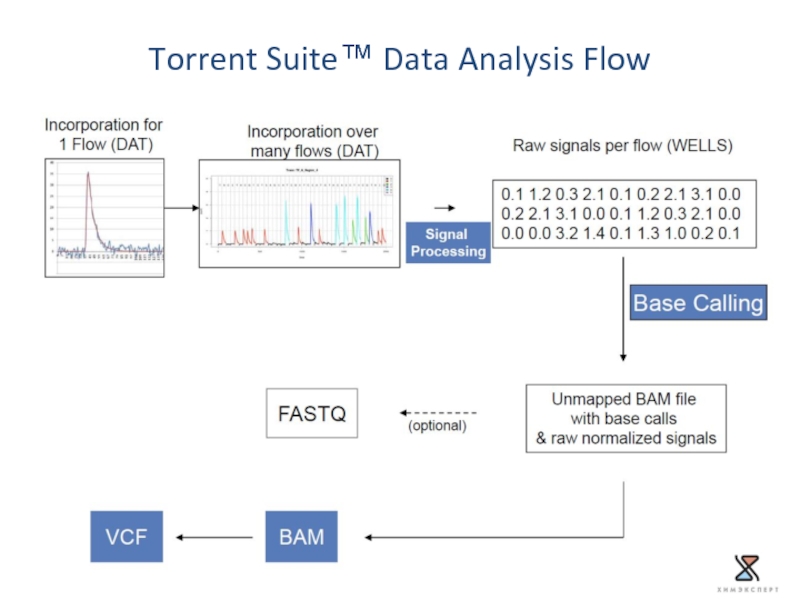
- 19. Torrent Suite™ Data Analysis Flow
- 20. Example shown for 100bp (260 flow) run
- 21. Example shown for 100bp (260 flow) run
- 22. Вторичный анализ (Обработка BAM файлов при помощи
- 23. Шаг 1: Тримминг по качеству 3’ конца
- 24. Анализ транскриптома. Рабочий процесс Life Technologies—Sample
- 25. Ion Proton™ System Enables High Quality Transcriptome
- 26. Дополнительный шаг по обрезанию концов. Низкое
- 27. Программа FastQC использует FASTQ файл в качестве
- 28. Ошибки можно не только удалять, но и
- 29. Что такое kmer size? Как собрать геном
- 30. Для анализа транскриптома следует использовать параметр с
- 31. SAM-формат (Sequence Alignment/Map format) – текстовый
- 32. BAM-формат (Binary Sequence Alignment/Map) - Сжатый
- 33. Integrative Genomics Viewer – программа для визуального просмотра BAM-файлов
- 34. Ion Torrent data from RNA-Seq analysis of
- 35. Повтор Прочтения YIPF2 с антисмысловой цепи Структура
- 36. EWSR1/FLI1 fusion protein type 1 (EWSR1/FLI1 fusion)
- 37. Шаг 4: Подсчет картированных прочтений Скрипт htseq-count.py
- 38. Spike-in РНК: известна последовательность известна конечная концентрация
- 39. Контроль ERCC RNA Spike-In Control Mixes
- 40. TaqMan Expression Data with MAQC Ion Proton™
- 41. Высокий коэффициент корреляции с данными микрочипов Усредненные
- 42. Скрипт на Perl анализирует SAM файл на
- 43. Нормализация данных RNA-Seq необходима из-за различий в
- 44. Обобщенные статистические подходы к анализу транскриптомных данных
- 45. Анализ микроРНК. Рабочий процесс Life Technologies—Sample
- 46. FASTX-toolkit – программный пакет набора инструментов для
- 47. Первый шаг картирования – шпильки микроРНК.
- 48. Шаг 4: Подсчет картированных прочтений Скрипт htseq-count.py
- 49. Скрипт на Perl анализирует SAM файл на
- 50. Torrent Server Torrent Browser Оценка качества
- 51. Облако Biological Interpretation Partek® Genomics Suite™ Partek®
- 52. Характеристики Интеграция с Torrent Suite™ Software
- 53. Количественное картирование транскриптома Обнаружение новых транскриптов
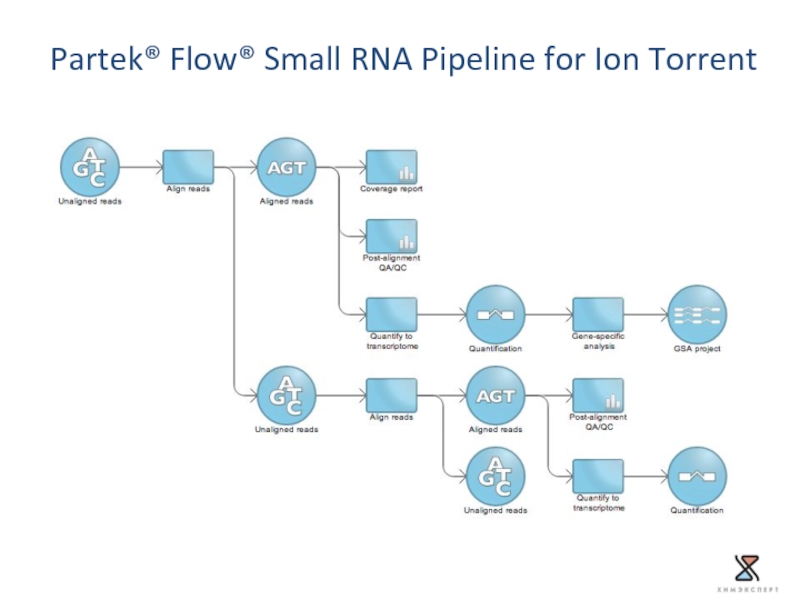
- 54. Partek® Flow® Small RNA Pipeline for Ion Torrent
Слайд 1«Агентство Химэксперт»
Next-generation sequencing
RNA-Seq
RNA-Seq “Анализ”
Первичный анализ и как его делать
Что такое вторичный анализ и для чего он нужен? (картирование, форматы файлов, контроль). Анализ транскриптома. Анализ микроРНК.
Слайд 3Особенности программного обеспечения
для анализа данных NGS
Анализ данных NGS. Брагин Антон,
Модульная организация (за каждую часть анализа отвечает своя небольшая «подпрограмма»)
Для получения результата подпрограммы должны выстраиваться в цепочки, которые называются pipeline. Результат одной подпрограммы в цепочке является исходными данными для следующей подпрограммы.
Каждый компонент pipeline имеет множество настроек. «Правильные» настройки зависят от результата, который требуется получить, и часто неизвестны заранее.
Большинство подпрограмм созданы для операционной системы Linux и не имеют интерфейса пользователя.
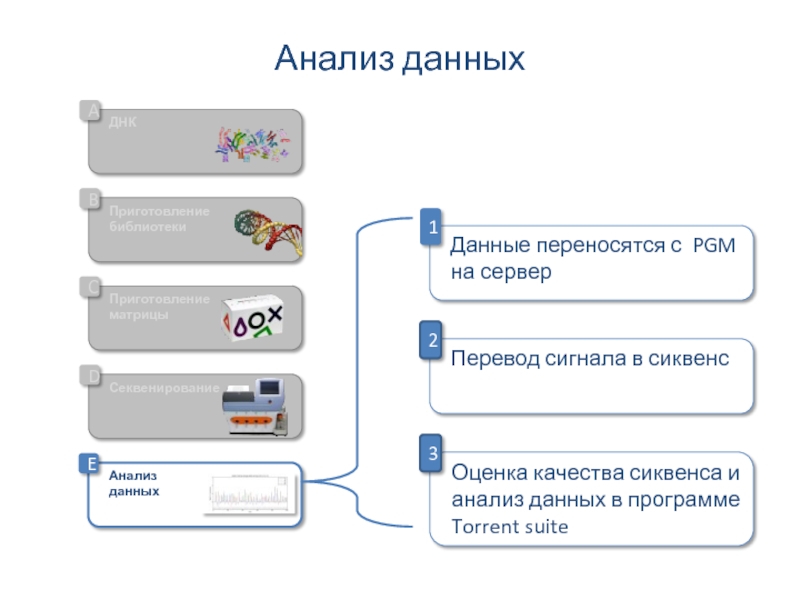
Слайд 5Анализ данных Ion Torrent
Определение положений на чипе, в которых находятся последовательности
Перевод
Фильтрация по качеству
Выравнивание на референсный геном
Поиск генетических вариантов
Анализ данных NGS. Брагин Антон, Sequoia Genetics. 2013.
Слайд 6Ошибки случаются…
Эксперимент. Ошибки постановки эксперимента
Биоинформатика. Ошибки в чтениях возникают из-за неточности
Контроль и улучшение качества результатов
секвенирования
Barry Merriman, Ion Torrent R&D Team, Jonathan M. Rothberg. Progress in Ion Torrent semiconductor chip based sequencing
Electrophoresis 2012, 33, 3397–3417 3397
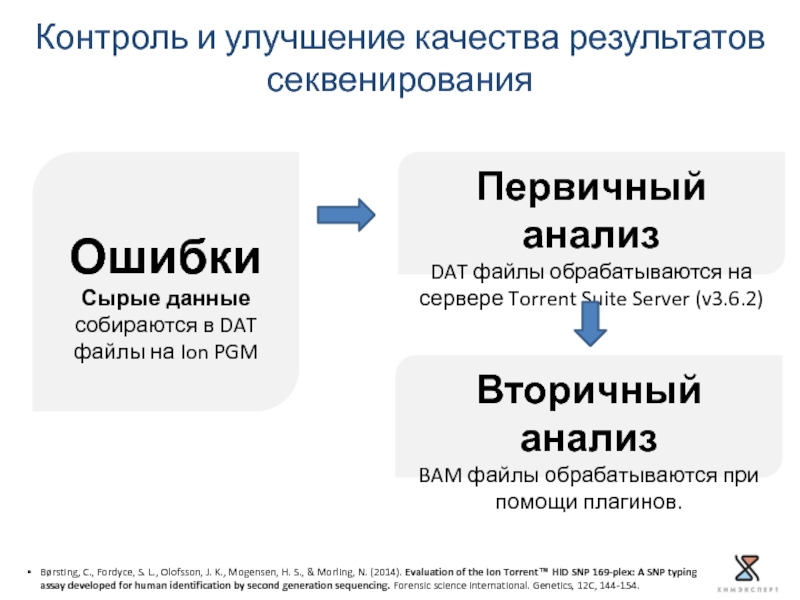
Слайд 7Контроль и улучшение качества результатов
секвенирования
Ошибки Сырые данные собираются в DAT файлы
Первичный анализ
DAT файлы обрабатываются на сервере Torrent Suite Server (v3.6.2)
Вторичный анализ
BAM файлы обрабатываются при помощи плагинов.
Børsting, C., Fordyce, S. L., Olofsson, J. K., Mogensen, H. S., & Morling, N. (2014). Evaluation of the Ion Torrent™ HID SNP 169-plex: A SNP typing assay developed for human identification by second generation sequencing. Forensic science international. Genetics, 12C, 144-154.
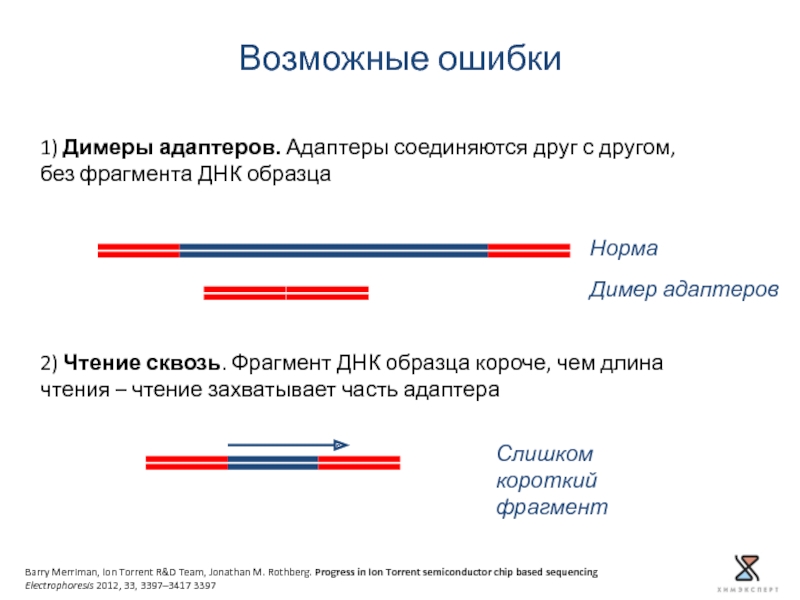
Слайд 81) Димеры адаптеров. Адаптеры соединяются друг с другом, без фрагмента ДНК
Возможные ошибки
Barry Merriman, Ion Torrent R&D Team, Jonathan M. Rothberg. Progress in Ion Torrent semiconductor chip based sequencing
Electrophoresis 2012, 33, 3397–3417 3397
Норма
Димер адаптеров
Слишком короткий фрагмент
2) Чтение сквозь. Фрагмент ДНК образца короче, чем длина чтения – чтение захватывает часть адаптера
Слайд 9Возможные ошибки
Barry Merriman, Ion Torrent R&D Team, Jonathan M. Rothberg. Progress
Electrophoresis 2012, 33, 3397–3417 3397
3) Фазировка. Отдельные олигонуклеотиды в кластере со временем начинают обгонять или отставать от остальных – секвенатору сложнее определить букву
Чем дольше идёт прогон, тем больше будет накапливаться отстающих и опережающих олигонуклеотидов
Слайд 10Тримминг - удаление ошибок секвенирования
Две задачи тримминга:
1. Удаление последовательности адаптера в
2. Отсечение с конца чтений нуклеотидов с низким качеством (например, Q<15)
Technical Note. Trimming and Filtering
http://mendel.iontorrent.com/ion-docs/Technical-Note---Filtering-and-Trimming_6455370.html
(англ. trim – приводить в порядок)
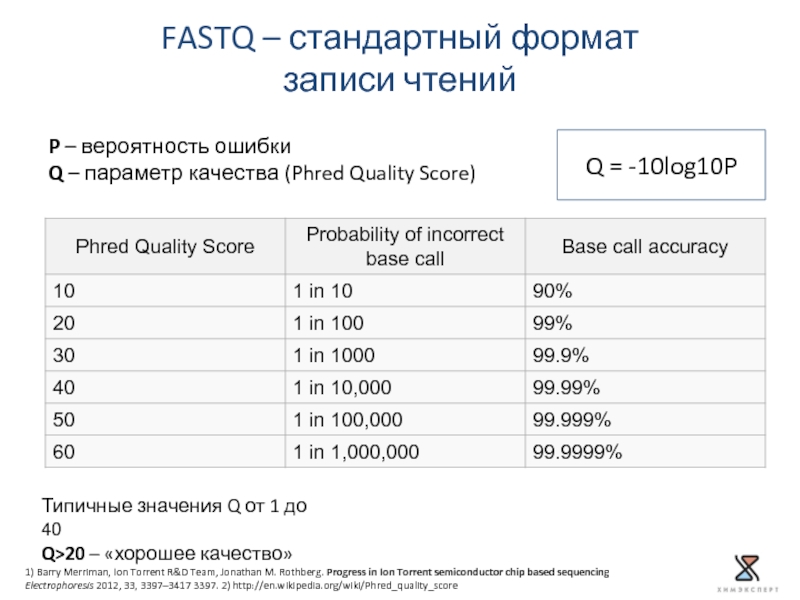
Слайд 11P – вероятность ошибки
Q – параметр качества (Phred Quality Score)
FASTQ –
записи чтений
1) Barry Merriman, Ion Torrent R&D Team, Jonathan M. Rothberg. Progress in Ion Torrent semiconductor chip based sequencing
Electrophoresis 2012, 33, 3397–3417 3397. 2) http://en.wikipedia.org/wiki/Phred_quality_score
Типичные значения Q от 1 до 40
Q>20 – «хорошее качество»
Q = -10log10P
Слайд 12• Синяя линия –
среднее качество
• Красная линия –
медиана
• Жёлтая рамка –
интерквартиль
(50%
попадает в эти
границы)
• Чёрные засечки
– 80% чтений
попадает в эти
границы
Слайд 13FASTQ – общепринятый формат записи чтений.
FASTQ – стандартный формат
записи чтений
Barry Merriman,
Electrophoresis 2012, 33, 3397–3417 3397
Последовательность
Качество
Слайд 14Качество в формате FASTQ закодировано в ASCII – символах
Существовало несколько стандартов
Многим программам важно, чтения во Phred+33 или Phred+64
FASTQ – стандартный формат
записи чтений
http://en.wikipedia.org/wiki/FASTQ_format
Слайд 15Первичный анализ
Børsting, C., Fordyce, S. L., Olofsson, J. K., Mogensen, H.
1. Оценивается фоновый сигнал пустых лунок и вычитается из необработанного сигнала лунок с ионосферами.
2. Необработанные сигналы нормализуются в соответствии с ключевой последовательностью (TCAG) в адаптере.
3. Проводится присваивание названий нуклеотидам (base calling) и проводится оценка качества каждой буквы (оценка Phred).
Слайд 16Первичный анализ
Børsting, C., Fordyce, S. L., Olofsson, J. K., Mogensen, H.
4. Удаляются последовательности низкого качества, димеры праймеров и последовательности от поликлональных ионосфер.
5. Последовательности обрезаются в соответствии с баллами Phred (среднее значение Phred на протяжении 30 п.о. <15), в соответствии с дисбалансом сигнала (> 3% нуклеотидов в последовательности были 0.5-0.59 или 1.4-1.49 п.о. для одной буквы и 1.5-1.59 или 2.4-2.49 п.о. для двух букв) и по последовательности нуклеотидов в 3‘ адаптере.
6. Последовательности выравниваются на референс (эталонная последовательность генома человека – 19) и генерируется бинарный файл с координатами выравнивания (BAM файл)
Слайд 17Фильтры для ридов
Варианты фильтрации прочтений:
1. Удаление коротких прочтений
2. Удаление димеров адаптеров
3.
4. Удаление прочтений с зашкаливающим (off-scale) сигналом
5. Удаление поликлональных прочтений
Technical Note. Trimming and Filtering
http://mendel.iontorrent.com/ion-docs/Technical-Note---Filtering-and-Trimming_6455370.html
Слайд 18Фильтр на удаление поликлональных прочтений
Technical Note. Trimming and Filtering
http://mendel.iontorrent.com/ion-docs/Technical-Note---Filtering-and-Trimming_6455370.html
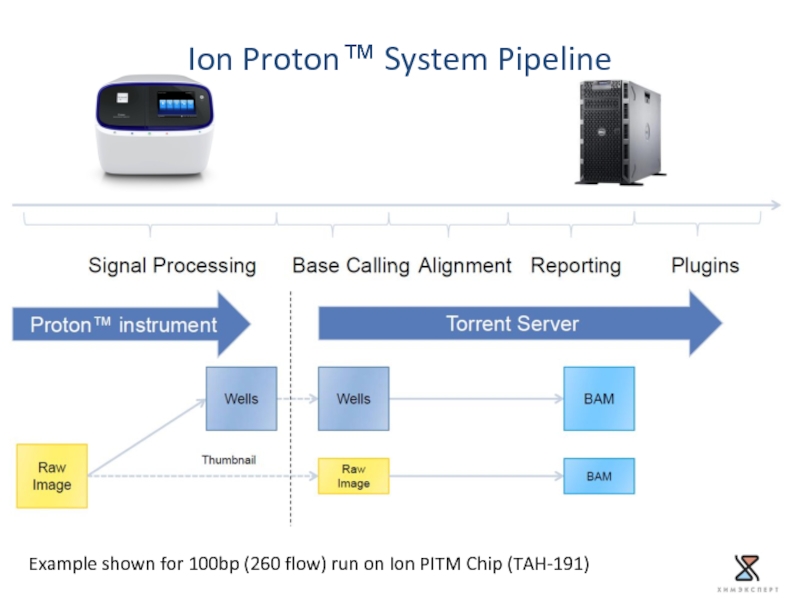
Слайд 20Example shown for 100bp (260 flow) run on Ion PITM Chip
Ion Proton™ System Pipeline
Слайд 21Example shown for 100bp (260 flow) run on Ion PITM Chip
Ion Proton™ System Pipeline
Слайд 22Вторичный анализ
(Обработка BAM файлов при помощи плагинов)
Methods, tools, and pipelines for
Слайд 23Шаг 1: Тримминг по качеству 3’ конца и Ion P1B адаптеру
Шаг
Шаг 3: Картирование на референсный геном или транскриптом
Шаг 4: Подсчет картированных прочтений
Шаг 5: Статистический анализ
Основные задачи
при работе с прочтениями на Ion PGM™ Torrent Server
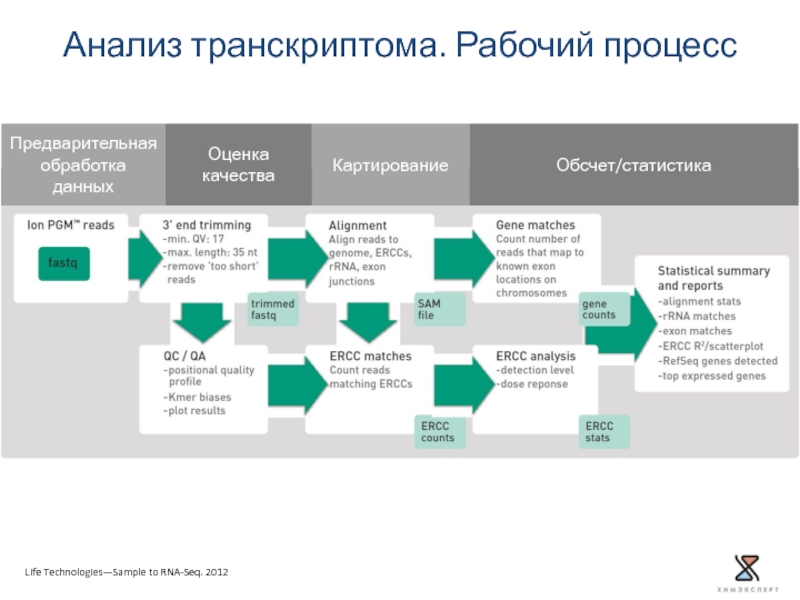
Слайд 24Анализ транскриптома. Рабочий процесс
Life Technologies—Sample to RNA-Seq. 2012
Обсчет/статистика
Картирование
Оценка качества
Предварительная обработка данных
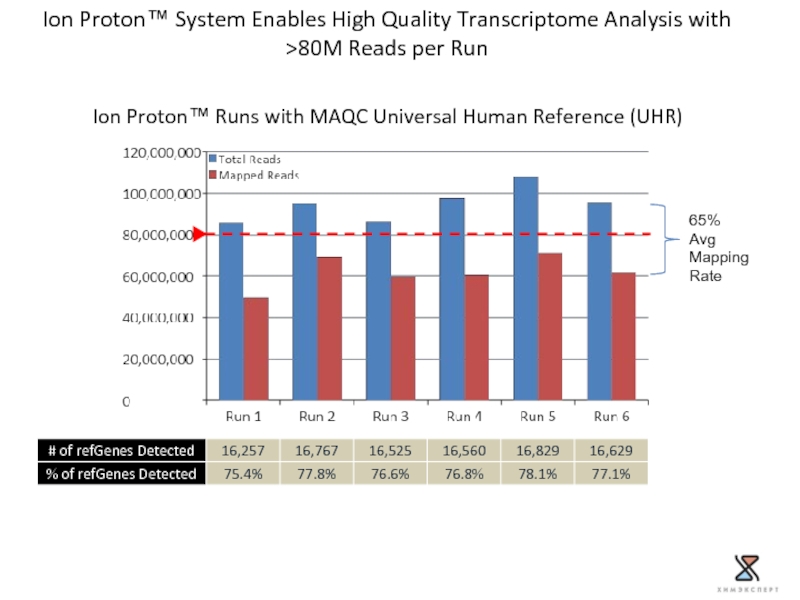
Слайд 25Ion Proton™ System Enables High Quality Transcriptome Analysis with >80M Reads
Ion Proton™ Runs with MAQC Universal Human Reference (UHR)
65% Avg
Mapping
Rate
Слайд 26Дополнительный шаг по обрезанию концов.
Низкое качество и фрагменты адаптерной последовательности приводят
FASTX-toolkit – программный пакет набора инструментов для обработки и оценки FASTQ файлов.
Инструмент fastq_quality_trimmer применяют таким образом, что последовательности ниже минимального значения качества Phred (QV) 17 при сканировании от 5 'к 3' концу прочтения отделяются. Если длина прочтения после обрезка падает ниже 35 оснований, то оно исключается из дальнейшего анализа, чтобы обеспечить более высокую специфичность при выравнивании на референсный геном.
Шаг 1: Тримминг по качеству
3’ конца и Ion P1B адаптеру
Слайд 27Программа FastQC использует FASTQ файл в качестве входных данных
Качество прочтений по
Оценка качества сиквенса
Содержание GC
Содержание недостоверных (N) оснований
Распределение длин прочтений
Повторяющиеся последовательности
Анализ представленности последовательностей
Анализ представленности Kmer-ов
Этот анализ может оказаться полезным, когда на референсный геном картируется меньше прочтений, чем ожидалось.
Шаг 2: Оценка и контроль качества
Per Base Sequence Quality
Per Sequence Quality Scores
Per Base Sequence Content
Per Base GC Content
Per Sequence GC Content
Per Base N Content
Sequence Length Distribution
Duplicate Sequences
Overrepresented Sequences
Overrepresented Kmers
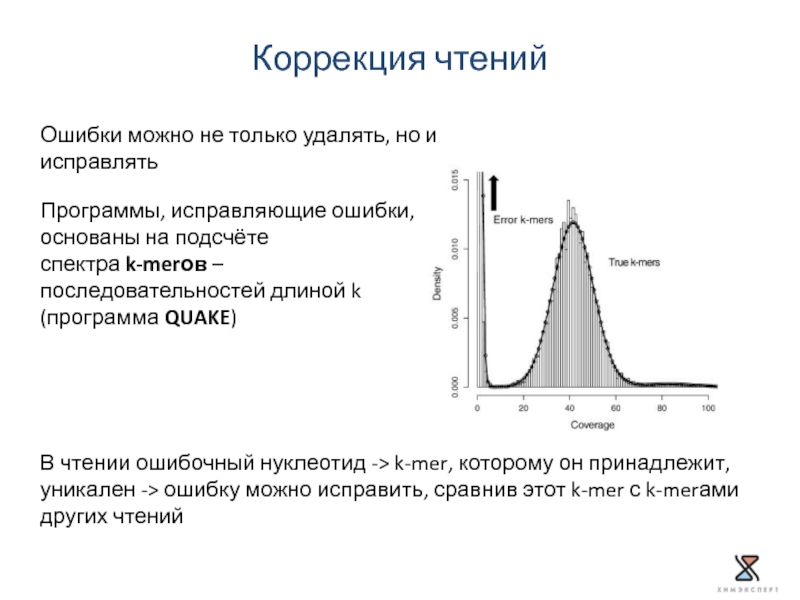
Слайд 28Ошибки можно не только удалять, но и исправлять
Коррекция чтений
Программы, исправляющие ошибки,
спектра k-merов – последовательностей длиной k
(программа QUAKE)
В чтении ошибочный нуклеотид -> k-mer, которому он принадлежит, уникален -> ошибку можно исправить, сравнив этот k-mer с k-merами других чтений
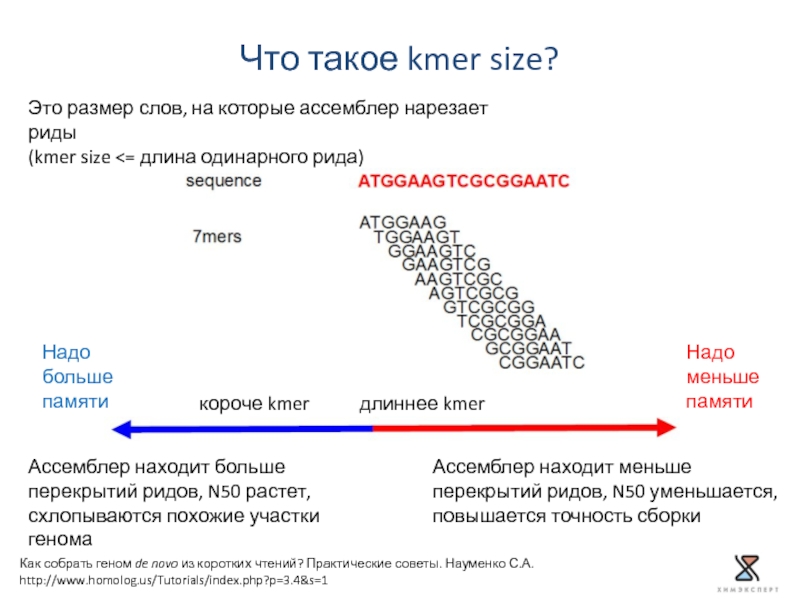
Слайд 29Что такое kmer size?
Как собрать геном de novo из коротких чтений?
http://www.homolog.us/Tutorials/index.php?p=3.4&s=1
Это размер слов, на которые ассемблер нарезает риды
(kmer size <= длина одинарного рида)
короче kmer длиннее kmer
Надо больше
памяти
Надо меньше
памяти
Ассемблер находит больше перекрытий ридов, N50 растет, схлопываются похожие участки генома
Ассемблер находит меньше перекрытий ридов, N50 уменьшается, повышается точность сборки
Слайд 30Для анализа транскриптома следует использовать параметр с длиной фрагмента для анализа
На выходе: файл формата SAM (Sequence Alignment Map)
Бинарный вариант этого формата: файл BAM
(файл формата BAM можно получить при помощи SamTools)
Шаг 3: Картирование
Программа-картировщик TMAP
(The Torrent Mapping Alignment Program for Ion Torrent Data)
Слайд 31SAM-формат
(Sequence Alignment/Map format)
– текстовый формат, предназначенный для представления информация о
r001/1 и r001/2 спаренные риды
r003 химерный рид
r004 разделенное выравнивание
ID чтения
хромосома и координата, куда "легло" чтение
качество картирования согласно картировщику
Слайд 32BAM-формат
(Binary Sequence Alignment/Map)
- Сжатый бинарный вариант формата SAM. Для быстрого
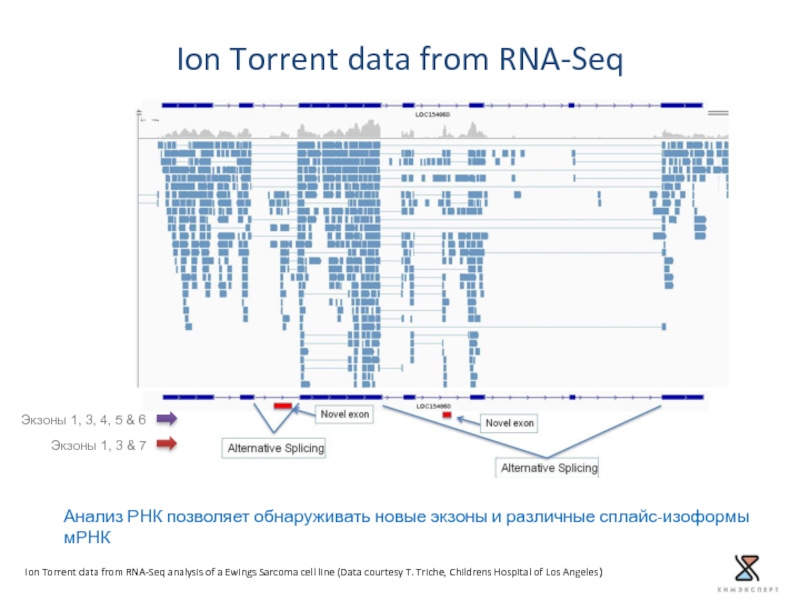
Слайд 34Ion Torrent data from RNA-Seq analysis of a Ewings Sarcoma cell
Экзоны 1, 3 & 7
Экзоны 1, 3, 4, 5 & 6
Ion Torrent data from RNA-Seq
Анализ РНК позволяет обнаруживать новые экзоны и различные сплайс-изоформы мРНК
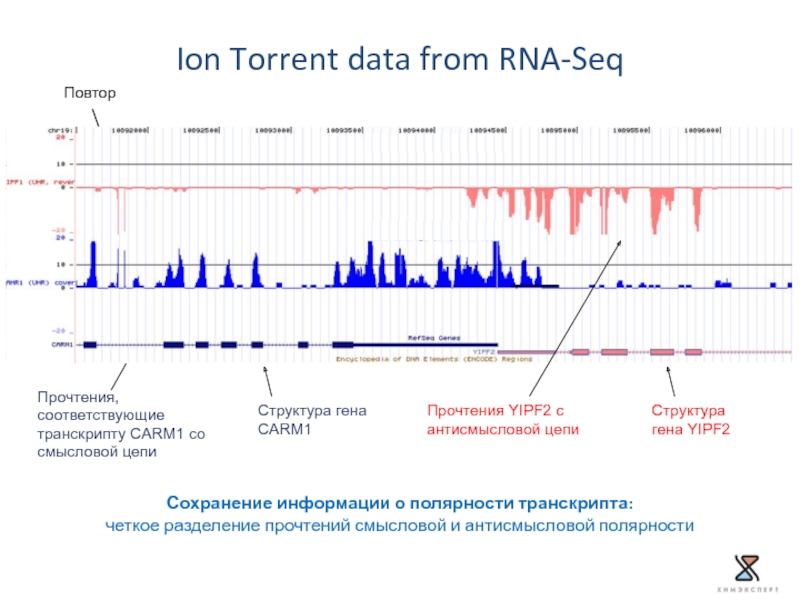
Слайд 35Повтор
Прочтения YIPF2 с антисмысловой цепи
Структура гена YIPF2
Структура гена CARM1
Прочтения, соответствующие
Ion Torrent data from RNA-Seq
Сохранение информации о полярности транскрипта:
четкое разделение прочтений смысловой и антисмысловой полярности
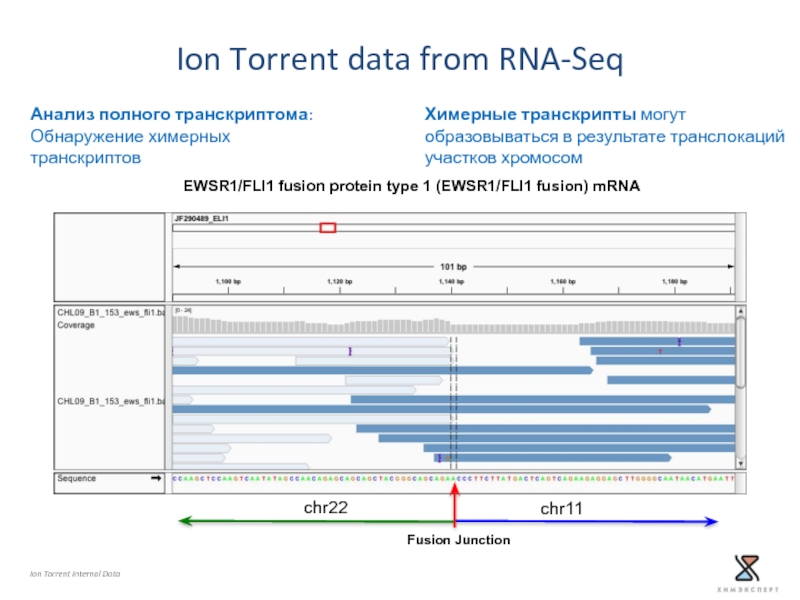
Слайд 36EWSR1/FLI1 fusion protein type 1 (EWSR1/FLI1 fusion) mRNA
Ion Torrent Internal Data
Ion
Анализ полного транскриптома:
Обнаружение химерных транскриптов
Химерные транскрипты могут образовываться в результате транслокаций участков хромосом
Слайд 37Шаг 4: Подсчет картированных прочтений
Скрипт htseq-count.py для подсчета картированных прочтений
Скрипт –
Что делает:
из SAM-файла извлекает координаты картированного прочтения
определяет перекрывание с известными экзонами из RefSeq.
Информацию об известных экзонах можно загрузить с сайта UCSC Genome Browser в формате файла RefGene GTF.
Для подсчета Контроля (92 варианта последовательностей ERCC) – добавляем их в файл RefGene GTF.
Слайд 38Spike-in РНК:
известна последовательность
известна конечная концентрация
используется для оценки точности измерений дифференциальной
92 варианта полиаденилированных транскриптов
размер транскриптов 250-2000 нукл.
«мимикрирует» под естественную эукариотическую мРНК
Контроль
ERCC RNA Spike-In Control Mixes
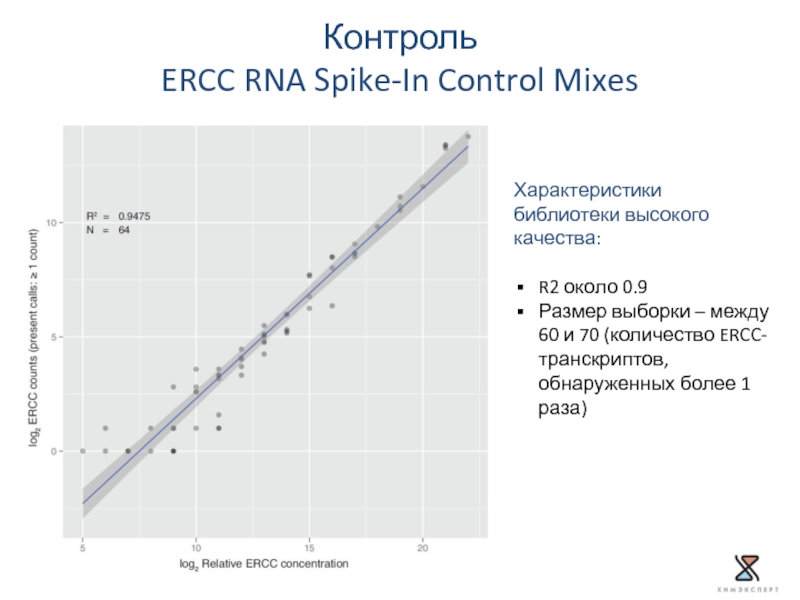
Слайд 39Контроль
ERCC RNA Spike-In Control Mixes
Характеристики библиотеки высокого качества:
R2 около 0.9
Размер
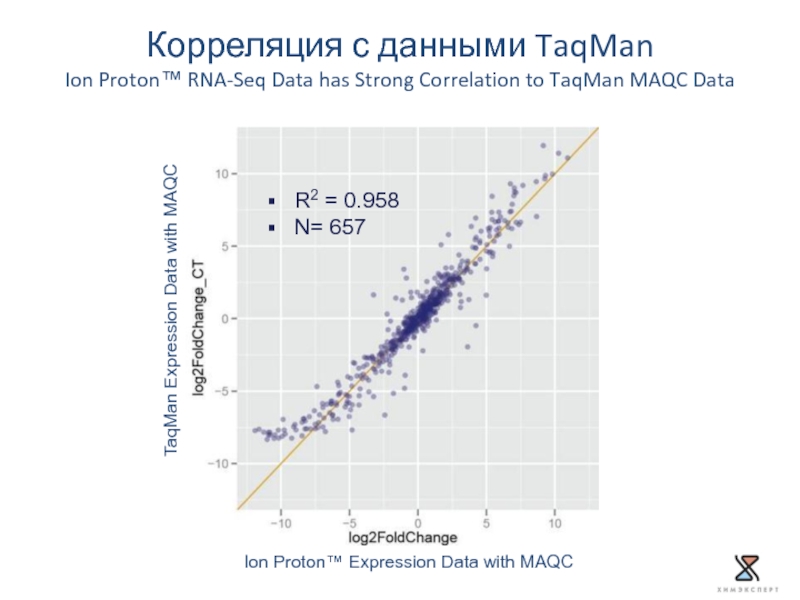
Слайд 40TaqMan Expression Data with MAQC
Ion Proton™ Expression Data with MAQC
R2 =
N= 657
Корреляция с данными TaqMan
Ion Proton™ RNA-Seq Data has Strong Correlation to TaqMan MAQC Data
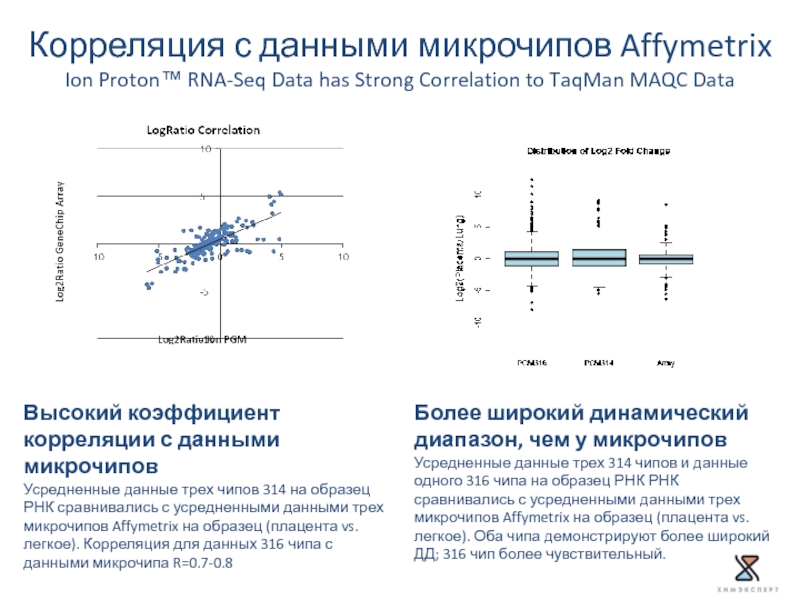
Слайд 41Высокий коэффициент корреляции с данными микрочипов
Усредненные данные трех чипов 314 на
Более широкий динамический диапазон, чем у микрочипов
Усредненные данные трех 314 чипов и данные одного 316 чипа на образец РНК РНК сравнивались с усредненными данными трех микрочипов Affymetrix на образец (плацента vs. легкое). Оба чипа демонстрируют более широкий ДД; 316 чип более чувствительный.
Корреляция с данными микрочипов Affymetrix
Ion Proton™ RNA-Seq Data has Strong Correlation to TaqMan MAQC Data
Слайд 42Скрипт на Perl анализирует SAM файл на базовую статистику по картированию:
общее количество картированных ридов,
риды, картировавшиеся на экзоны,
слияния экзонов,
рРНК,
риды, картировавшиеся на неаннотированные участки генома.
Также можно провести нормализацию библиотеки.
Шаг 5: Статистика
Слайд 43Нормализация данных RNA-Seq необходима из-за различий в
глубине секвенирования,
длине генов,
отличий между образцами по количеству молекул,
покрытии разных мРНК (сильно различается)
Нормализация данных RNA-Seq
Цифровая нормализация (digital normalization) – уменьшение покрытия слишком представленных транскриптов
Нормализация с помощью программы khmer позволяет уменьшить количество чтений ~ в сто раз без потери качества сборки.
Скорость сборки также увеличивается ~ в сто раз
Также нормализация поможет при single-cell секвенировании
Слайд 44Обобщенные статистические подходы к анализу транскриптомных данных и рекомендации по данному
В случае данных абсолютной экспрессии RNA-Seq возможны прямые сравнения величин.
Удобство прямых сравнений заключается в возможности анализа различных комбинаций экспрессионных данных.
В случае RNA-Seq используются обобщенные линейные модели (ОЛМ), среди которых наиболее распространены логистическая и Пуассоновская регрессия.
С развитием и внедрением RNA-Seq совершенствуются и математические подходы, хотя они являются производными от классических ОЛМ.
Статистические подходы к анализу
экспрессионных данных
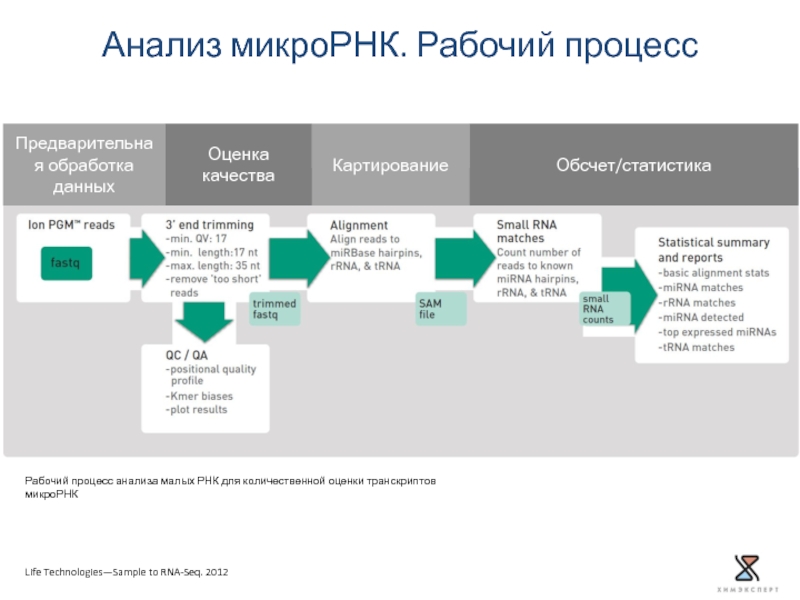
Слайд 45Анализ микроРНК. Рабочий процесс
Life Technologies—Sample to RNA-Seq. 2012
Обсчет/статистика
Картирование
Оценка качества
Предварительная обработка данных
Рабочий
Слайд 46FASTX-toolkit – программный пакет набора инструментов для обработки и оценки FASTQ
fastq_quality_trimmer применяется таким же образом, как при WT:
последовательности ниже Phred (QV) 17 при сканировании от 5 'к 3' концу прочтения отделяются.
Но! Минимальная длина прочтения 17 оснований.
Шаг 1: Тримминг по качеству
3’ конца и Ion P1B адаптеру
Слайд 47Первый шаг картирования – шпильки микроРНК.
Предшественники микроРНК (60-90 нуклеотидов) хранятся
Второй шаг – картирование тРНК и рРНК.
Третий шаг – картирование пропущенных ранее последовательностей адаптера
На выходе: файл SAM формата либо файл BLAST alignment format.
Шаг 3: Картирование
SHRiMP – картировщик коротких прочтений на референс малых РНК, содержащий последовательности шпилечных структур микроРНК, тРНК, рРНК и 3’ адаптера.
Слайд 48Шаг 4: Подсчет картированных прочтений
Скрипт htseq-count.py для подсчета картированных прочтений
Что делает:
из
Что на выходе:
Файл с колонками IDs и подсчетами для каждого предшественника микроРНК, тРНК и рРНК
Слайд 49Скрипт на Perl анализирует SAM файл на базовую статистику по картированию:
общее количество картированных ридов,
риды, картировавшиеся на экзоны,
слияния экзонов,
рРНК,
риды, картировавшиеся на неаннотированные участки генома.
Дополнительно:
Количество адаптеров без вставки
Количество обнаруженных микроРНК из базы miRBase
Шаг 5: Статистика
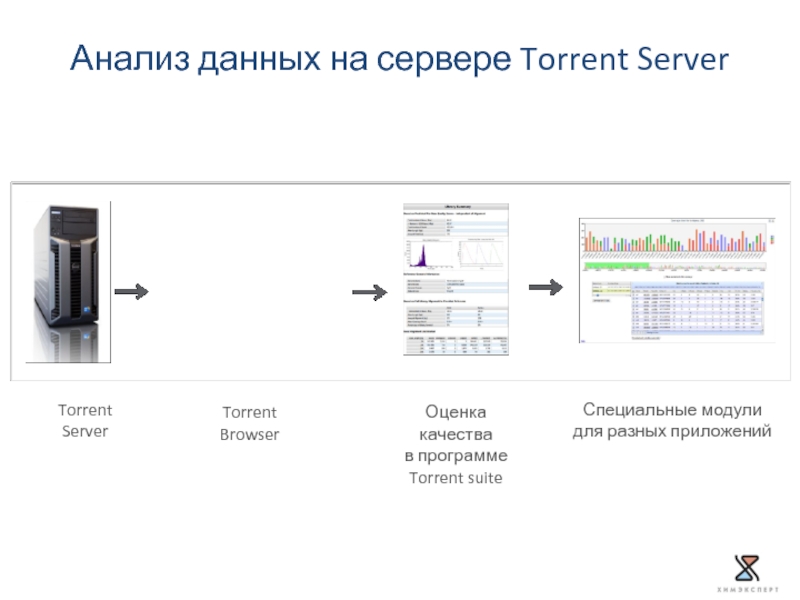
Слайд 50Torrent Server
Torrent Browser
Оценка качества
в программе
Torrent suite
Специальные модули
для разных приложений
Анализ
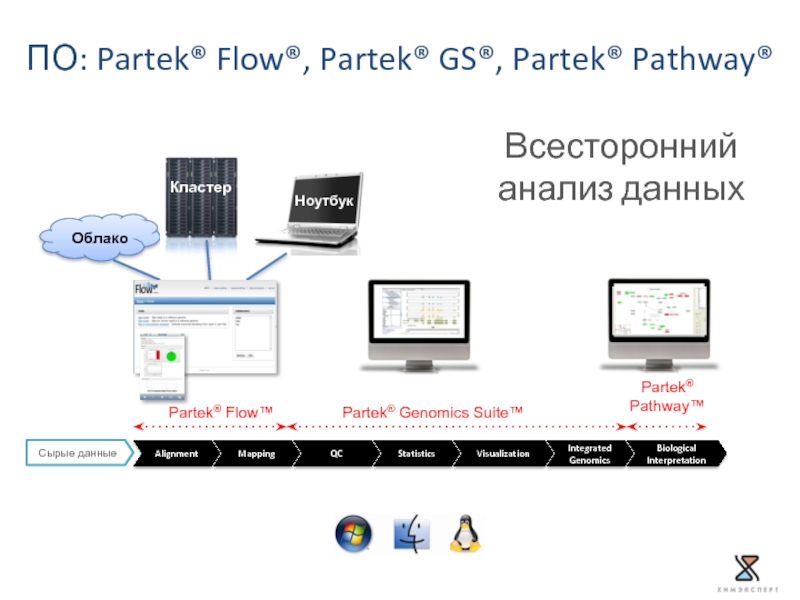
Слайд 51Облако
Biological
Interpretation
Partek® Genomics Suite™
Partek® Flow™
Partek®
Pathway™
Сырые данные
Copyright © 2011 Partek Incorporated. All
ПО: Partek® Flow®, Partek® GS®, Partek® Pathway®
Всесторонний анализ данных
Слайд 52Характеристики
Интеграция с Torrent Suite™ Software
Работает в облаке и на кластере
Online доступ
Pre-installed Analysis Pipelines for Ion Torrent™ RNA-Seq Data
AmpliSeq Pipeline
Whole Transcriptome Pipeline
Small RNA-Seq Pipeline
Partek® Flow® analysis software
Слайд 53Количественное картирование транскриптома
Обнаружение новых транскриптов
Использование всех доступных баз данных
Оценка представленности
Анализ дифференциальной экспрессии
Обнаружение дифференциальной экспрессии
Анализ ген-специфических параметров
Определение сплайс-вариантов
Variant Detection
Обнаружение фьюжн-генов
Обнаружение и аннотация SNPs
Обнаружение SNVs
Классификация (intronic, exonic, UTR, etc.)
Прогнозирование влияния на
аминокислотную последовательность
Partek® Flow® analysis software