Вашей последовательности
Дизайн праймеров
Анализ ДНК-состава
Повторы в ДНК
Как искать гены? (прокариоты, эукариоты)
Тривиальные случаи применения сборки фрагментов
- Главная
- Разное
- Дизайн
- Бизнес и предпринимательство
- Аналитика
- Образование
- Развлечения
- Красота и здоровье
- Финансы
- Государство
- Путешествия
- Спорт
- Недвижимость
- Армия
- Графика
- Культурология
- Еда и кулинария
- Лингвистика
- Английский язык
- Астрономия
- Алгебра
- Биология
- География
- Детские презентации
- Информатика
- История
- Литература
- Маркетинг
- Математика
- Медицина
- Менеджмент
- Музыка
- МХК
- Немецкий язык
- ОБЖ
- Обществознание
- Окружающий мир
- Педагогика
- Русский язык
- Технология
- Физика
- Философия
- Химия
- Шаблоны, картинки для презентаций
- Экология
- Экономика
- Юриспруденция
Что можно делать с одиночной последовательностью ДНК? презентация
Содержание
- 1. Что можно делать с одиночной последовательностью ДНК?
- 2. Как выявить векторные сегменты в Вашей последовательности?
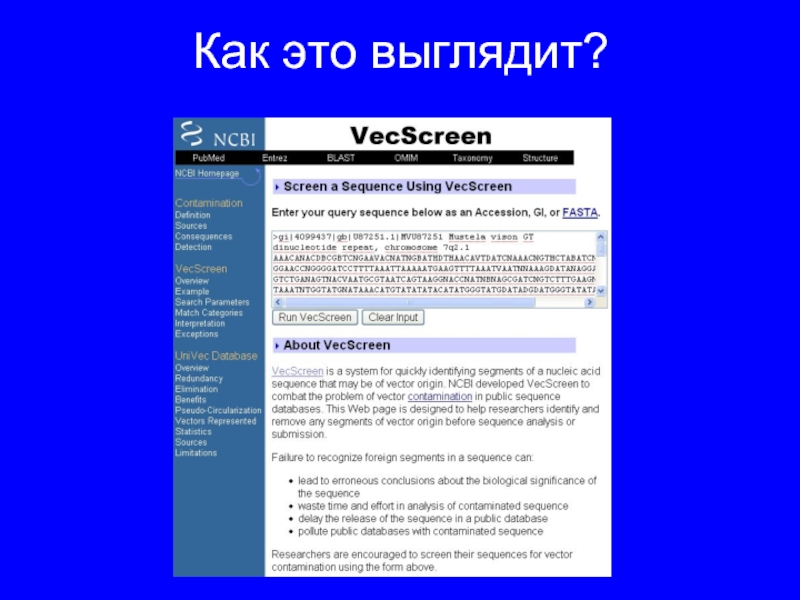
- 3. Как это выглядит?
- 4. VecScreen - output “Non-significant similarity found” – ok! В нашем случае:
- 5. Как интерпретировать результаты VecScreen? Если сегменты
- 6. Почему надо бояться загрязнения ДНК чужеродными сегментами?
- 7. Карта рестрикционных фрагментов Еще одна возможность
- 8. REBASE
- 9. RestrictionMapper
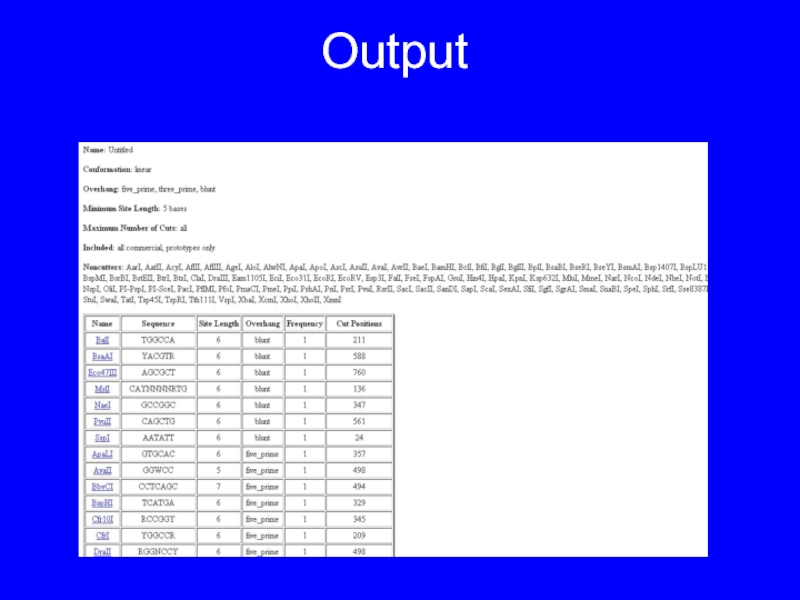
- 10. Output
- 11. Дизайн праймеров для PCR http://biotools.umassmed.edu/
- 12. Primer3 Output – простой текстовый формат, предлагает
- 13. Что можно варьировать? Искать только левый или
- 14. Анализ ДНК-состава G+C – состав Статистика ди-
- 15. Зачем анализировать статистику ДНК? GC-состав: (динамика плавления)
- 16. Как это делать? Это самые элементарные программы
- 17. Какие программы выбрать?
- 18. Как искать повторы в ДНК? Внутренние повторы
- 19. Dot-Plot approach http://arbl.cvmbs.colostate.edu/ molkit/
- 20. Как оценить сколько одинаковых слов много, а
- 21. Геном-специфические повторы: RepeatMasker http://www.repeatmasker.org/cgi-bin/WEBRepeatMasker
- 22. Поиск (белок-кодирующих) генов Прокариоты – просто поиск
- 23. Output Открытые рамки сортированы по длине
- 24. Более точное предсказание –GeneMark (HMM) http://opal.biology.gatech.edu/ GeneMark/
- 25. Heuristic Model input window Если Вы знаете
- 26. Output Графический формат – посмотреть дома!
- 27. Предсказание внутренних экзонов (позвоночные) Принцип: ищут
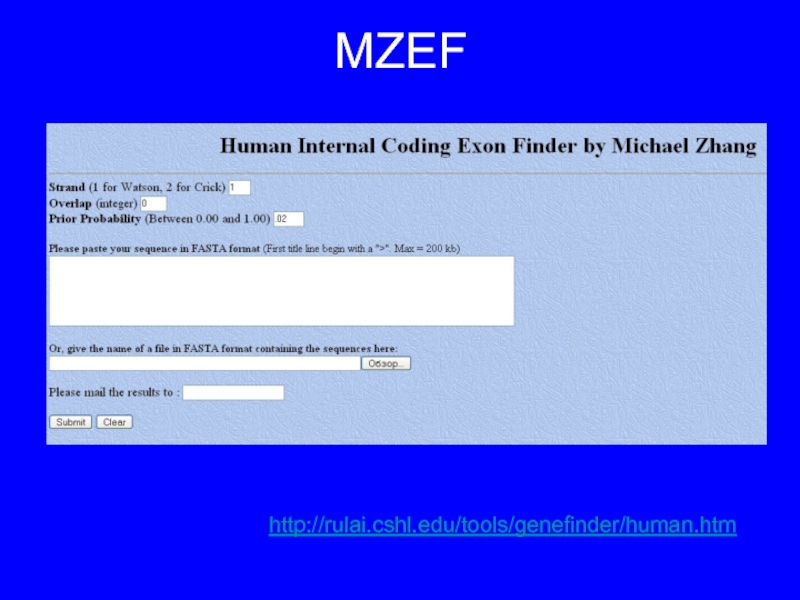
- 28. MZEF http://rulai.cshl.edu/tools/genefinder/human.htm
- 29. MZEF - output Результат работы программы на
- 30. Поиск генов: GenomeScan На основе HMM (учитывает
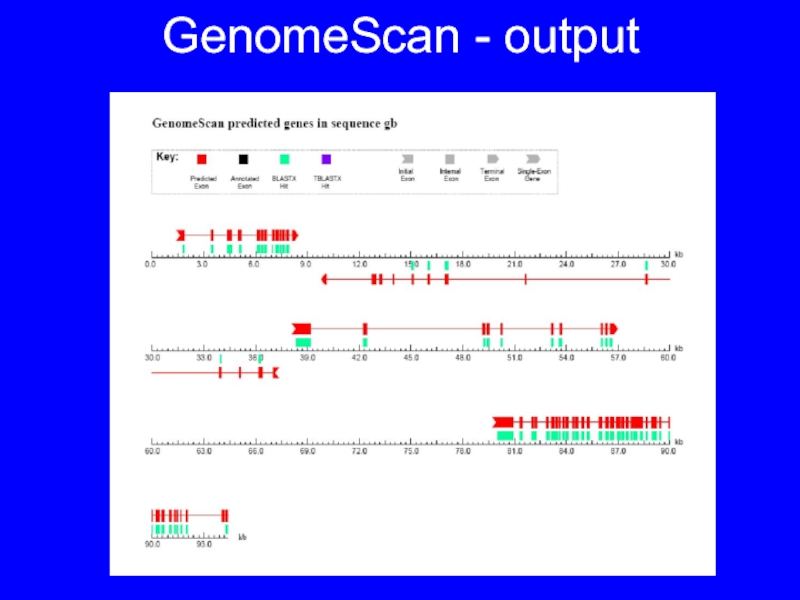
- 31. GenomeScan - output
- 32. Сборка геномных фрагментов в контиги: EGassembler http://egassembler.hgc.jp/
- 33. EGassembler - output
- 34. Поиски регуляторных сигналов Пока поиск слишком несовершенен
- 35. Positional weight matrix (PWM) I = Σj
Слайд 1Что можно делать с одиночной последовательностью ДНК?
Как исключить векторные фланки?
Рестрикционная карта
Слайд 2Как выявить векторные сегменты в Вашей последовательности?
Просто сравнить с исходным вектором?
VecScreen:
http://www.ncbi.nlm.nih.gov/VecScreen/VecScreen_docs.html
“VecScreen
is a system for quickly identifying segments of a nucleic acid sequence that may be of vector origin. NCBI developed VecScreen to minimize the incidence and impact of vector contamination“VecScreen is a system for quickly identifying segments of a nucleic acid sequence that may be of vector origin. NCBI developed VecScreen to minimize the incidence and impact of vector contamination in public sequence databases. GenBank Annotation Staff use VecScreen to verify that sequences submitted for inclusion in the database are free from contaminating vector sequence. Any sequence can be screened for vector contamination using the VecScreen Web site”
Слайд 5Как интерпретировать результаты VecScreen?
Если сегменты гомологии с векторов по краям –
просто удалить их
Если в нескольких местах по всей длине – проще всего… все это выбросить
(!) Не надо выбрасывать, если:
Вектор не ваш – он может быть просто родственным (100% сходство!)
Ваш ген мог быть основой для вектора
Но: если Вы видите неожиданную гомологию к E.coli или дрожжам – задумайтесь!
Если в нескольких местах по всей длине – проще всего… все это выбросить
(!) Не надо выбрасывать, если:
Вектор не ваш – он может быть просто родственным (100% сходство!)
Ваш ген мог быть основой для вектора
Но: если Вы видите неожиданную гомологию к E.coli или дрожжам – задумайтесь!
Слайд 6Почему надо бояться загрязнения ДНК чужеродными сегментами?
Быть уверенным в том, что
Вы анализируете (и не тратить время зря)
Ошибки распространяются по базам данных с экспоненциальной скоростью: неверная информация, проблемы сборки и т.п.
В Swiss-Prot даже были специальные записи (P39188 – P39195: Alu-derived белки)
Будьте внимательны при работах с базами данных! (неожиданно высокая гомология к бактериям в эукариотах и т.п.)
Ошибки распространяются по базам данных с экспоненциальной скоростью: неверная информация, проблемы сборки и т.п.
В Swiss-Prot даже были специальные записи (P39188 – P39195: Alu-derived белки)
Будьте внимательны при работах с базами данных! (неожиданно высокая гомология к бактериям в эукариотах и т.п.)
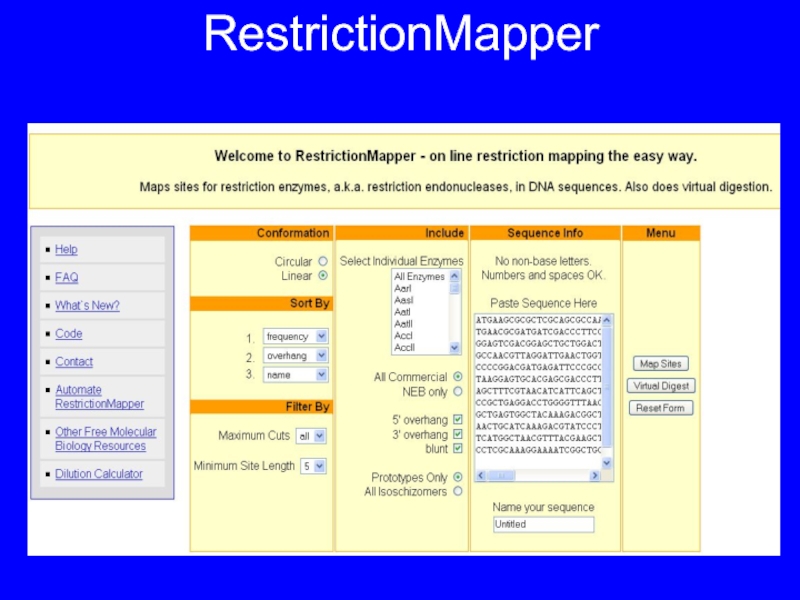
Слайд 7Карта рестрикционных
фрагментов
Еще одна возможность проверить сиквенс на идентичность с тем,
что Вы ожидаете (годится, также, для длинных геномных кусков вплоть до бактериальных геномов)
Все сайты рестрикции лежат в базе данных REBASE (http://rebase.neb.com/)
Как предсказать список рестрикционных фрагментов?
Все сайты рестрикции лежат в базе данных REBASE (http://rebase.neb.com/)
Как предсказать список рестрикционных фрагментов?
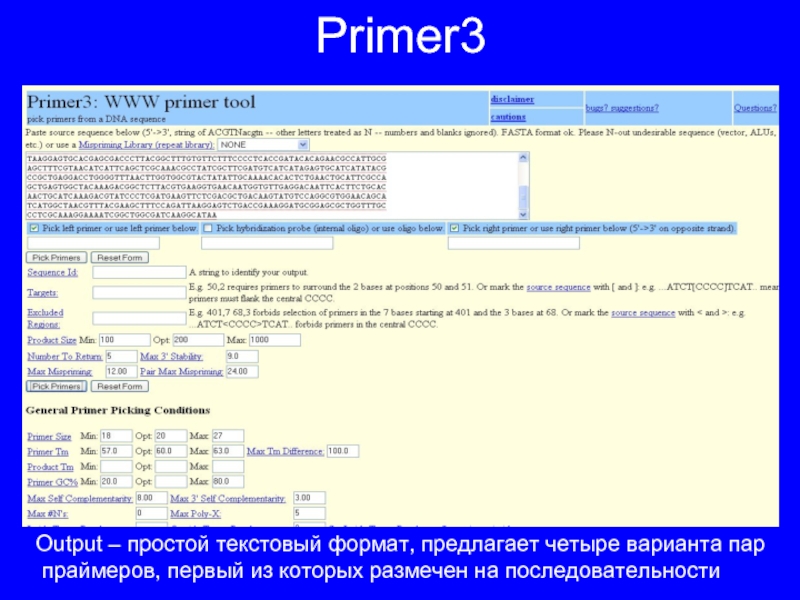
Слайд 12Primer3
Output – простой текстовый формат, предлагает четыре варианта пар
праймеров, первый
из которых размечен на последовательности
Слайд 13Что можно варьировать?
Искать только левый или правый праймер, или пробу для
гибридизации
Предлагать свой собственный левый или правый праймер
Выбрать последовательность, которую Вы хотите включить или наоборот исключить из амплифицированного фрагмента
Выбрать диапазон длины фрагмента
Выбрать диапазон размера олигонуклеотидов, GC-состав, точку плавления
…
Предлагать свой собственный левый или правый праймер
Выбрать последовательность, которую Вы хотите включить или наоборот исключить из амплифицированного фрагмента
Выбрать диапазон длины фрагмента
Выбрать диапазон размера олигонуклеотидов, GC-состав, точку плавления
…
Слайд 14Анализ ДНК-состава
G+C – состав
Статистика ди- и три- нуклеотидов
(не путайте
статистику тринуклеотидов и codon usage)
Частота более длинных слов
Частота более длинных слов
Слайд 15Зачем анализировать статистику ДНК?
GC-состав: (динамика плавления)
Ди- и тринуклеотиды - уникальная геномная
подпись:
Идентификация загрязнения вектором
Свидетельство параллельного переноса
Островки патогенности
Классификация метагеномных контигов
Выявление origin репликации
Более длинные слова – регуляторные сигналы
Идентификация загрязнения вектором
Свидетельство параллельного переноса
Островки патогенности
Классификация метагеномных контигов
Выявление origin репликации
Более длинные слова – регуляторные сигналы
Слайд 16Как это делать?
Это самые элементарные программы – обычно установлены на компьютере
EMBOSS
(European Molecular Biology Open Software Suite) – бесплатный пакет (~ 100 модулей, только под Unix)
Web:
http://www.genomatix.de/cgi-bin/tools/tools.pl http://bioweb.pasteur.fr/intro-uk.html
Осмысленно смотреть “скользящим окном”
Web:
http://www.genomatix.de/cgi-bin/tools/tools.pl http://bioweb.pasteur.fr/intro-uk.html
Осмысленно смотреть “скользящим окном”
Слайд 18Как искать повторы в ДНК?
Внутренние повторы – сегменты, встречающиеся чаще, чем
ожидается
Могут быть несовершенными – отличаться одной или несколькими буквами
Что лучше – 5 точных букв, 9 из 10 или 111 из 145? Разные score. Какой выбрать порог?
=> Много программ и несопоставимые результаты. Нельзя верить отрицательным результатам
Могут быть несовершенными – отличаться одной или несколькими буквами
Что лучше – 5 точных букв, 9 из 10 или 111 из 145? Разные score. Какой выбрать порог?
=> Много программ и несопоставимые результаты. Нельзя верить отрицательным результатам
Слайд 20Как оценить сколько одинаковых слов много, а сколько нет
Статистическая модель –
следует вероятность слова
Самый простой расчет: CTGA - 10 раз в последовательности длины 5000. Оценим вероятность: в каждой позиции - ¼*¼*¼*¼ = 1/256. Всего должно быть – 5000*1/256 ~ 20 раз
Если от ожидания отличается меньше, чем в 2 раза – все нормально. То есть от 10 до 40 раз - ок
Самый простой расчет: CTGA - 10 раз в последовательности длины 5000. Оценим вероятность: в каждой позиции - ¼*¼*¼*¼ = 1/256. Всего должно быть – 5000*1/256 ~ 20 раз
Если от ожидания отличается меньше, чем в 2 раза – все нормально. То есть от 10 до 40 раз - ок
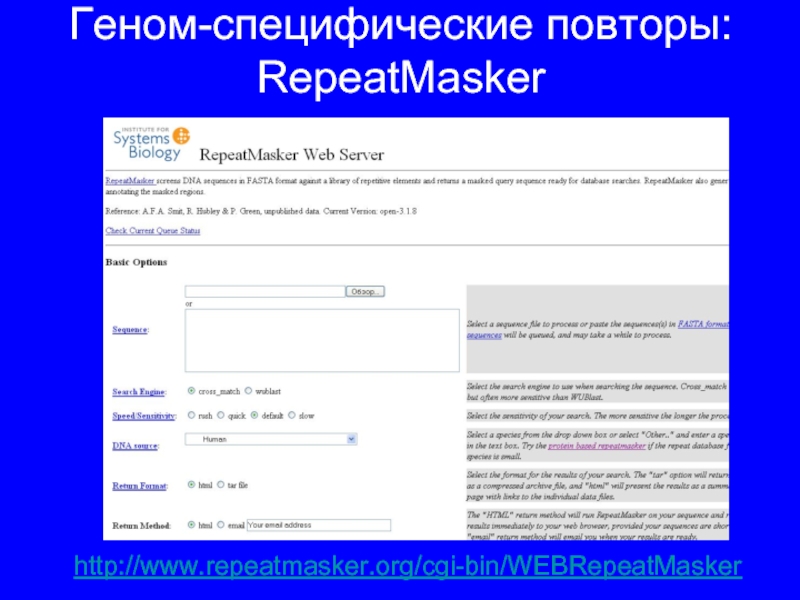
Слайд 21Геном-специфические повторы: RepeatMasker
http://www.repeatmasker.org/cgi-bin/WEBRepeatMasker
Слайд 22Поиск (белок-кодирующих) генов
Прокариоты – просто поиск длинных открытых рамок считывания (ORF)
(> 100 aa)
ORFing – например, ORF finder на сайте NCBI
ORFing – например, ORF finder на сайте NCBI
http://www.ncbi.nlm.nih.gov/
gorf/gorf.html
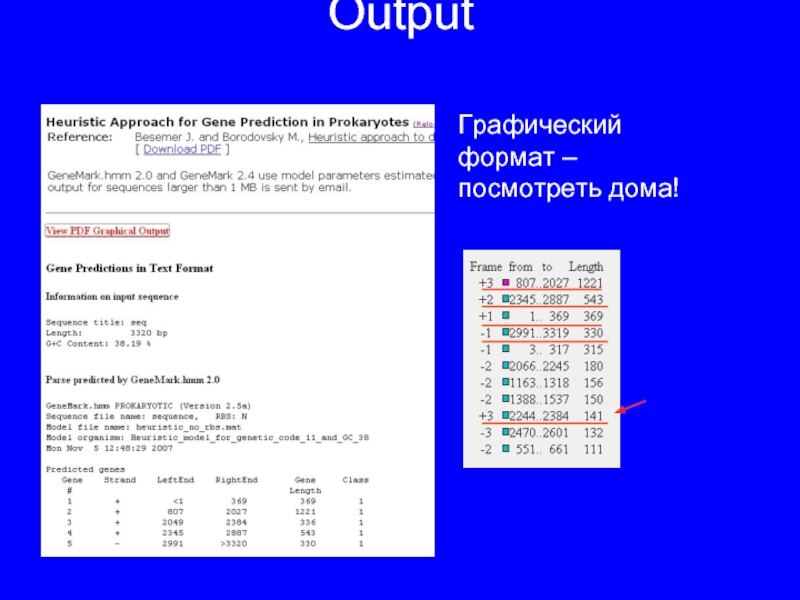
Слайд 23Output
Открытые рамки сортированы по длине
Графическое представление – ссылка на белковую последовательность,
соответствующую ORF
Можно сразу запустить бласт этой последовательности по разным подмножествам GenBank
Если надо найти CDS в эукариотической мРНК – абсолютно аналогично
Можно сразу запустить бласт этой последовательности по разным подмножествам GenBank
Если надо найти CDS в эукариотической мРНК – абсолютно аналогично
Слайд 24Более точное предсказание –GeneMark (HMM)
http://opal.biology.gatech.edu/
GeneMark/
Использует Hidden Markov Models
Более короткие
рамки
Выбор из нескольких перекрывающихся рамок
Более точное предсказание старта
Выбор из нескольких перекрывающихся рамок
Более точное предсказание старта
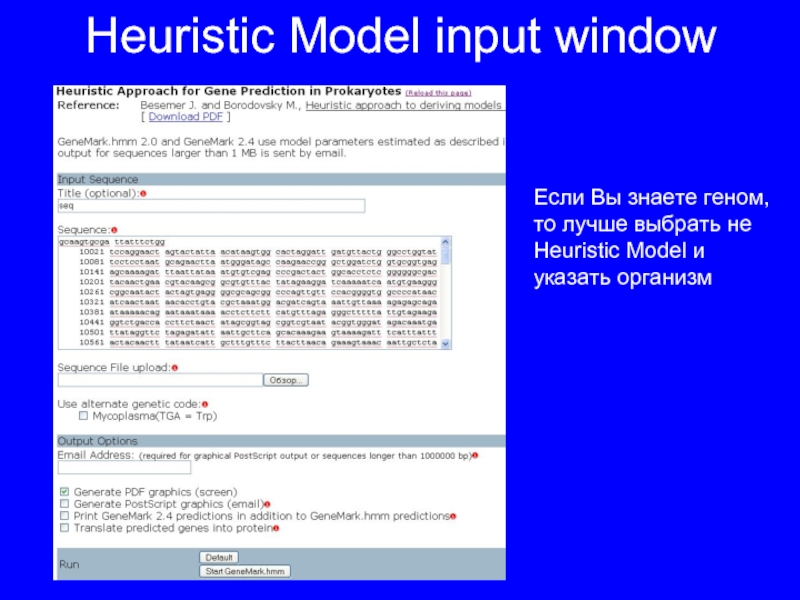
Слайд 25Heuristic Model input window
Если Вы знаете геном, то лучше выбрать не
Heuristic Model и указать организм
Слайд 27Предсказание внутренних экзонов (позвоночные)
Принцип:
ищут те участки, которые статистически похожи на
белок-кодирующие сегменты (codon usage, статистика ДНК)
Выбирают только те из них, которые фланкированы подходящими последовательностями (splicing sites)
То есть (!), ищут только внутренние, белок-кодирующие экзоны
Выбирают только те из них, которые фланкированы подходящими последовательностями (splicing sites)
То есть (!), ищут только внутренние, белок-кодирующие экзоны
Слайд 29MZEF - output
Результат работы программы на сегменте генома человека ~2 Kbp,
включающем 2 полных экзона и экзон на границе сегмента
Типичный выход – ~1/2
Типичный выход – ~1/2
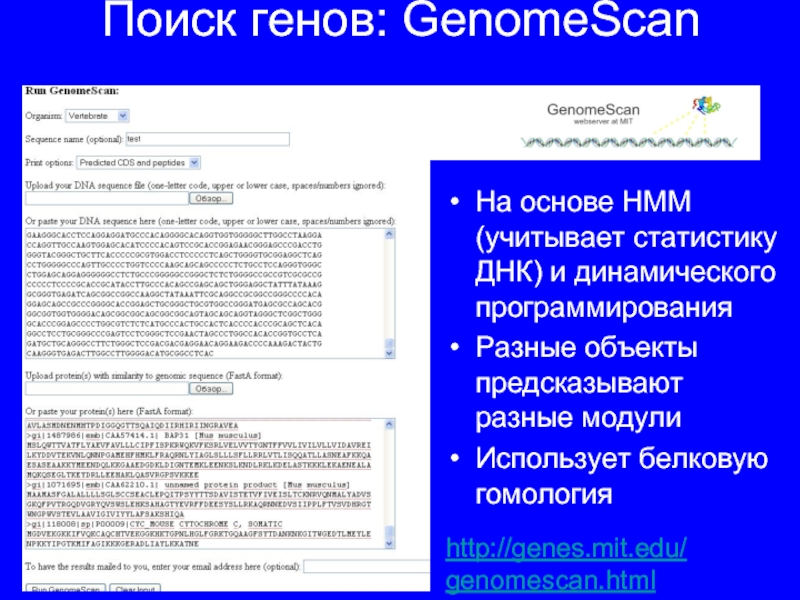
Слайд 30Поиск генов: GenomeScan
На основе HMM (учитывает статистику ДНК) и динамического программирования
Разные
объекты предсказывают разные модули
Использует белковую гомология
Использует белковую гомология
http://genes.mit.edu/
genomescan.html
Слайд 32Сборка геномных фрагментов в контиги: EGassembler
http://egassembler.hgc.jp/
Чистит последовательности
Маскирует повторы
Маскирует векторные сегменты
Маскирует сегменты
геномов органелл
Собирает контиги
Собирает контиги
Слайд 34Поиски регуляторных сигналов
Пока поиск слишком несовершенен
Самые лучшие программы не доступны on-line
Результаты
программ должен курировать специалист
Почти все подходы используют Positional Weight Matrix (PWM)
Почти все подходы используют Positional Weight Matrix (PWM)























![Positional weight matrix (PWM)I = Σj Σb f(b,j)[log f(b,j) / p(b)] Information content](/img/tmb/2/140145/5f59eef0550d05f0040cf9616baa12a8-800x.jpg)





