- Главная
- Разное
- Дизайн
- Бизнес и предпринимательство
- Аналитика
- Образование
- Развлечения
- Красота и здоровье
- Финансы
- Государство
- Путешествия
- Спорт
- Недвижимость
- Армия
- Графика
- Культурология
- Еда и кулинария
- Лингвистика
- Английский язык
- Астрономия
- Алгебра
- Биология
- География
- Детские презентации
- Информатика
- История
- Литература
- Маркетинг
- Математика
- Медицина
- Менеджмент
- Музыка
- МХК
- Немецкий язык
- ОБЖ
- Обществознание
- Окружающий мир
- Педагогика
- Русский язык
- Технология
- Физика
- Философия
- Химия
- Шаблоны, картинки для презентаций
- Экология
- Экономика
- Юриспруденция
Методы изучения регуляторных районов генов презентация
Содержание
- 1. Методы изучения регуляторных районов генов
- 3. Fig. 1. Focused versus dispersed transcription initiation.
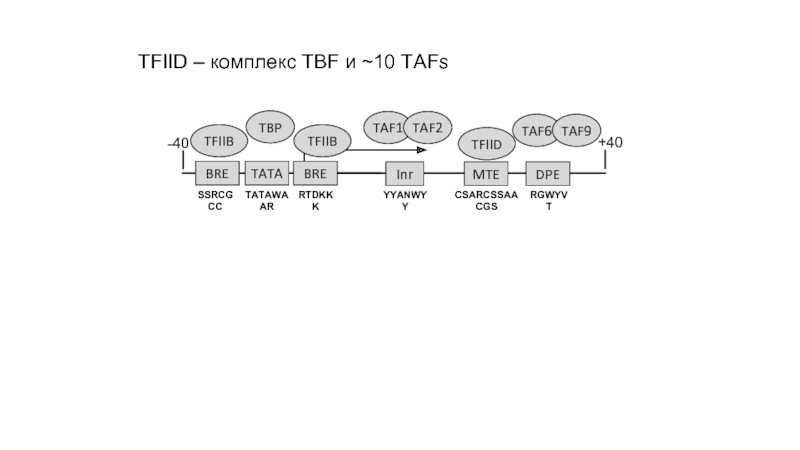
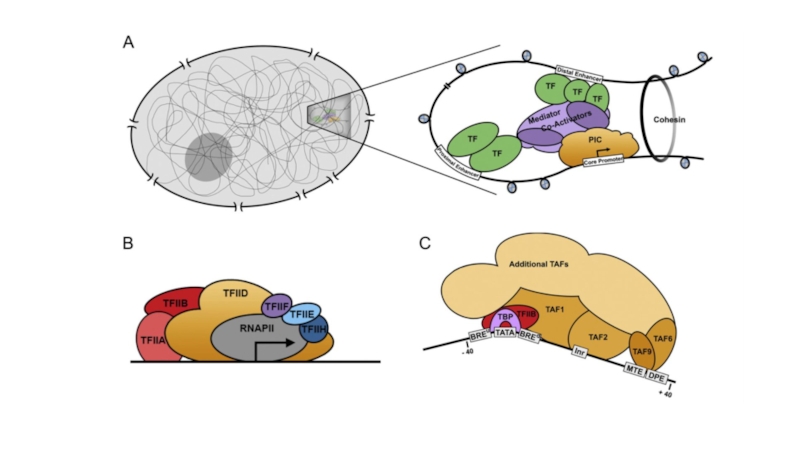
- 4. TFIID – комплекс TBF и ~10 TAFs
- 7. Структурно-функциональная организация промоторного района гена Xist полевки
- 9. Orphan CGIs are sites of transcriptional initiation.
- 10. Untranslated exons 1A, 1B, or 1C are
- 11. Genomic organization of mouse plectin (A), human
- 12. Genome-wide distribution of human and mouse genes
- 13. 5’
- 14. RACE Procedure Transcriptional Regulation in Eukaryotes: Concepts,
- 15. RNase Protection Transcriptional Regulation in Eukaryotes: Concepts,
- 16. Интересующий промотор Репортерный ген (Люцефераза или САТ)
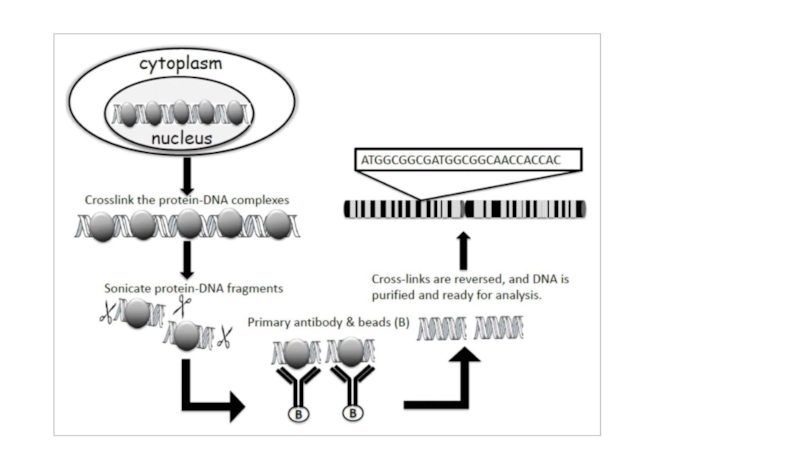
- 18. Robert L. Strausberg, Elise A. Feingold, Lynette H. Grouse, Jeffery
- 19. mRNA mRNA mRNA mRNA mRNA mRNA mRNA mRNA mRNA mRNA mRNA mRNA
- 21. The eukaryotic promoter database in its 30th
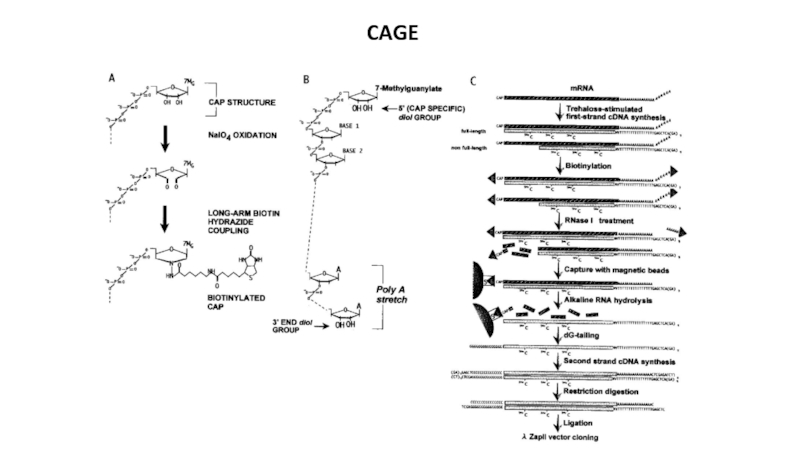
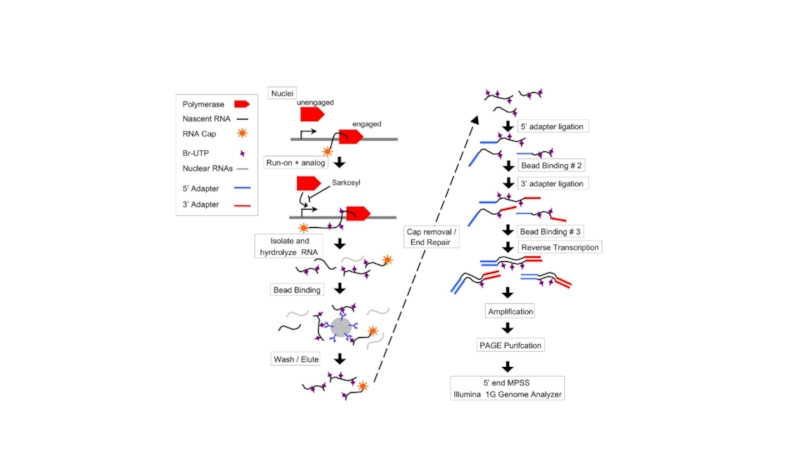
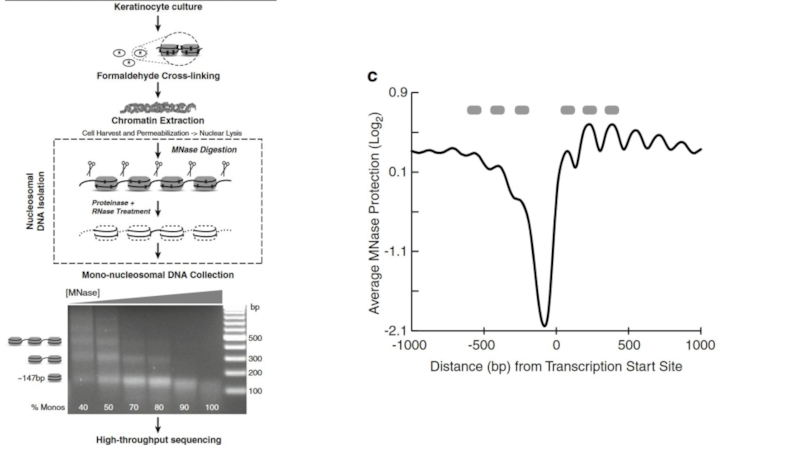
- 22. CAGE
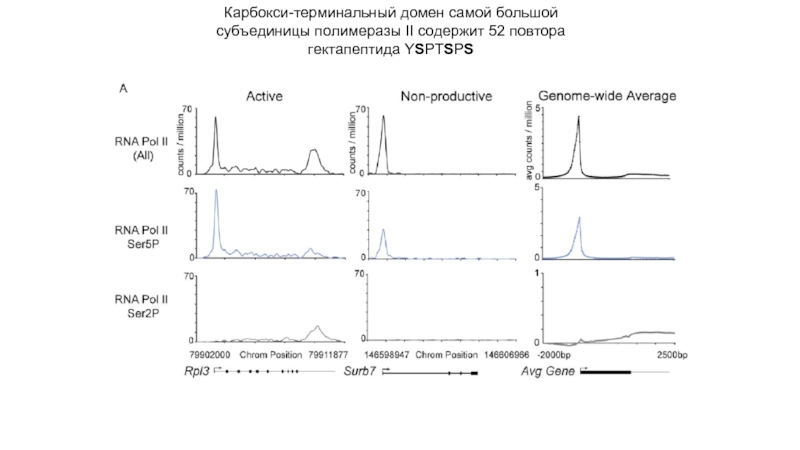
- 28. Карбокcи-терминальный домен самой большой субъединицы полимеразы II содержит 52 повтора гектапептида YSPTSPS
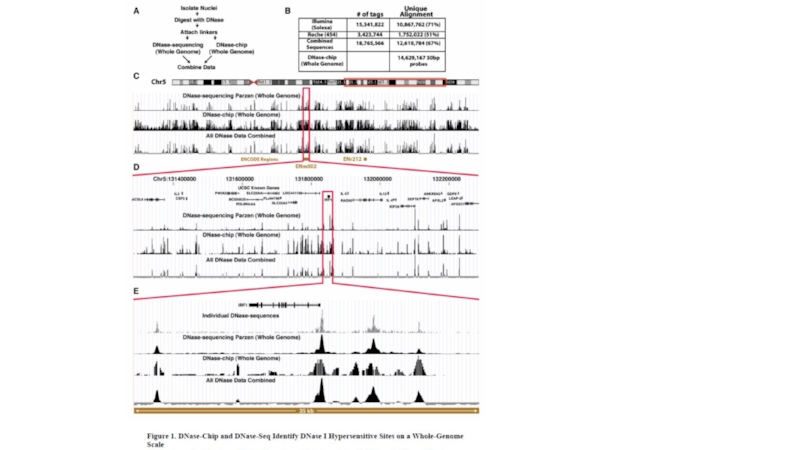
- 31. ENCODE Project Scientific Management: National Human Genome
- 32. The ENCODE Project Consortium. 2004. The ENCODE
- 33. Интересующий промотор Репортерный ген (Люцефераза или САТ)
- 34. Clustergram of 642 putative promoter fragments.
Слайд 1Меркулова Татьяна Ивановна
Институт цитологии и генетики СО РАН
Методы изучения регуляторных
районов генов
Лекция
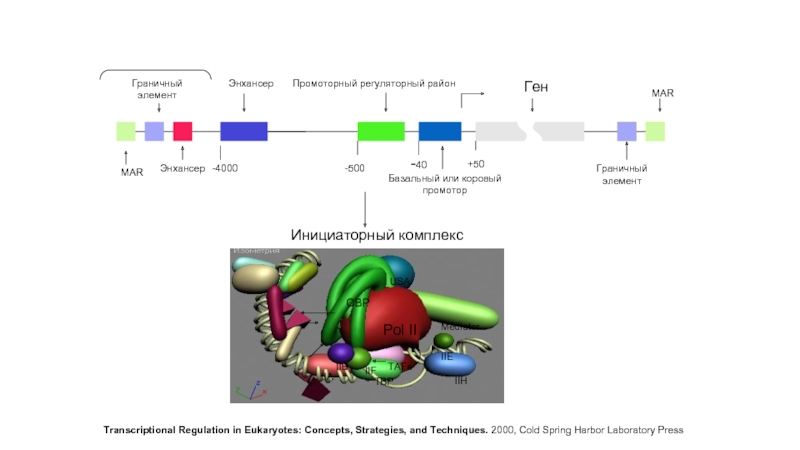
Слайд 2
MAR
Граничный
элемент
Энхансер
-4000
Энхансер
Промоторный регуляторный район
-500
Ген
-40
+50
Базальный или коровый
промотор
Граничный
элемент
MAR
Инициаторный комплекс
Pol II
Mediator
IIH
USA
IIA
TBP
TAFs
IIB
IIF
CBP
IIE
Transcriptional Regulation in Eukaryotes:
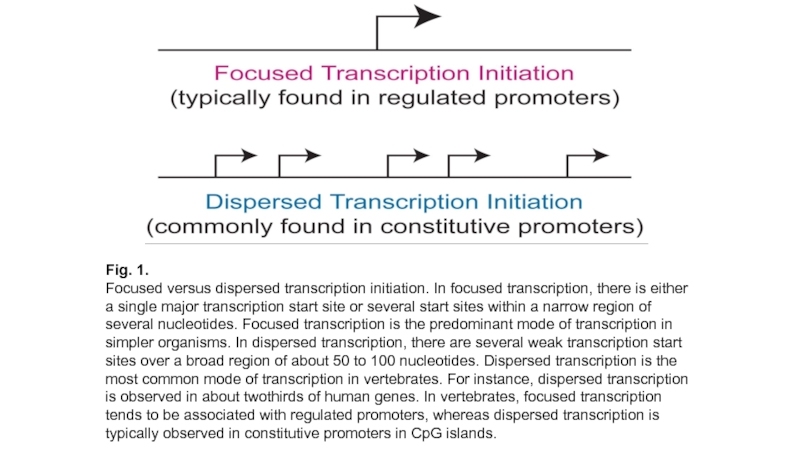
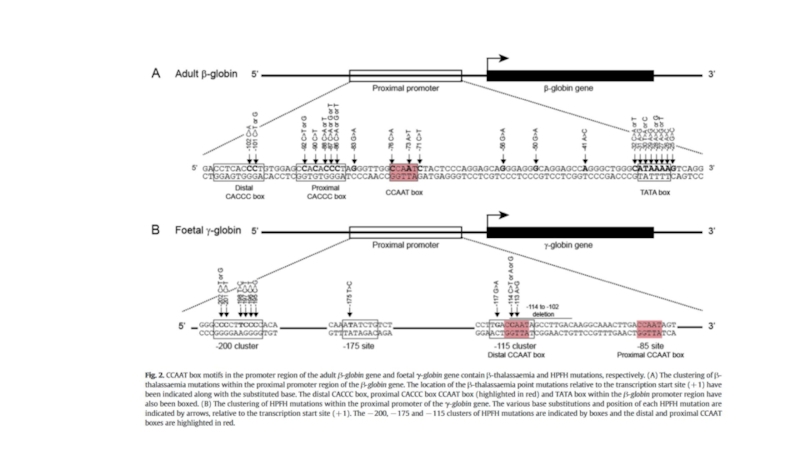
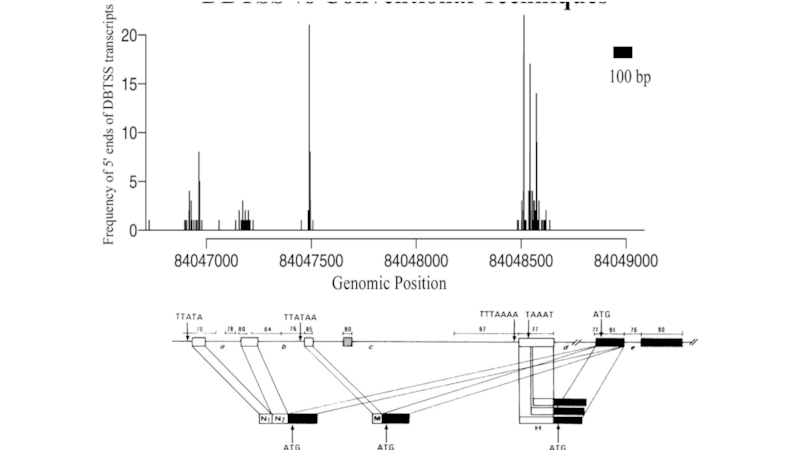
Слайд 3Fig. 1.
Focused versus dispersed transcription initiation. In focused transcription, there is
Слайд 7Структурно-функциональная организация промоторного района гена Xist полевки Microtus rossiaemeridionalis
рСх1-рСх14Ме –
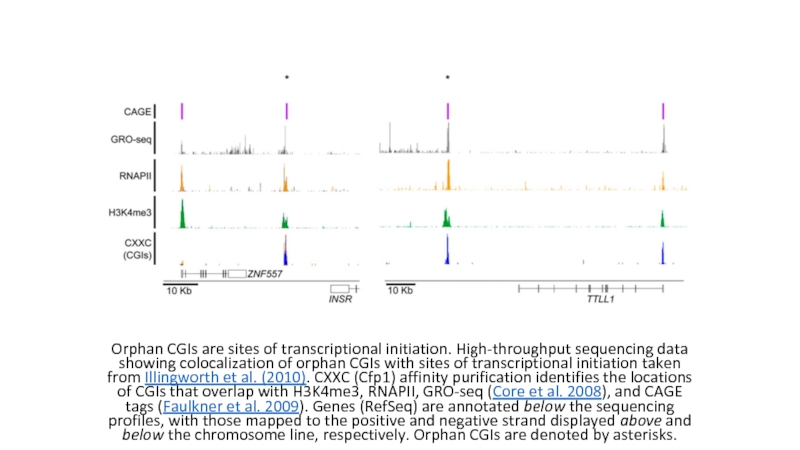
Слайд 9Orphan CGIs are sites of transcriptional initiation. High-throughput sequencing data showing
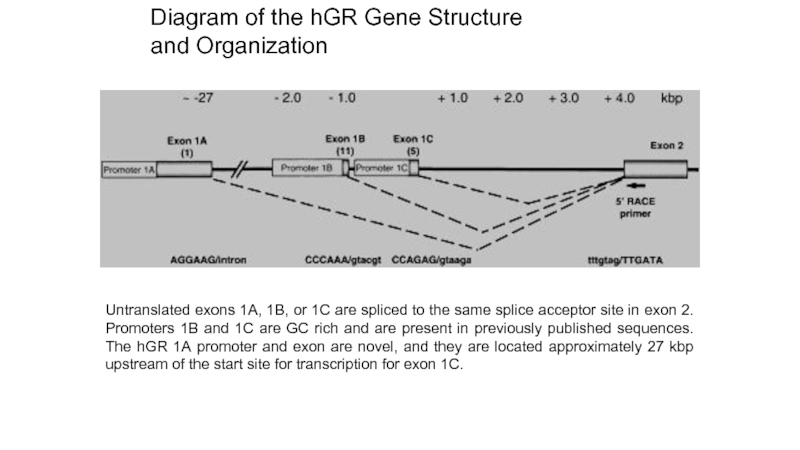
Слайд 10Untranslated exons 1A, 1B, or 1C are spliced to the same
Diagram of the hGR Gene Structure
and Organization
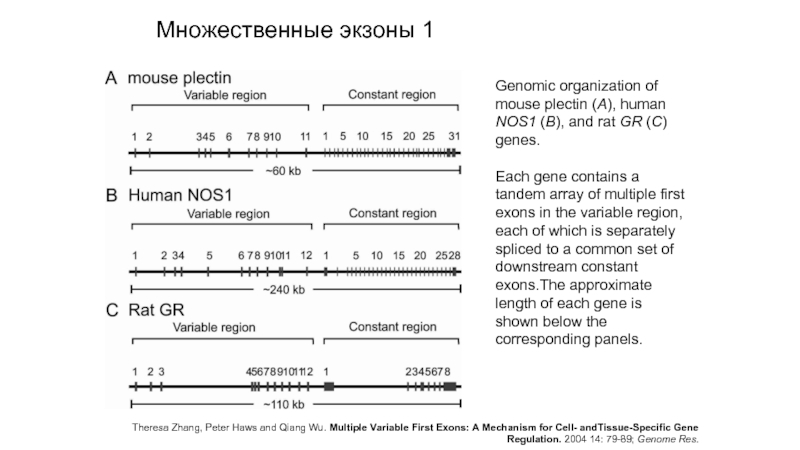
Слайд 11Genomic organization of mouse plectin (A), human NOS1 (B), and rat
Each gene contains a tandem array of multiple first exons in the variable region, each of which is separately spliced to a common set of downstream constant exons.The approximate length of each gene is shown below the corresponding panels.
Theresa Zhang, Peter Haws and Qiang Wu. Multiple Variable First Exons: A Mechanism for Cell- andTissue-Specific Gene Regulation. 2004 14: 79-89; Genome Res.
Множественные экзоны 1
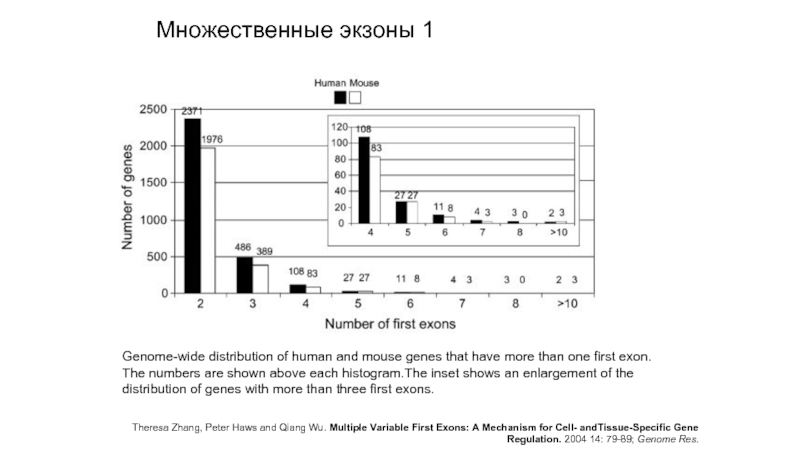
Слайд 12Genome-wide distribution of human and mouse genes that have more than
The numbers are shown above each histogram.The inset shows an enlargement of the distribution of genes with more than three first exons.
Theresa Zhang, Peter Haws and Qiang Wu. Multiple Variable First Exons: A Mechanism for Cell- andTissue-Specific Gene Regulation. 2004 14: 79-89; Genome Res.
Множественные экзоны 1
Слайд 13
5’ 3’
5’
3’
3’
5’
87nt
G
Extended
primer
87 nt
Free excess
primer
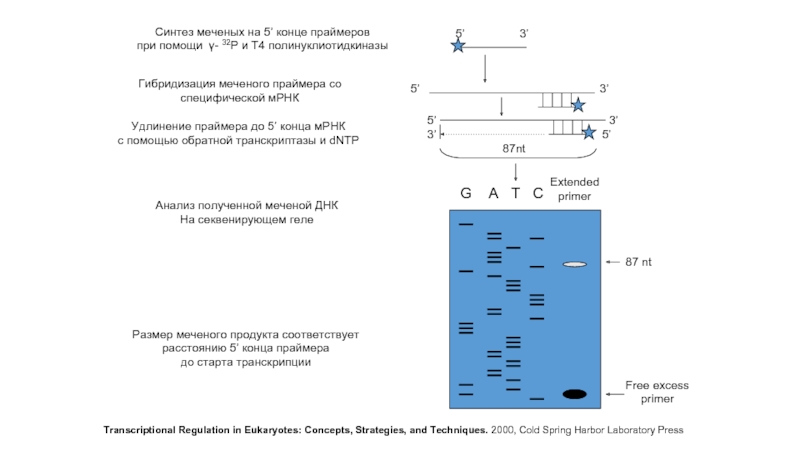
Синтез меченых на 5’ конце праймеров
при помощи γ- 32P и Т4 полинуклиотидкиназы
Гибридизация меченого праймера со
специфической мРНК
5’
3’
Удлинение праймера до 5’ конца мРНК
c помощью обратной транскриптазы и dNTP
Анализ полученной меченой ДНК
На секвенирующем геле
Размер меченого продукта соответствует
расстоянию 5’ конца праймера
до старта транскрипции
Transcriptional Regulation in Eukaryotes: Concepts, Strategies, and Techniques. 2000, Cold Spring Harbor Laboratory Press
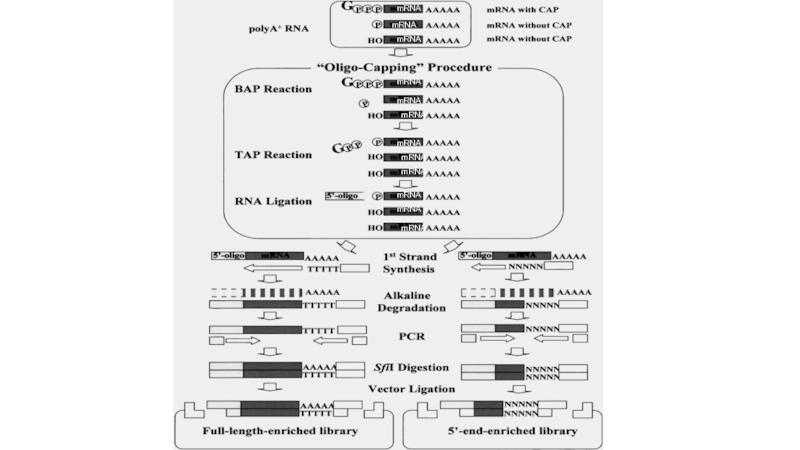
Слайд 14RACE Procedure
Transcriptional Regulation in Eukaryotes: Concepts, Strategies, and Techniques. 2000, Cold
Hybridize primer 100–200 nucleotides from 5′ end of mRNA.
Extend primer to 5′ end of mRNA using reverse transcriptase
and dNTPs.
Ligate oligo of known sequence to 3′ end of cDNA using RNA
ligase. Alternatively, extend cDNA using terminal transferase
(TdT) and dGTP.
Perform PCR using primer complementary to ligated oligo and a downstream primer that is slightly internal to the primer used for
cDNA synthesis above.
(Optional)
Sequence PCR products
Sequence
Individual clones
Determine size on
sequencing gel (if
radiolabeled primer
used for PCR).
Insert PCR
product into
vector
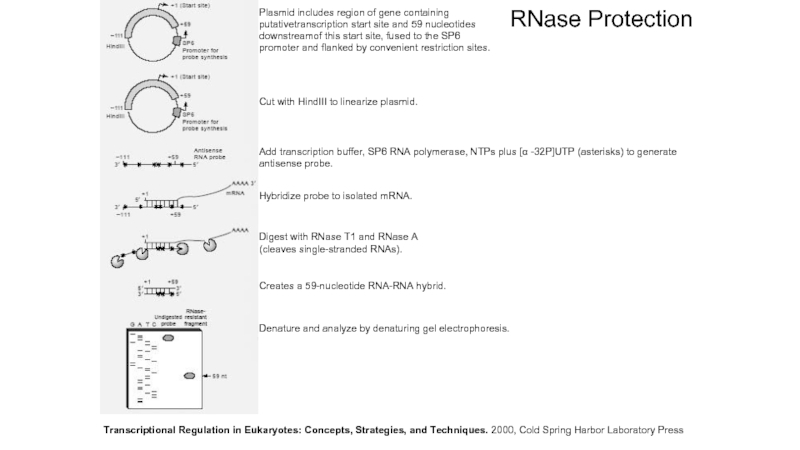
Слайд 15RNase Protection
Transcriptional Regulation in Eukaryotes: Concepts, Strategies, and Techniques. 2000, Cold
Plasmid includes region of gene containing
putativetranscription start site and 59 nucleotides
downstreamof this start site, fused to the SP6
promoter and flanked by convenient restriction sites.
Cut with HindIII to linearize plasmid.
Add transcription buffer, SP6 RNA polymerase, NTPs plus [α -32P]UTP (asterisks) to generate antisense probe.
Hybridize probe to isolated mRNA.
Digest with RNase T1 and RNase A
(cleaves single-stranded RNAs).
Creates a 59-nucleotide RNA-RNA hybrid.
Denature and analyze by denaturing gel electrophoresis.
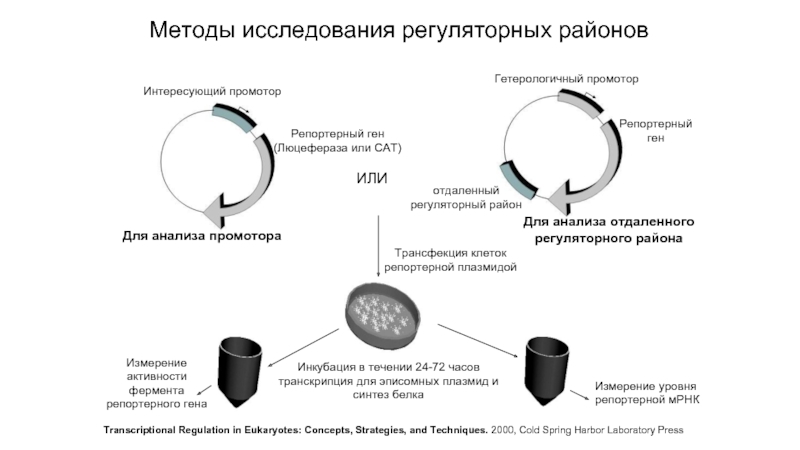
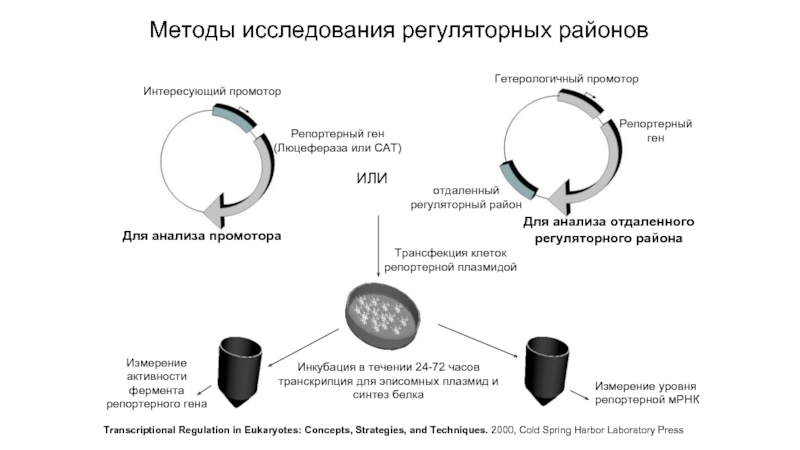
Слайд 16Интересующий промотор
Репортерный ген
(Люцефераза или САТ)
Гетерологичный промотор
Репортерный
ген
Для анализа промотора
ИЛИ
Для анализа
регуляторного района
отдаленный
регуляторный район
Трансфекция клеток
репортерной плазмидой
Инкубация в течении 24-72 часов
транскрипция для эписомных плазмид и
синтез белка
Измерение
активности
фермента
репортерного гена
Измерение уровня
репортерной мРНК
Transcriptional Regulation in Eukaryotes: Concepts, Strategies, and Techniques. 2000, Cold Spring Harbor Laboratory Press
Методы исследования регуляторных районов
Слайд 18Robert L. Strausberg, Elise A. Feingold, Lynette H. Grouse, Jeffery G. Derge, Richard D. Klausner, Francis
Contributed by Francis S. Collins, et al.
Generation and initial analysis of more than 15,000 full-length human and mouse cDNA sequences
Mammalian Gene Collection (MGC) Program Team*, 2000.
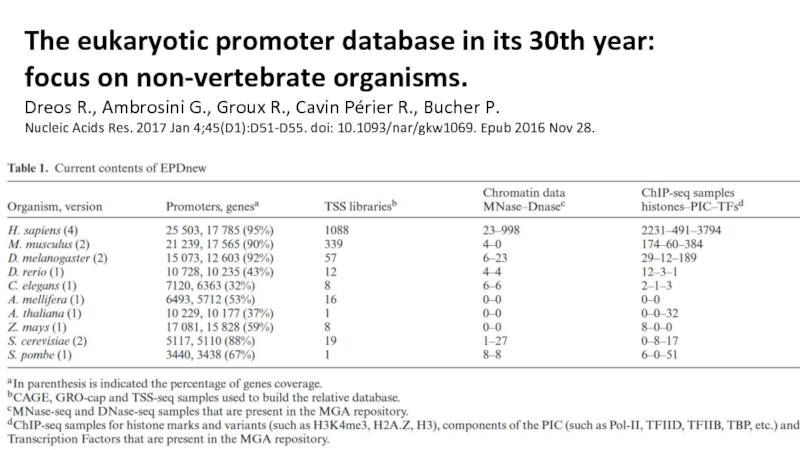
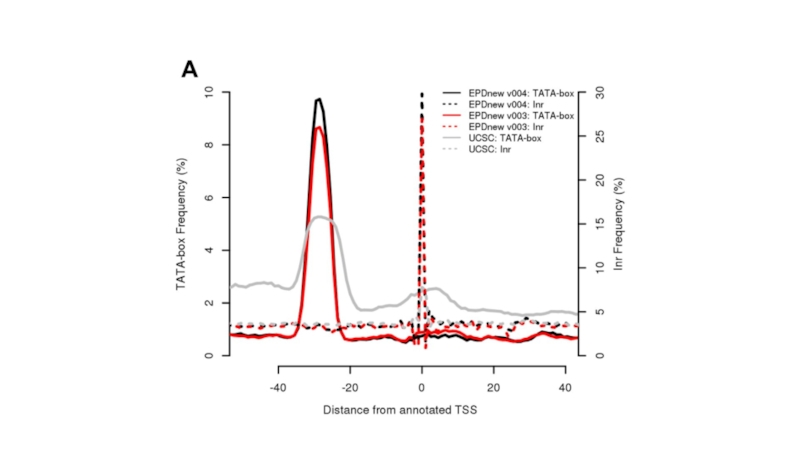
Слайд 21The eukaryotic promoter database in its 30th year:
focus on non-vertebrate
Dreos R., Ambrosini G., Groux R., Cavin Périer R., Bucher P.
Nucleic Acids Res. 2017 Jan 4;45(D1):D51-D55. doi: 10.1093/nar/gkw1069. Epub 2016 Nov 28.
Слайд 28Карбокcи-терминальный домен самой большой субъединицы полимеразы II содержит 52 повтора гектапептида
Слайд 31ENCODE Project Scientific Management:
National Human Genome Research Institute (E. A.
Feingold, P.
K. Wetterstrand, F. S. Collins).
Initial ENCODE Pilot Phase Participants:
Affymetrix, Inc. (T. R. Gingeras, D. Kampa, E. A. Sekinger,
J. Cheng, H. Hirsch, S. Ghosh, Z. Zhu, S. Patel, A. Piccolboni, A. Yang, H. Tammana, S. Bekiranov, P. Kapranov, R. Harrison, G. Church, K. Struhl); Ludwig Institute for Cancer Research (B. Ren, T. H. Kim, L. O. Barrera, C. Qu, S. Van Calcar, R. Luna, C. K. Glass, M. G. Rosenfeld); Municipal Institute of Medical Research (R. Guigo, S. E. Antonarakis, E. Birney, M. Brent, L. Pachter, A. Reymond, E. T. Dermitzakis, C. Dewey, D. Keefe, F. Denoeud, J. Lagarde, J. Ashurst, T. Hubbard, J. J. Wesselink, R. Castelo, E. Eyras); Stanford University (R. M. Myers, A. Sidow, S. Batzoglou, N. D. Trinklein, S. J. Hartman, S. F. Aldred, E. Anton, D. I. Schroeder, S. S. Marticke, L. Nguyen, J.Schmutz, J.Grimwood,M.Dickson, G. M. Cooper, E. A. Stone, G. Asimenos, M. Brudno); University of Virginia (A. Dutta, N. Karnani, C. M. Taylor, H. K. Kim, G. Robins); University of Washington (G. Stamatoyannopoulos, J. A. Stamatoyannopoulos, M. Dorschner, P. Sabo, M. Hawrylycz, R. Humbert, J. Wallace, M. Yu, P. A. Navas, M. McArthur, W. S. Noble); Wellcome Trust Sanger Institute (I. Dunham, C. M. Koch, R.M.Andrews,G. K.Clelland, S. Wilcox, J. C. Fowler, K.D.
James, P. Groth, O. M. Dovey, P. D. Ellis, V. L. Wraight, A. J. Mungall, P. Dhami, H. Fiegler, C. F. Langford, N. P. Carter,
D. Vetrie); Yale University (M. Snyder, G. Euskirchen, A. E.
Urban, U. Nagalakshmi, J. Rinn, G. Popescu, P. Bertone, S.
Hartman, J. Rozowsky, O. Emanuelsson, T. Royce, S. Chung, M. Gerstein, Z. Lian, J. Lian, Y. Nakayama, S. Weissman, V. Stolc, W. Tongprasit, H. Sethi).
Additional ENCODE Pilot Phase Participants:
British Columbia Cancer Agency Genome Sciences Centre (S. Jones, M. Marra, H. Shin, J. Schein); Broad Institute (M. Clamp, K. Lindblad-Toh, J. Chang, D. B. Jaffe, M. Kamal, E. S. Lander, T. S. Mikkelsen, J. Vinson, M. C. Zody); Children’s Hospital Oakland Research Institute
(P. J. de Jong, K. Osoegawa,M.Nefedov, B. Zhu); National Human Genome Research Institute/Computational Genomics Unit (A. D. Baxevanis, T. G. Wolfsberg); National Human Genome Research Institute/Molecular Genetics Section (F. S. Collins, G. E. Crawford, J. Whittle, I. E. Holt, T. J. Vasicek, D. Zhou, S. Luo); NIH Intramural
Sequencing Center/National Human Genome Research Institute (E. D. Green, G. G. Bouffard, E. H. Margulies, M. E. Portnoy, N. F. Hansen, P. J. Thomas, J. C. McDowell, B. Maskeri, A. C. Young, J. R. Idol, R. W. Blakesley); National Library of Medicine (G. Schuler); Pennsylvania State University (W. Miller, R. Hardison, L. Elnitski, P. Shah); The Institute for Genomic Research (S. L. Salzberg, M. Pertea, W. H. Majoros); University of California, Santa Cruz (D. Haussler, D. Thomas, K. R. Rosenbloom, H. Clawson, A. Siepel, W. J. Kent).
ENCODE Technology Development Phase Participants:
Boston University (Z. Weng, S. Jin, A. Halees, H. Burden, U. Karaoz, Y. Fu, Y. Yu, C. Ding, C. R. Cantor); Massachusetts General Hospital (R. E. Kingston, J. Dennis); NimbleGen Systems, Inc. (R. D. Green,M. A. Singer, T. A. Richmond, J. E. Norton, P. J Farnham, M. J. Oberley, D. R. Inman); NimbleGen Systems, Inc. (M. R. McCormick, H. Kim, C. L. Middle, M. C. Pirrung); University of California, et al!
The ENCODE Project Consortium. 2004. The ENCODE (ENCyclopedia Of DNA Elements) Project. Science 306: 636–640.
The ENCODE Project Consortium
Слайд 32The ENCODE Project Consortium. 2004. The ENCODE (ENCyclopedia Of DNA Elements)
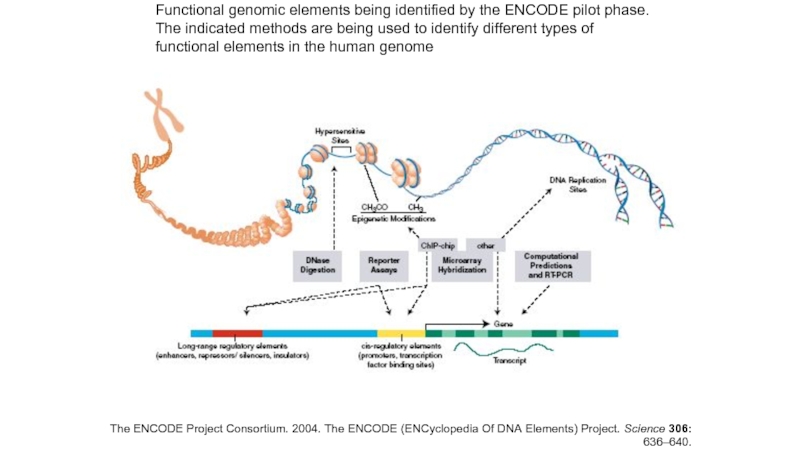
Functional genomic elements being identified by the ENCODE pilot phase.
The indicated methods are being used to identify different types of
functional elements in the human genome
Слайд 33Интересующий промотор
Репортерный ген
(Люцефераза или САТ)
Гетерологичный промотор
Репортерный
ген
Для анализа промотора
ИЛИ
Для анализа
регуляторного района
отдаленный
регуляторный район
Трансфекция клеток
репортерной плазмидой
Инкубация в течении 24-72 часов
транскрипция для эписомных плазмид и
синтез белка
Измерение
активности
фермента
репортерного гена
Измерение уровня
репортерной мРНК
Transcriptional Regulation in Eukaryotes: Concepts, Strategies, and Techniques. 2000, Cold Spring Harbor Laboratory Press
Методы исследования регуляторных районов
Слайд 34Clustergram of 642 putative promoter fragments.
The clustergram illustrates the hierarchical
Each row indicates the promoter activity of a fragment in each of the cell lines, with red indicating the degree of activity and black indicating no activity.
Promoter activity has been normalized and log transformed to reflect comparable values between cell lines.
Area A represents a cluster of promoter fragments with strong, ubiquitous activity in all cell lines and area B represents a cluster of promoter fragments that exhibit variable function across the 16 cell types.
Comprehensive analysis of transcriptional promoter structure and function in 1% of the human genome
Sara J. Cooper, Nathan D. Trinklein, Elizabeth D. Anton, Loan Nguyen and Richard M. Myers Genome Res., 2006 16: 1-10;
Comprehensive analysis of transcriptional promoter structure and function in 1% of the human genome/ Sara J. Cooper, Nathan D. Trinklein, et al. Genome Res., 2006 16: 1-10